条件重编程(conditional reprogramming,CR)培养技术目前应用于多种原代细胞系的培养,由人诱导多能干细胞培养体系改良,并经过试验专用于培养动物组织来源的上皮细胞。CR培养的具体步骤是细胞与辐射过的小鼠成纤维J2细胞(滋养细胞)在配有Rho相关激酶(Rho-associated kinase,ROCK)抑制剂(Y-27632)的条件培养基中共培养。ROCK抑制剂可提高骨髓源性间充质干细胞和人角质形成细胞的增殖能力,诱导细胞无限增殖,故加入Y-27632的培养基可以用来增加患者来源的正常或肿瘤上皮细胞的细胞株,以及肝细胞系的存活能力。该模型保持了原代细胞的生物学特性,含有药物代谢酶及相应的辅助因子,可为药敏试验提供相关信息,为研究药物代谢和药物相互作用提供了良好的体外模型,应用于肿瘤细胞培养有较好前景。CR细胞培养技术作为原代细胞培养技术的新兴技术,目前仅用于膀胱癌、前列腺癌、非小细胞肺癌(non-small cell lung cancer,NSCLC)、小鼠神经母细胞瘤等相关领域。随着技术的逐步成熟,CR培养技术将广泛应用于各个系统肿瘤。本文对CR培养技术应用于不同肿瘤细胞及其在药物代谢中的应用进展进行了综述。
CR培养体系概述 多年来科学家致力于寻找一种能对人类正常细胞和肿瘤细胞进行永久体外培养的培养体系[1],但是传统的细胞体外培养方法会改变细胞的表型、染色体组型、蛋白表达量。更为重要的是肿瘤细胞在体外培养会丧失原代肿瘤细胞的异质性,同时没有基因突变的正常细胞由于端粒酶等限制无法在体外培养中长时间存活,阻碍了对不同疾病进行分子和细胞层面的深入研究。
一般采用SV病毒(simian vacuolating virus)大T抗原与管家蛋白相互作用[2]诱导细胞体外永生化或者过表达人类端粒酶逆转录酶(human telomerase reverse transcriptase,hTERT)[3],使细胞获得持续增殖的生物学特性。然而,通过基因操作的方式对细胞进行培养可能会造成染色体不稳定,比如在传代之后造成不可逆的关键基因或者性状的丢失。大多数原代细胞难以维持体外扩增,一般经过有限代数就会停止生长并逐渐死亡。
CR培养技术由人诱导多能干细胞培养体系改良并经过试验,专用于培养动物组织来源的上皮细胞。经过CR培养体系建立的原代细胞可以维持来源组织的染色体特征,并保持正常组织来源的生物学特性。例如人类前列腺癌的CR细胞维持了来源组织第13号染色体的突变,故原代细胞具有在基质胶中生长和在免疫缺陷小鼠皮下成瘤等生物学特征[4]。CR技术用于肺部上皮、乳腺上皮[5]、结肠上皮、食管上皮[6]、包皮上皮和子宫颈阴道部及尿液脱落膀胱癌细胞,均获得成功[7-8]。同时,重编程培养细胞(CR cell,CRC)在成人上皮细胞中表现出干细胞特性[9]。不同于其他细胞模型,CR技术可以早期建立患者来源的正常或肿瘤细胞培养株,并保持无限扩增而无需基因处理,从而保留了原代细胞的性状和染色体特性。Y-27632对细胞的永生化诱导和人乳头瘤病毒(human papilloma virus,HPV-16)E6和E7致癌基因诱导永生化的作用相同,E6和滋养细胞的作用都是激活端粒酶,然而E7和Y-27632都能破坏肌动蛋白细胞骨架并使Rho13失活[10]。CR培养是可逆的,将培养细胞移出CR培养基,根据培养条件不同,细胞停止增殖或终末分化。CRC区别于胚胎干细胞(embryonic stem cell,EC)和诱导多能干细胞(induced pluripotent stem cell,iPC),CRC不是挑选的结果,也不是通过基因操作诱导的结果。CR技术能快速建立所需细胞模型,维持原代细胞的表型、肿瘤异质性(主要指肿瘤内异质性)[11]、染色体组型和药物敏感性等特征。
近年来,患者来源的异种移植物(patient-derived tumor xenograft,PDX)逐渐发展成为更加系统的研究肿瘤发生、发展过程的模型。泛组织来源的CRC可用于类器官PDX模型的建立,因此CR技术具有基础研究、临床诊断、治疗和再生医学的应用前景(图 1)。
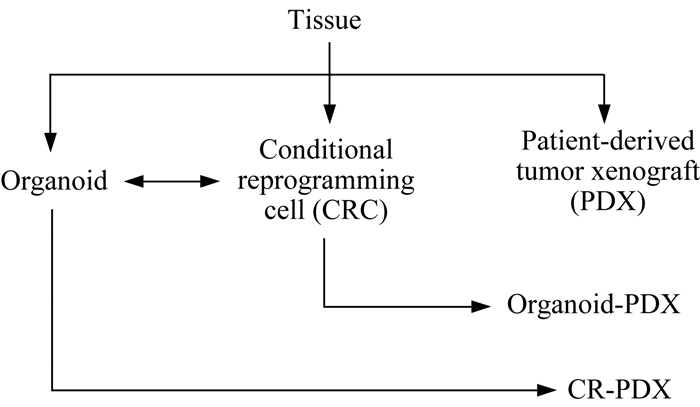
|
| 图 1 泛组织来源培养体系的工作流程 Fig 1 Workflow of extensive tissue-derived culture |
CR应用
CR建立稳定的患者来源的PDX细胞株 目前抗肿瘤活性药物的筛查依旧使用标准的癌症细胞系。由于肿瘤细胞系易于扩增的特性,利用这些细胞系已经对癌症的恶变机制以及药物反应有了一定的了解。进一步研究发现,在药物毒性作用下肿瘤细胞系选择压力导致细胞系发生分化,故难以代表原来肿瘤的特性。因此需要建立更多的代表肿瘤生物学特性的细胞株,用于药物临床前期的抗癌药效筛选及验证。
PDX是患者肿瘤和基质组织直接移植入严重免疫缺陷小鼠,并在体内维持,可用于临床前药物试验和转化医学试验[12]。PDX模型相比于标准癌症细胞株,优点在于维持了肿瘤的生物学特性,缺点是动物消耗大,培养时间长,移植率不一致,缺乏持续的体外生长,不适合大批量使用。传统方法培养的PDX细胞多数停止增殖或在几代内发生凋亡。
Borodovsky等[13]将CR技术与PDX技术结合,在体外以较高成功率传代培养患者来源的PDX组织。标准肿瘤细胞系不能明确代表原代肿瘤的遗传学和异质性,所以在临床药物敏感试验以及转化医学中作用有限。作者通过300个基因对晚期传代的CR-PDX细胞系进行靶向序列分析,数据显示CR-PDX成功维持了同位基因的频率,没有发生基因漂移,证明CR-PDX维持了遗传学的稳定性。另一方面,结果证明CR-PDX与原发肿瘤的药物敏感性相同,可以用作药物筛查试验。标准肿瘤细胞系的优点在于能够通过靶向敲除基因改变基因表达来判断基因型和表型的关系。用靶向或打乱的对照小干扰RNA(small interfering RNA,siRNA)转染CR-PDX细胞,通过实时荧光定量PCR(qRT-PCR)证明敲除成功,在研究结束时评估基因敲除对存活率的影响,研究数据显示CR-PDX细胞可以用来进行基因操作研究,以探讨细胞生理学,并为临床相关细胞群的机制研究提供证据[13]。
CR培养的尿液来源的膀胱癌细胞在精准治疗膀胱癌中的作用在中国,膀胱癌是泌尿生殖系统肿瘤中最常见的肿瘤[14]。膀胱癌的症状不明显,一经发现大部分是中晚期,膀胱癌的治疗方案不仅和临床分期有关,而且与患者个体临床情况有关。根据膀胱癌的不同分型,无肌层浸润膀胱癌的5年生存率为60%~70%[15],肌层浸润性膀胱癌的5年生存率仅为35%。因此,需要一种新型无创模型来早期诊断膀胱癌,并根据患者个体临床情况选择适当的放化疗及手术,以提高患者生存率和改善患者生存质量。
膀胱癌患者进行活检来获取肿瘤组织构建PDX的药敏模型和确认膀胱癌患者的临床分期属于有创操作。尿液病理能提供精确、动态的临床信息,可以捕获复杂的基因突变情况以及原发或转移肿瘤的特点,同时尿液是易于获取且无创检查,已广泛应用于临床,通过细胞学和生物标记物来诊断膀胱癌。因此,通过CR技术建立的尿液来源的肿瘤细胞培养是临床上理想的方法(图 2)。
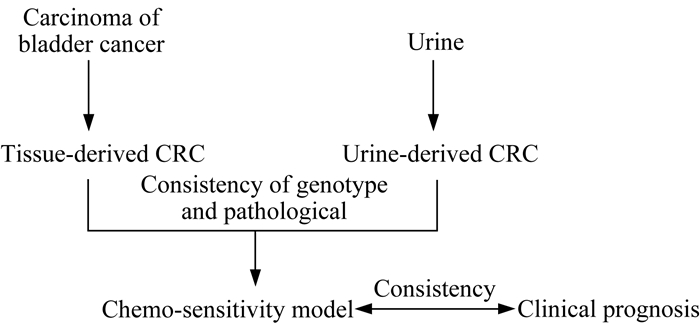
|
| 图 2 CR原代培养在尿液中的应用 Fig 2 Application of CR culture in urine |
我们实验室通过CR技术建立源于原发肿瘤样本和尿液的肿瘤细胞集落,并进行3D培养形成球体,HE染色结果显示CRC表现出的肿瘤细胞特性与相应的肿瘤病理切片相同;使用IHC染色尿液中CRC观察细胞类型特异性标记物(GATA3、P40、P63)的表达,发现与相应原发性肿瘤细胞相一致;对尿液CRC进行全外显子测序(whole exome sequencing,WES)分析,与相应的亲代肿瘤进行比较,发现CR细胞与原发肿瘤组织共有79.7%~82.6%的变异谱。对原发肿瘤和尿液CRC变异率分析表明,单核苷酸变异和插入、缺失在培养过程中保持一致[16]。因此,CR技术维持了原发肿瘤细胞的分子特性以及遗传结构。这种快速有效建立尿液或患者来源的膀胱癌CRC的方法有利于进行大范围的药物筛查试验,以及了解细胞发生耐药性的机制,这种新颖、无创、快速建立体外膀胱肿瘤系统的方法为特异患者的药物反应和个体化药物治疗提供了基础[17]。
CR用于体外长期培养原代肝细胞 尽管肝细胞在体内拥有相当强的增殖能力,但在体外保持增殖和分化能力相当困难,理想的培养方法应满足细胞数量大、密度高、细胞功能强而持久、易获取等临床应用需求。前期研究主要在于培养永生化肝细胞,但永生化肝细胞势必会丢失一部分原代肝细胞性状,且在体外不能维持正常增殖和功能,所以需要一种新型技术培养非永生化肝细胞以维持原代肝细胞基因型、表型和功能。通过评价CYP3A4、1A1和2C9的活性以及白蛋白、s100a4、krt8、krt18、cyp1a1、cyp3a4、cyp2b6、cyp2c8、cyp2c9和cyp2d6的mRNA表达情况来评估肝细胞功能;同时使用短串联重复序列(short tandem repeats,STR)分析比较这些细胞的DNA指纹图谱:它们与原代肝组织拥有同一组织来源[18]。来源于年轻患者的原代肝细胞培养的CRC能在体外培养长达3个月,即使来源于年老患者的原代肝细胞培养的CRC也能培养长达2~3个月,远远超过市面上的肝细胞株,最为重要的是这些CRC在培养早期保持较高水平的CYP3A4、1A1和2C9,意味着CRC在早期具有较强的肝细胞功能。CR培养的成本低,耗材少,可为临床肝病研究以及解决肝源不足提供可行的体外培养模型(图 3)。
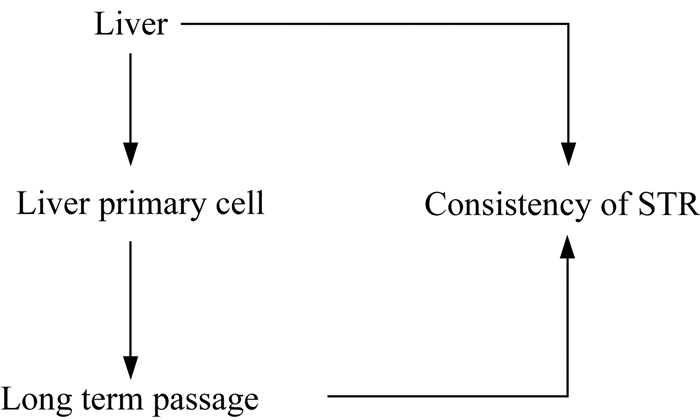
|
| 图 3 肝脏细胞原代培养 Fig 3 Primary culture of liver cells |
CR提供去势抵抗性前列腺癌药物筛查模型(图 4) 据统计,至2019年曾经被诊断患有前列腺癌美国男性将超360万人,确诊患者的中位年龄为66岁[19]。早期前列腺癌可以通过外科手段干预治疗,进展期前列腺癌缺乏有效的治疗手段,常规的去势药物或者手术治疗均会导致前列腺癌发展成为去势抵抗性前列腺癌(castration-resistant prostate cancer,CRPC)[20]。个体化癌症治疗基于患者的肿瘤谱系、组织病理学、表达分析和/或肿瘤DNA或RNA分析,因此需要一个标准的CRPC模型来进行药物和放疗疗效筛查[21]。不同于其他肿瘤细胞,前列腺癌细胞不易获取,来源单一,最常见的前列腺癌模型是PC-3、DU145和LNCaP,均来源于转移癌,既不能涵盖全部前列腺癌,也不能代表个体原代前列腺癌细胞[22]。
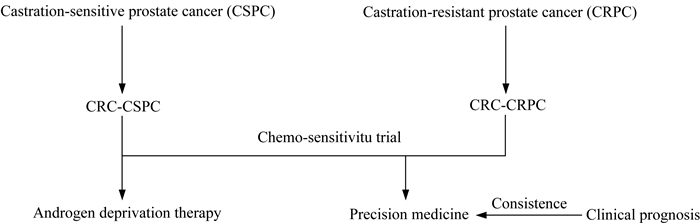
|
| 图 4 CR培养在CRPC中的应用 Fig 4 Application of CR culture in CRPC |
人类前列腺的上皮组织有3种类型:分泌性上皮细胞、基底细胞和神经内分泌细胞[23]。分泌性上皮细胞是柱状上皮细胞,含有前列腺特异性抗原、前列腺酸性磷酸酶和细胞标记物,如细胞角蛋白18(cytokeratin 18,CK18)和雄激素受体(androgen receptor,AR)。基底细胞位于分泌性上皮细胞下层,表达标记物包括CK5和CK14,仅表达低水平AR,基底细胞具有干细胞功能[24]。神经内分泌细胞是罕见的细胞类型,表达内分泌标志物,如突触素和嗜铬蛋白A,而不表达AR。建立和长期维持来源于患者的前列腺肿瘤组织样本的体外培养一直难以实现。近年来CR技术的诞生使原代细胞体外培养有了突破性的进展,生成了患者来源的去势敏感的前列腺肿瘤到去势抵抗的前列腺肿瘤的CRC,以及正常前列腺组织来源的CRC作为对照组[25]。通过qRT-PCR和流式细胞术比较源于患者肿瘤病理标本的CRC和已建立的前列腺癌细胞模型LNCaP,所有CRC都表达高水平的基底细胞标记物CK5、CK14和TP63,几乎不表达分泌性上皮细胞标记物(如AR和CK18),蛋白质表达水平与LNCaP相似,用基因组学、蛋白组学对这些CRC进一步确认,表明CR技术成功培养了细胞模型。通过CR技术获取的CRC-CRPC用于药物敏感试验,分别使用306种临床或新兴抗肿瘤药物,分成5种浓度阶梯,在72 h后检测细胞的生存率,以确定肿瘤细胞对药物的敏感性[26-27],显示出几种潜在抗肿瘤药物的疗效,如紫杉烷类、甲帕克林、奥沙利铂和韦纳托克拉司。最有效的是Navitoclax(ABT-263),它与bcl-2家族成员过表达和诱导细胞凋亡有关。该研究建立的CRC培养体系既验证了新兴的抗前列腺癌药物的疗效,也为CRPC新药的重新定位提供了依据。CR技术可以用来对前列腺癌进行大范围的药物筛查,以获取最好的药效学(图 4)。
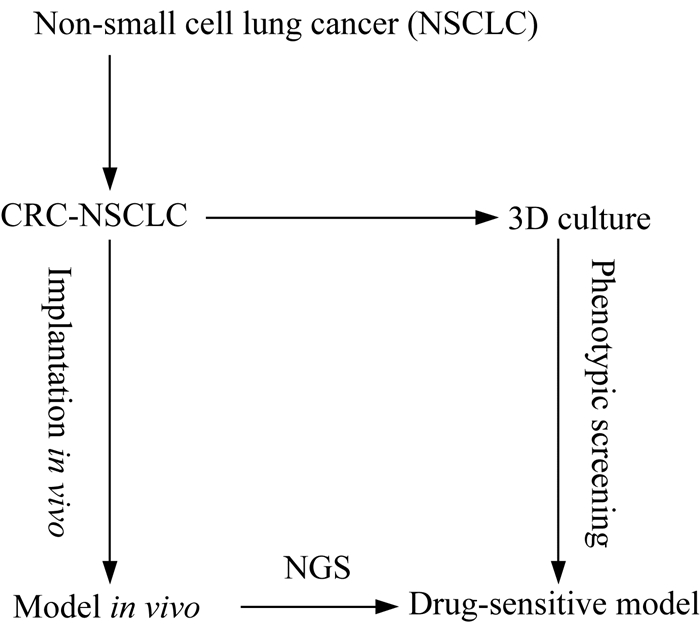
CR用于人类NSCLC建模 NSCLC占肺癌总数13%,NSCLC占肺癌总数87%。肺癌诊断时多数为中晚期,约18%选择手术治疗,62%采取放化疗。化疗是治疗恶性肿瘤最重要的手段之一,肿瘤细胞对化疗药物产生耐药常常导致化疗失败。临床前试验有助于新药的开发和个体化治疗。目前的NSCLC临床前模型不能很好地反映患者肿瘤内和肿瘤间的异质性,因此以细胞为基础的NSCLC体外模型成为研究热点。Hynds等[28]分享了从12例肺腺癌患者(Ⅱ期9例,Ⅲ期3例)中获得原发性NSCLC细胞培养的经验,以及如何提高NSCLC体外培养的成功率。
利用CR技术对NSCLC原代细胞与小鼠成纤维细胞J3共培养(加入ROCK抑制剂),获得10组(83.3%)原代上皮细胞培养物。为了验证CR技术培养的原代肿瘤细胞是否被正常基底细胞污染,对其中4个细胞株进行3D培养分化,结果显示细胞形成管腔,形成了含有分化的气道上皮细胞类型的上皮球状物,提示在肿瘤原代细胞培养中扩增了正常气道基底细胞,并过度增殖。通过向免疫缺陷小鼠注射1×106细胞悬液进行高通量测序,仅1例表现原代肿瘤细胞特性,表明原代肿瘤细胞培养中NSCLC样本被正常基底细胞所污染。因此,CR技术用于NSCLC培养的关键在于分离肿瘤细胞和正常基底细胞。除疾病阶段对细胞培养成功率有影响外,肿瘤基因型可能也是培养成功的决定因素之一。在进行肺部肿瘤原代培养时应彻底分离肿瘤细胞和正常基底细胞,以防在细胞培养过程中因正常基底细胞过度生长而抑制原代肿瘤细胞生长。CR技术有望作为建立NSCLC模型的新手段(图 5)。
|
| 图 5 CR培养在NSCLC中的应用 Fig 5 Application of CR culture in NSCLC |
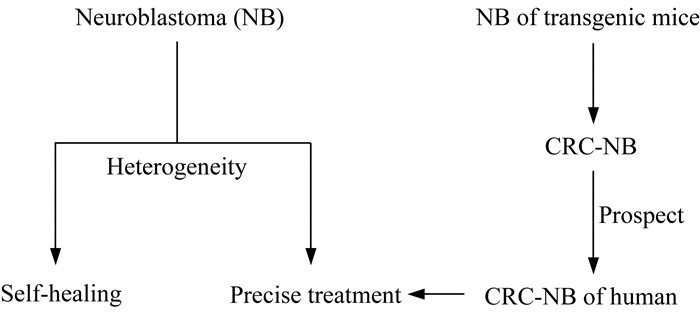
CR培养的小鼠神经母细胞瘤保留了原代肿瘤的异质性(图 6) 神经母细胞瘤是婴幼儿最常见的颅外肿瘤,属于神经内分泌肿瘤,起源于交感神经系统的任意神经脊部分,常见发生部位是肾上腺。近一半的神经母细胞瘤发生于2岁以内的婴幼儿。不同神经母细胞瘤患者预后差别很大,约一半的神经母细胞瘤患者尽管接受了强化的多模式治疗,但预后不佳,而其他患者肿瘤可自行消退或分化为良性神经节神经瘤[29],即神经母细胞瘤间存在广泛的肿瘤异质性。因此,需要准确的临床前模型测试新的治疗方法。CR技术可以利用患者来源的少量细胞进行快速繁殖,并维持肿瘤细胞的异质性,克服传统细胞系的缺点。现阶段CR技术成功建立了以TH-MYCN小鼠转基因纤维母细胞瘤模型中分离出来的肿瘤细胞株为原代细胞的细胞系(图 6),保留了悬浮生长的能力,并成功传代、冷冻和生物包埋,细胞系维持了原代肿瘤细胞的异质性,有利于解决成纤维母细胞瘤患者中广泛存在的肿瘤间异质性的问题[30]。
|
| 图 6 CR培养在NB中的应用 Fig 6 Application of CR culture in NB |
CR的优缺点 CR能快速有效地体外培养原代细胞,并且保持染色体组型的完整性,只需要微量细胞量即可建系。CR能迅速将患者肿瘤相关信息提供给临床,达到精准的抗肿瘤药物敏感性筛查以及免疫治疗。对患者肿瘤穿刺标本进行CR培养,短时间内就能提供精确的治疗方案,即个体化医疗。CR还可以用于扩增癌旁组织(或癌前病变组织)、明确癌症侵袭范围及提前发现癌症。
CR技术用于3T3小鼠成纤维细胞引起的人基质细胞的生长,以至于无法评估人基质细胞对肿瘤生长发育及药物敏感性的影响。其次,Y-27632可能潜在干扰肿瘤细胞的迁移实验和入侵试验。第三,人类活组织标本中含有正常细胞和肿瘤细胞,在CR培养时正常细胞可能生长快于肿瘤细胞,甚至侵占整个培养基。最后,滋养细胞与目标细胞共培养存在污染目标细胞的可能。
结语 CR技术作为一门新兴方法,相比于传统细胞培养能够快速、有效地建立细胞系,为基础和临床试验提供系统的分子模型,有助于从细胞分子角度了解肿瘤的发病机制。科学家致力于建立人类肿瘤样本来源相关的成纤维细胞模型,以期为基础研究和转化研究提供一个更具潜力的癌症模型系统,用以捕获肿瘤细胞群和基质成分(成纤维细胞)的相互作用。目前正在尝试用CR技术解决疾病,如通过基因编辑技术修复CRC,校正导致疾病的缺陷基因,再将修复的细胞导入机体,重新生长得到这些校正的健康细胞,由于源自自身组织,避免了免疫排斥。
| [1] |
CUNDERLIKOVA B. Issues to be considered when studying cancer in vitro[J]. Crit Rev Oncol Hematol, 2013, 85(2): 95-111.
[DOI]
|
| [2] |
ALI SH, DECAPRIO JA. Cellular transformation by SV40 large T antigen:interaction with host proteins[J]. Semin Cancer Biol, 2001, 11(1): 15-23.
[DOI]
|
| [3] |
SHAY JW, WRIGHT WE. Senescence and immortalization:role of telomeres and telomerase[J]. Carcinogenesis, 2005, 26(5): 867-874.
[DOI]
|
| [4] |
LIU X, ORY V, CHAPMAN S, et al. ROCK inhibitor and feeder cells induce the conditional reprogramming of epithelial cells[J]. Am J Pathol, 2012, 180(2): 599-607.
[URI]
|
| [5] |
ALAMRI AM, KANG K, GROENEVELD S, et al. Primary cancer cell culture:mammary-optimized vs conditional reprogramming[J]. Endocr Relat Cancer, 2016, 23(7): 535-554.
[DOI]
|
| [6] |
JENSEN TJ, FOSTER C, SAYEJ W, et al. Conditional reprogramming of pediatric human esophageal epithelial cells for use in tissue engineering and disease investigation[J]. J Vis Exp, 2017(121).
[URI]
|
| [7] |
TIMOFEEVA OA, PALECHOR-CERON N, LI G, et al. Conditionally reprogrammed normal and primary tumor prostate epithelial cells:a novel patient-derived cell model for studies of human prostate cancer[J]. Oncotarget, 2017, 8(14): 22741-22758.
[DOI]
|
| [8] |
MONDAL AM, ZHOU H, HORIKAWA I, et al. Delta133p53alpha, a natural p53 isoform, contributes to conditional reprogramming and long-term proliferation of primary epithelial cells[J]. Cell Death Dis, 2018, 9(7): 750.
[DOI]
|
| [9] |
SUPRYNOWICZ FA, UPADHYAY G, KRAWCZYK E, et al. Conditionally reprogrammed cells represent a stem-like state of adult epithelial cells[J]. Proc Natl Acad Sci U S A, 2012, 109(49): 20035-20040.
[DOI]
|
| [10] |
YUE J, SHUKLA R, ACCARDI R, et al. Cutaneous human papillomavirus type 38 E7 regulates actin cytoskeleton structure for increasing cell proliferation through CK2 and the eukaryotic elongation factor 1A[J]. J Virol, 2011, 85(17): 8477-8494.
[DOI]
|
| [11] |
CORREA BRS, HU J, PENALVA LOF, et al. Patient-derived conditionally reprogrammed cells maintain intra-tumor genetic heterogeneity[J]. Sci Rep, 2018, 8(1): 4097.
[URI]
|
| [12] |
CASSIDY JW, CALDAS C, BRUNA A. Maintaining tumor heterogeneity in patient-derived tumor xenografts[J]. Cancer Res, 2015, 75(15): 2963-2968.
[DOI]
|
| [13] |
BORODOVSKY A, MCQUISTON TJ, STETSON D, et al. Generation of stable PDX derived cell lines using conditional reprogramming[J]. Mol Cancer, 2017, 16(1): 177.
[DOI]
|
| [14] |
CHEN W, ZHENG R, BAADE PD, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115-132.
[DOI]
|
| [15] |
BERDIK C. Unlocking bladder cancer[J]. Nature, 2017, 551(7679): S34-S35.
[DOI]
|
| [16] |
JIANG S, WANG J, YANG C, et al. Continuous culture of urine-derived bladder cancer cells for precision medicine[J]. Protein Cell, 2019, 10(12): 902-907.
[DOI]
|
| [17] |
KETTUNEN K, BOSTROM PJ, LAMMINEN T, et al. Personalized drug sensitivity screening for bladder cancer using conditionally reprogrammed patient-derived cells[J]. Eur Urol, 2019, 76(4): 430-434.
[DOI]
|
| [18] |
SU S, DI POTO C, ROY R, et al. Long-term culture and characterization of patient-derived primary hepatocytes using conditional reprogramming[J]. Exp Biol Med (Maywood), 2019, 244(11): 857-864.
[DOI]
|
| [19] |
MILLER KD, NOGUEIRA L, MARIOTTO AB, et al. Cancer treatment and survivorship statistics, 2019[J]. CA Cancer J Clin, 2019, 69(5): 363-385.
[DOI]
|
| [20] |
BAHNSON RR. Value in prostate cancer treatment[J]. J Surg Oncol, 2016, 114(3): 288-290.
[DOI]
|
| [21] |
KODACK DP, FARAGO AF, DASTUR A, et al. Primary patient-derived cancer cells and their potential for personalized cancer patient care[J]. Cell Rep, 2017, 21(11): 3298-3309.
[DOI]
|
| [22] |
PEEHL DM. Primary cell cultures as models of prostate cancer development[J]. Endocr Relat Cancer, 2005, 12(1): 19-47.
[DOI]
|
| [23] |
ITTMANN M. Anatomy and histology of the human and murine prostate[J]. Cold Spring Harb Perspect Med, 2018, 8(5): a030346.
[DOI]
|
| [24] |
ISAACS JT, COFFEY DS. Etiology and disease process of benign prostatic hyperplasia[J]. Prostate Suppl, 1989, 2: 33-50.
[PubMed]
|
| [25] |
SAEED K, RAHKAMA V, ELDFORS S, et al. Comprehensive drug testing of patient-derived conditionally reprogrammed cells from castration-resistant prostate cancer[J]. Eur Urol, 2017, 71(3): 319-327.
[DOI]
|
| [26] |
YADAV B, PEMOVSKA T, SZWAJDA A, et al. Quantitative scoring of differential drug sensitivity for individually optimized anticancer therapies[J]. Sci Rep, 2014, 4: 5193.
[PubMed]
|
| [27] |
PEMOVSKA T, KONTRO M, YADAV B, et al. Individualized systems medicine strategy to tailor treatments for patients with chemorefractory acute myeloid leukemia[J]. Cancer Discov, 2013, 3(12): 1416-1429.
[DOI]
|
| [28] |
HYNDS RE, AISSA ABEN, GOWERS KHC, et al. Expansion of airway basal epithelial cells from primary human non-small cell lung cancer tumors[J]. Int J Cancer, 2018, 143(1): 160-166.
[URI]
|
| [29] |
DUBOIS SG, PARK JR. Neuroblastoma and histone demethylation[J]. N Engl J Med, 2018, 379(15): 1476-1477.
[DOI]
|
| [30] |
KRAWCZYK E, HONG SH, GALLI S, et al. Murine neuroblastoma cell lines developed by conditional reprogramming preserve heterogeneous phenotypes observed in vivo[J]. Lab Invest, 2020, 100(1): 38-51.
[DOI]
|
 2020, Vol. 47
2020, Vol. 47


