急性呼吸窘迫综合征(acute respiratory distress syndrome,ARDS)是由非心源性肺水肿引起的急性呼吸衰竭综合征。引起ARDS的最常见的临床疾病是脓毒症,在剧烈炎症应激反应刺激下,机体多个器官受累,其中最常见的脓毒症相关功能障碍器官为肺,导致炎症相关肺损伤,进而恶化为ARDS [1-2]。ARDS的特征是对肺泡-毛细血管屏障的急性和弥漫性炎症损伤,血管通透性增加,肺组织顺应性降低,损害气体交换。在组织病理学上,这种情况被称为弥漫性肺泡损伤,包括对肺泡上皮细胞和毛细血管内皮细胞的永久性损伤,随后形成透明膜影响肺的通气和换气[3]。2020年以来COVID-19大流行导致ARDS增加,对ARDS治疗带来挑战[3-4]。
细菌或者病毒感染导致的脓毒症相关ARDS促进机体免疫系统迅速应答以对抗疾病的进展[5]。目前国内外研究大多关注脓毒症相关ARDS在急性期(即24 h)的免疫浸润和基因表达变化,对于随着病情迁延进展,免疫细胞和基因表达发生变化的研究较少。本研究旨在基于生物信息学方法分析Gene Expression Omnibus(GEO)数据库中脓毒症相关ARDS患者入院时和1周后的芯片表达数据,研究外周血免疫细胞变化,鉴定差异表达基因(differentially expressed gene,DEG),探索脓毒症相关ARDS患者的免疫细胞组成特点及其在病情变化中作用,为ARDS患者的免疫治疗手段提供新的依据和方向。
材料和方法数据集获取及分组 GEO数据库是美国国家生物技术信息中心(NCBI)平台下最大的完全公开的高通量分子信息数据库[6]。其发表的数据包括基于微阵列研究的mRNA、蛋白质分子以及基于非阵列研究的基因表达序列和质谱蛋白质[6]。从GEO数据库GSE32707获取数据集,基于GPL10558平台下载矩阵信息。对该数据集入院第0天脓毒症(n=18)和脓毒症相关ARDS(n=18)患者,入院第7天脓毒症(n=13)和脓毒症相关ARDS(n=13)的患者分别进行数据分析。我们根据2016年Surviving Sepsis Campaign的建议[7],将脓毒症定义为:对于感染乃至疑似感染的患者,若患者序贯器官衰竭评分(sequential organ failure assessment,SOFA)变化程度≥2分,表示存在器官功能障碍。根据ALI/ARDS的柏林诊断标准[1],ARDS由以下参数定义:(1)机械通气和呼气末正压或持续气道正压≥5 cmH2O(1 cmH2O=0.098 kPa,下同);(2)胸片或CT上双侧浸润急性发作;(3)重度(PaO2/FiO2≤100 mmHg)、中度(PaO2/FiO2=100~200 mmHg)或轻度(PaO2/FiO2=200~300 mmHg,1 mmHg=0.133 kPa,下同);(4)无胸水、肺塌陷、肺部结节或心源性肺水肿。脓毒症相关ARDS的诊断需同时满足脓毒症和ARDS的诊断标准。
免疫细胞组成分析 每个患者的外周血应用CIBERSORT得到每个样本22种免疫细胞的含量矩阵,筛选出P<0.05的样本。使用R软件的pheatmap包、vioplot包将脓毒症患者和脓毒症相关ARDS患者的免疫细胞组成呈现出来,进行主成分分析(principal component analysis,PCA),并用ggplot2包将结果可视化呈现。
筛选DEGs 使用Perl软件对GEO芯片数据进行注释,将平台文件探针矩阵转换为基因名称。使用R软件,加载limma包,以校正P<0.05、|logFC|>1为标准筛选差异基因,使用pheatmap包和ggplot2包绘制热图和火山图。
DEG关联分析 利用STRING数据库,将筛选所得的DEG输入数据库,通过蛋白质相互作用(protein protein interaction,PPI)网络,筛选联合评分>0.4的高置信度互作节点数据,隐藏无关联的基因,绘制基因关联图。以联合评分>0.4的高置信度进行筛选旨在提高基因关联的可靠性。
富集分析 为了分析差异基因参与的信号途径及可能的功能作用,利用R软件的clusterPronler包对其进行基因本体论(Gene ontology,GO)和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)富集分析。
蛋白质相互作用网络构建及分析 将构建得到的PPI网络导入Cytoscape 3.9.1软件,使用分子复合物检测分析插件(MCODE)构建核心模块,然后采用cytoHubba插件以MCC、MNC和DMNC 3种算法取交集获得核心基因。
统计学分析 所有统计均用R软件(版本4.1.3)完成。第0天、第7天脓毒症与脓毒症合并ARDS两组患者的免疫细胞浸润和差异基因表达采用独立样本t检验。使用双侧检验,P<0.05为差异有统计学意义。
结果数据集介绍 GSE32707数据集由美国哈佛大学医学院布莱根妇女医院朱迪·安·豪瑞拉克教授在2011年10月9日上传,最近更新于2020年12月7日。研究招募了4组接受机械通气的插管受试者:仅脓毒症(sepsis)、脓毒症+ARDS(se/ARDS)、全身炎症反应综合征(systemic inflammatory response syndrome,SIRS)以及非脓毒症、SIRS或ARDS的受试者。在入院当天(第0天)和7天后从患者身上采集血液。从全血样品中分离RNA并制备微阵列。本研究主要对该数据集中第0天脓毒症(n=18)和脓毒症相关ARDS(n=18)患者,第7天脓毒症(n=13)和脓毒症相关ARDS(n=13)的患者分别进行免疫细胞浸润分析和DEG分析。
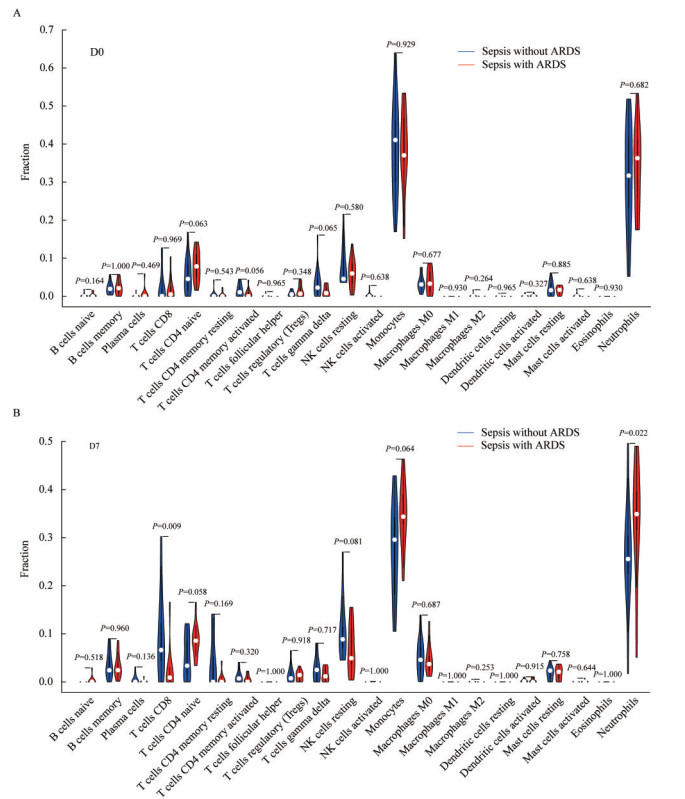
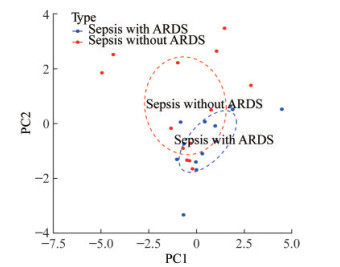
免疫细胞浸润变化 第0天脓毒症和脓毒症相关ARDS的免疫细胞分布无明显差异(图 1A),但是在第7天脓毒症相关ARDS外周血里的CD8+T淋巴细胞比例(2.13%±1.23%)较脓毒症患者(9.32%±2.42%)明显下降(P=0.009),中性粒细胞占比ARDS患者(33.65%±3.31%)较单脓毒症患者(26.27%±3.02%)明显升高(P=0.022)(图 1B)。PCA结果显示:第7天脓毒症及脓毒症相关ARDS的外周血免疫细胞组成有较大差异(图 2)。
|
| A: Percentage of immune cells on admission; B: Percentage of immune cells 1 week after admission. 图 1 不同时间点脓毒症患者和脓毒症相关ARDS患者的22种免疫细胞占比变化 Fig 1 Changes in the proportion of 22 immune cells in sepsis patients and sepsis-related ARDS patients at different time points |
|
| 图 2 第7天脓毒症患者和脓毒症相关ARDS患者的免疫细胞组成的PCA分析 Fig 2 PCA analysis of immune cell composition in sepsis patients and sepsis- related ARDS patients on day 7 |
关键基因变化 鉴于第7天脓毒症患者和脓毒症相关ARDS的免疫细胞组成有明显差异,本研究筛选出2组患者基因芯片数据中34个差异基因表达(图 3),其中表达上调的基因有10个,表达下调的基因有24个。

|
| A: DEGs are shown by heatmap. The horizontal coordinates represent each sample. Red represents up-regulated genes and blue represents down-regulated genes. B: DEGs are shown by Volcano plot. Red represents up-regulated genes and green represents down-regulated. C: Correlation between DEGs. 图 3 第7天脓毒症患者和脓毒症相关ARDS患者的DEGs Fig 3 DEGs in sepsis patients and sepsis-related ARDS patients on day 7 |
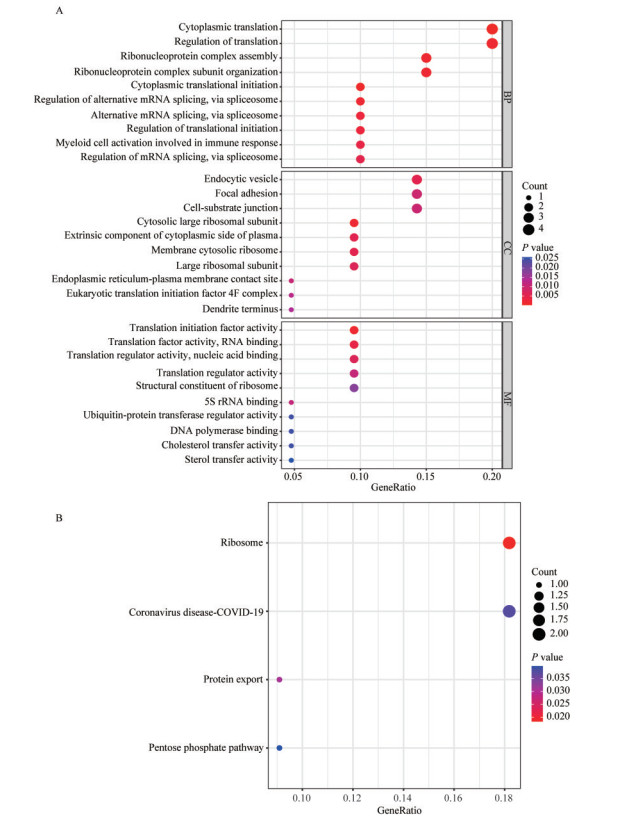
富集通路分析 通过GO分析显示,DEG主要富集的通路是:细胞质翻译的启动及调节、核糖核蛋白复合物组装及亚单位组织、通过剪接体调节选择性mRNA剪接、通过剪接体选择性剪接mRNA及相关调节,翻译起始的调节、参与免疫反应的髓细胞活化、内吞囊泡、黏着斑、细胞-基质连接、胞质大核糖体亚基、细胞质膜细胞质侧的外在成分、胞质核糖体、大核糖体亚基、内质网-质膜接触部位、真核翻译起始因子4F复合物、树突末端、翻译起始因子和结合因子的活性、翻译调节活性,核酸结合、核糖体的结构成分、5S rRNA结合、泛素-蛋白质转移酶活性调节、DNA聚合酶结合、胆固醇转移活性、甾醇转移活性(图 4A)。KEGG分析显示,DEG主要富集于表达通路:核糖体、冠状病毒病-COVID-19、蛋白转运、戊糖磷酸途径(图 4B)。
|
| The size of the circle represents the number of genes enriched, the larger the circle, the more genes are enriched. The color represents the degree of enrichment, the redder the color, the higher the degree of enrichment. 图 4 DEGs的GO分析(A)和KEGG通路分析(B) Fig 4 GO analysis (A) and KEGG pathway analysis (B) of DEGs |
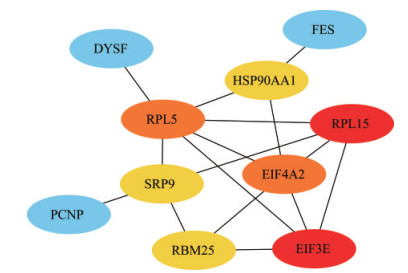
差异基因表达的蛋白相互作用 使用Cytoscape构建可视化核心模块,将10个DEG之间的相关性呈现出来,节点颜色按度值大小由浅至深排列(图 5)。通过MCC、MNC和DMNC 3种算法取交集获得14个DEG,其中RPL5、EIF4A2、RPL15、EIF3E、SRP9、HSP90AA1、RBM25这7个基因为核心基因(hub gene),颜色越红代表该节点的度值越高。
|
| 图 5 蛋白质相互作用网络 Fig 5 Protein interaction network |
ARDS病情进展迅速,尽管采用肺保护性通气策略、俯卧位通气、体外膜肺等多种治疗措施可以改善ARDS患者氧和,减缓病情的恶化,但ARDS患者的死亡率仍然居高不下,轻症住院死亡率为34.9%(95%CI:31.4%~38.5%),中症住院死亡率为40.3%(95%CI:37.4%~43.3%),重症46.1%(95%CI:41.9%~50.4%)[8-9]。
第0天时脓毒症患者和脓毒症相关ARDS患者免疫细胞亚群无明显差异,到第7天脓毒症相关ARDS患者的CD8+T淋巴细胞明显下降,中性粒细胞表达明显升高,推测在炎症信号的刺激下,机体中性粒细胞活跃增殖以向肺微血管系统、间质和肺泡腔中迅速积聚[10]。ARDS患者的中性粒细胞主要吞噬、杀伤、防御病原微生物,同时释放许多抗菌因子,例如活性氧、蛋白酶和中性粒细胞胞外酶。这些因素对宿主细胞也有毒性作用,往往加剧周围肺组织损伤。因此,ARDS患者中性粒细胞的过度积累可能导致疾病进展[11]。
本研究提示随着ARDS病情的进展以细胞杀伤为代表的CD8+T淋巴细胞数量下降。当具有重要免疫防御作用的CD8+T淋巴细胞减少时,宿主对微生物吞噬杀伤能力下降,免疫防御机制进一步受损,更易加重ARDS病情的进展[12]。研究发现呼吸道病毒(流感病毒、H1N1等)感染会抑制CD8+T淋巴细胞潜在的抗病毒潜能,其可能的机制是通过上调树突状细胞上的抑制分子PD-L1,这在抑制CD8+T淋巴细胞活性的情况下尤为重要[13]。树突状细胞上的PD-L1与CD8+T细胞上的PD-1相互作用导致T淋巴细胞衰竭并促进呼吸道病毒诱导的肺损伤[13-15]。此外,病毒感染还可直接诱导CD8+T淋巴细胞上的PD-1表达,从而导致功能障碍,如溶细胞活性降低(如穿孔素/颗粒酶产生)和关键细胞因子(如IL-2、TNF-α和IFN-γ)分泌下降[13]。
为了进一步分析在ARDS病变进展时中性粒细胞升高、CD8+T淋巴细胞减少与基因表达的相关性,我们利用蛋白互作网络分析DEG得到RPL5、EIF4A2、RPL15、EIF3E、SRP9、HSP90AA1、RBM25这7个核心基因。RPL5和RPL15基因表达的蛋白都是核糖体的组成部分。核糖体蛋白RPL5是一种负责细胞内蛋白质合成的大型核糖核蛋白复合体,参与促发核仁应激,抑制细胞增殖,本研究脓毒症合并ARDS患者中RPL5表达下降,推测其可能促进中性粒细胞增殖[16-17]。RPL15是核糖体大亚基60S的组成部分,参与核糖体亚基的组装过程并参与rRNA的加工[14]。在肿瘤条件下,RPL15基因表达下降会诱导损伤相关分子模式的分泌并促进CTL细胞增加,由此推测RPL15基因可能参与脓毒症合并ARDS患者CD8+T淋巴细胞数量的调节[18]。EIF4A是DEAD-box蛋白家族中最小的蛋白[23],eIF4A2优先在增殖能力低的组织中表达,推测可能参与脓毒症相关ARDS患者外周血CD8+T淋巴细胞耗竭的过程[24]。EIF3e是翻译起始因子家族的重要成员,是EIF3复合物的一个组件,EIF3e通过诱导细胞周期停止凋亡来沉默细胞的增殖,因此本研究中推测该基因可能参与中性粒细胞浸润的调控[19]。信号识别颗粒(signal recognition particle,SRP)促进蛋白质通过内质网膜或进入内质网膜的共翻译易位,并具有伸长阻滞功能。SRP9是SRP的6个蛋白质亚基之一,通过与SRP14形成异二聚体结构而发挥伸长阻滞活性[25]。动物研究提示,SRP基因表达会随着急性肺损伤病情的好转而下降[26]。本研究中,脓毒症合并ARDS患者HSP90AA1基因编码热休克蛋白90α(HSP90α)在脓毒症时往往表达增多,严重脓毒症和SIRS中细胞外HSP90α增加与急性炎症代谢应激反应诱导的多器官功能衰竭相关[20]。研究表明人类T淋巴细胞中在炎症刺激下,细胞因子IL-2、IL-4和IL-13可以显著提高HSP90α的表达水平[21]。然而,表达增多的HSP90α通过促进STAT1的去磷酸化途径刺激CD8+T淋巴细胞表达耗竭相关分子PD-1,促进CD8+T淋巴细胞的耗竭,导致CD8+T淋巴细胞数量减少[22]。RNA结合基序蛋白25(RBM25)是一种公认的剪接因子,在真核生物谱系中高度保守,是多种人类细胞系增殖所必需的蛋白。高通量测序显示,RBM25促进在整个人类基因组中包含至少20%的选择性剪接盒外显子,其活性是基本细胞功能所必需的,因此RBM25也是参与脓毒症相关ARDS免疫细胞浸润调控的重要基因[27]。
综上所述,随着病情的进展,脓毒症相关ARDS患者免疫细胞表达模式,相关基因调节会与脓毒症患者有着明显的变化。恰当的免疫治疗可帮助脓毒症相关ARDS机体抵御损伤,减少淋巴细胞耗竭,促进免疫功能恢复,加速疾病的康复。
作者贡献声明 严蕾 论文构思、撰写和修订,数据采集,统计分析。童朝阳 研究构思,论文指导。
利益冲突声明 所有作者均声明不存在利益冲突。
| [1] |
RANIERI VM, RUBENFELD GD, THOMPSON BT, et al. Acute respiratory distress syndrome: the Berlin Definition[J]. JAMA, 2012, 307(23): 2526-2533.
|
| [2] |
HUPPERT LA, MATTHAY MA, WARE LB. Pathogenesis of acute respiratory distress syndrome[J]. Semin Respir Crit Care Med, 2019, 40(1): 31-39.
[DOI]
|
| [3] |
BATAH SS, FABRO AT. Pulmonary pathology of ARDS in COVID-19: a pathological review for clinicians[J]. Respir Med, 2021, 176: 106239.
[DOI]
|
| [4] |
XU Z, SHI L, WANG Y, et al. Pathological findings of COVID-19 associated with acute respiratory distress syndrome[J]. Lancet Respir Med, 2020, 8(4): 420-422.
[DOI]
|
| [5] |
FEIN AM, CALALANG-COLUCCI MG. Acute lung injury and acute respiratory distress syndrome in sepsis and septic shock[J]. Crit Care Clin, 2000, 16(2): 289-317.
[DOI]
|
| [6] |
BARRETT T, SUZEK TO, TROUP DB, et al. NCBI GEO: mining millions of expression profiles--database and tools[J]. Nucleic Acids Res, 2005, 33(Database issue): D562-D566.
|
| [7] |
SINGER M, DEUTSCHMAN CS, SEYMOUR CW, et al. The third international consensus definitions for sepsis and septic shock (Sepsis-3)[J]. JAMA, 2016, 315(8): 801-810.
[DOI]
|
| [8] |
HASAN S S, CAPSTICK T, AHMED R, et al. Mortality in COVID-19 patients with acute respiratory distress syndrome and corticosteroids use: a systematic review and meta-analysis[J]. Expert Rev Respir Med, 2020, 14(11): 1149-1163.
[DOI]
|
| [9] |
BELLANI G, LAFFEY JG, PHAM T, et al. Epidemiology, patterns of care, and mortality for patients with acute respiratory distress syndrome in intensive care units in 50 countries[J]. JAMA, 2016, 315(8): 788-800.
[DOI]
|
| [10] |
SWEENEY RM, MCAULEY DF. Acute respiratory distress syndrome[J]. Lancet, 2016, 388(10058): 2416-2430.
[DOI]
|
| [11] |
WILLIAMS AE, CHAMBERS RC. The mercurial nature of neutrophils: still an enigma in ARDS?[J]. Am J Physiol Lung Cell Mol Physiol, 2014, 306(3): L217-L230.
[DOI]
|
| [12] |
VANDERS RL, MURPHY VE, GIBSON PG, et al. CD8 T cells and dendritic cells: key players in the attenuated maternal immune response to influenza infection[J]. J Reprod Immunol, 2015, 107: 1-9.
[DOI]
|
| [13] |
WHERRY EJ, HA SJ, KAECH SM, et al. Molecular signature of CD8+ T cell exhaustion during chronic viral infection[J]. Immunity, 2007, 27(4): 670-684.
[DOI]
|
| [14] |
SHI R, LIU Z. RPL15 promotes hepatocellular carcinoma progression via regulation of RPs-MDM2-p53 signaling pathway[J]. Cancer Cell Int, 2022, 22(1): 150.
[DOI]
|
| [15] |
ZDRENGHEA MT, JOHNSTON SL. Role of PD-L1/PD-1 in the immune response to respiratory viral infections[J]. Microbes Infect, 2012, 14(6): 495-499.
[DOI]
|
| [16] |
ZHANG H, LIU J, DANG Q, et al. Ribosomal protein RPL5 regulates colon cancer cell proliferation and migration through MAPK/ERK signaling pathway[J]. BMC Mol Cell Biol, 2022, 23(1): 48.
[DOI]
|
| [17] |
MA X, LI Y, ZHAO B. Ribosomal protein L5 (RPL5)/E2F transcription factor 1 (E2F1) signaling suppresses breast cancer progression via regulating endoplasmic reticulum stress and autophagy[J]. Bioengineered, 2022, 13(4): 8076-8086.
[DOI]
|
| [18] |
YAMADA S, KITAI Y, TADOKORO T, et al. Identification of RPL15 60S ribosomal protein as a novel topotecan target protein that correlates with DAMP secretion and antitumor immune activation[J]. J Immunol, 2022, 209(1): 171-179.
[DOI]
|
| [19] |
ZHANG L, WANG X, WU J, et al. MiR-335-3p inhibits cell proliferation and induces cell cycle arrest and apoptosis in acute myeloid leukemia by targeting EIF3E[J]. Biosci Biotechnol Biochem, 2021, 85(9): 1953-1961.
[DOI]
|
| [20] |
FITROLAKI MD, DIMITRIOU H, VENIHAKI M, et al. Increased extracellular heat shock protein 90α in severe sepsis and SIRS associated with multiple organ failure and related to acute inflammatory-metabolic stress response in children[J]. Medicine (Baltimore), 2016, 95(35): e4651.
[DOI]
|
| [21] |
METZ K, EZERNIEKS J, SEBALD W, et al. Interleukin-4 upregulates the heat shock protein HSP90alpha and enhances transcription of a reporter gene coupled to a single heat shock element[J]. FEBS Lett, 1996, 385(1-2): 25-28.
[DOI]
|
| [22] |
LIU K, HUANG J, LIU J, et al. HSP90 mediates IFNγ-induced adaptive resistance to anti- PD-1 immunotherapy[J]. Cancer Res, 2022, 82(10): 2003-2018.
[DOI]
|
| [23] |
LU WT, WILCZYNSKA A, SMITH E, et al. The diverse roles of the eIF4A family: you are the company you keep[J]. Biochem Soc Trans, 2014, 42(1): 166-172.
[DOI]
|
| [24] |
NIELSEN PJ, TRACHSEL H. The mouse protein synthesis initiation factor 4A gene family includes two related functional genes which are differentially expressed[J]. EMBO J, 1988, 7(7): 2097-2105.
[DOI]
|
| [25] |
ERDOĞAN G, TRABULUS DC, TALU CK, et al. Investigation of SRP9 protein expression in breast cancer[J]. Mol Biol Rep, 2022, 49(1): 531-537.
[DOI]
|
| [26] |
CHO WH, KIM YH, HEO HJ, et al. Ginsenoside ameliorated ventilator-induced lung injury in rats[J]. J Intensive Care, 2020, 8(1): 89.
[DOI]
|
| [27] |
CARLSON SM, SOULETTE CM, YANG Z, et al. RBM25 is a global splicing factor promoting inclusion of alternatively spliced exons and is itself regulated by lysine mono-methylation[J]. J Biol Chem, 2017, 292(32): 13381-13390.
[DOI]
|
 2023, Vol. 50
2023, Vol. 50


