2. 复旦大学儿童发育与疾病转化医学研究中心 上海 201102;
3. 上海市出生缺陷防治重点实验室 上海 201102;
4. 国家卫生健康委员会新生儿疾病重点实验室(复旦大学) 上海 201102
2. Translational Medical Center for Child Development and Disease, Shanghai 201102, China;
3. Shanghai Key Laboratory of Birth Defect Prevention and Control, Shanghai 201102, China;
4. National Health Commission Key Laboratory of Neonatal Diseases (Fudan University), Shanghai 201101, China
NPTX1是神经元五聚体(neuronal pentraxins,NPs)蛋白家族中的一员,是一种重要的神经系统分泌蛋白。在神经系统发育分化过程中,干细胞表达或胞外来源的NPTX1可诱导干细胞分化成为神经细胞[1]。NPTX1主要在海马、小脑和大脑皮层神经元中表达[2],参与调控神经突触重塑[3]和神经元凋亡[4]等生物过程。NPTX1突变或表达水平改变可导致多种疾病,如共济失调[5-6]、动眼障碍[7]等,与NPTX1内突变相关。NPTX1在胶质瘤[8]、胰腺癌[9]、肺癌[10]、结直肠癌[11]、宫颈癌[12-13]等肿瘤中表达明显下调,上调NPTX1表达则可抑制该类肿瘤的转移或降低其侵袭性;而在胃癌[14]、胸腺瘤[15]等肿瘤中,NPTX1表达明显上调,并与肿瘤转移或不良预后相关。
增强子是一类可远距离调控基因表达水平的短DNA序列,在调控基因表达的过程中有重要作用[16-18]。增强子调控基因表达的机制之一是被转录因子识别和结合,使得基因表达随细胞内转录因子谱的变化而改变[19],继而影响生物体的发育和疾病的发生发展[20-21]。转录因子通常识别6~12 bp长的DNA序列并结合到DNA上,所以增强子内如有突变,或增强子发生拷贝数变异,该增强子靶向调控的基因的表达模式可能会改变,继而导致发育异常或疾病,如PAX6远端增强子内的单核苷酸突变与虹膜发育不全高度相关,即增强子活性下降,PAX6表达量降低,导致虹膜发育不全[22];SNCA和PTF1A远端增强子的单碱基对点突变可在不引起相关蛋白序列改变的情况下,引起散发性帕金森病和胰腺发育不全[23-24];超级增强子的丢失或获得,也与多种癌症的发生相关[25-26]。NPTX1在多种肿瘤组织中表达下调,其中一种机制是其启动子区域甲基化水平升高[9-11],而NPTX1相关增强子的鉴定及其与疾病的关系则缺少探索和研究。
增强子可通过比较基因组学寻找[27-29]。在VISTA数据库[30]中,搜索NPTX1上下游100 kb范围内的增强子,仅发现1个序列保守区mm2006(chr17:78,488,791-778,490,654),该保守区经实验验证无增强子活性。活性增强子附近富集有几种组蛋白修饰,尤其是p300和CREB结合蛋白(CREB-binding protein,CBP)介导的H3K27乙酰化修饰[31-32]。染色质免疫共沉淀测序技术(chromatin immunoprecipitation sequencing,ChIP-seq)依赖于组蛋白修饰与特定DNA元件的交联,与DNA序列的保守性无直接关系,有助于探索物种间DNA序列不保守的增强子。我们通过分析CistromeDB数据库中多种组织H3K27ac的ChIP-seq数据,在人神经源细胞系SH-SY5Y和人肾源细胞系293T上进行双荧光素酶实验,初步探索了NPTX1附近的增强子及其在不同组织细胞内的活性。
材料和方法仪器设备 二氧化碳细胞培养箱、超净工作台(美国Thermo Fisher公司);PCR仪、离心机(德国Eppendorf公司);Milli-q超纯水系统(德国Millipore公司)。
主要试剂 DMEM培养基、胎牛血清、opti-MEM培养基(美国Gibco公司);Lipo 3000试剂(美国Invitrogen公司);双荧光素酶检测试剂盒(美国Promega公司);2×Taq Mix PCR试剂、无缝克隆试剂盒、质粒小提试剂盒(南京诺唯赞生物有限公司);去内毒素质粒小提试剂盒(美国Omega公司);DH5a感受态细胞(上海擎科生物有限公司)。
实验细胞系 293T细胞系由复旦大学附属儿科医院桂永浩课题组馈赠。SH-SY5Y细胞系由复旦大学附属华山医院董强课题组馈赠。
研究起讫时间 2021年12月31日—2022年11月31日。
ChIP-seq数据获取与分析 从CistromeDB数据库中,下载人不同组织来源H3K27ac ChIP-seq的peak数据文件,使用Wang等[33]构建的ChIP-seq数据分析方法,以脑组织为实验组,以肺、肝、肾等数据为对照组,筛选评分为800以上的脑组织中重要的和特异的H3K27ac富集片段。寻找位于NPTX1上下游100 kb内的H3K27ac富集片段,将这些片段视为假定的NPTX1相关增强子。
富集片段序列保守性分析 使用ECR浏览器,设定DNA分析长度为100 bp,以VISTA数据库中的1个神经系统特异性增强子为参考,对比分析5个H3K27ac富集片段在人(hg19)、小鼠(mm10)和斑马鱼(danRer7)之间的序列相似性。
增强子活性鉴定质粒构建 设计带有酶切位点两侧同源序列的引物,PCR获得两侧带有同源序列的目标片段。纯化回收酶切载体和目标片段,将回收产物通过无缝克隆连接,并转化至DH5a感受态细胞中;挑选菌落,使用去内毒素试剂盒抽提质粒并测序,以验证质粒序列。
细胞转染与双荧光素酶活性检测 取出冻存细胞系,在6 cm细胞培养皿中,使用含10%胎牛血清的DMEM细胞培养液复苏和培养细胞。待细胞密度达到80%~90%时,消化、稀释细胞并接种于24孔板中。接种12 h后,配制转染混合物:取一EP管加入25 μL Opti-MEM,1.5 μL Lipofectamine 3000;取另一EP管加入25 μL Opti-MEM、2 μL P300、500 ng待检测质粒和20 ng pRL-TK质粒。将两个EP管内容物混合,静置10 min,吸取全部混合物加入至24孔板的细胞培养基中。转染24 h后,吸去细胞培养基,使用PBS溶液清洗细胞2次。加入100 μL细胞裂解液,置于摇床30 min后,每孔取50 μL细胞裂解液,转移至96孔酶标板中。使用酶标仪检测,先加入50 μL溶液Ⅰ,检测萤光亮度rum1;再加入50 μL溶液Ⅱ,检测萤光亮度rum2。使用rum1/rum2获得双荧光素酶活性比值rate1。每种待检测质粒做3个复孔。pGL3-E1b为阴性对照组,pGL3-SV40-Promoter为阳性对照组。各孔的rate1除以同批次pGL3-E1b的rate1平均值,即为该孔的双荧光素酶相对活性R。在SH-SY5Y细胞系上检测获得的双荧光素酶相对活性计为RS。每孔的rate1除以同一报告质粒的RS,即为该片段的相对增强子活性。
统计学分析 所有数据使用GraphPad Prism 9.0分析处理,数据结果用x±s表示,两组数据之间比较采用单因素方差分析。P<0.05为差异有统计学意义。
结果人NPTX1两侧100 kb内发现5个H3K27ac富集片段 我们从CistromeDB数据库中下载了多个组织的H3K27ac数据集,使用Wang等[33]开发的增强子分析和评分方法,在NPTX1上下游100 kb的范围内,共发现5个H3K27ac的富集片段(表 1),均位于NPTX1基因两侧(图 1)。将每个富集片段依次命名为NPTX1-E1至NPTX1-E5,其中NPTX1-E1仅为脑组织重要片段,NPTX1-E2为脑组织重要且特异性片段,NPTX1-E3、NPTX1-E4、NPTX1-E5仅为脑组织特异性片段。
| Peak | Chromosomal localization(hg19) |
| NPTX1-E1 | chr17:78389074-78389432 |
| NPTX1-E2 | chr17:78419185-78419616 |
| NPTX1-E3 | chr17:78419703-78419906 |
| NPTX1-E4 | chr17:78427074-78427515 |
| NPTX1-E5 | chr17:78492954-78494359 |
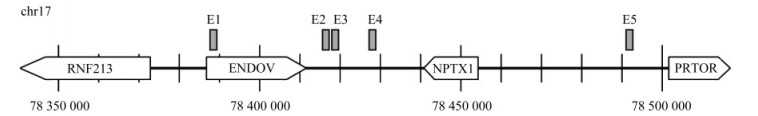
|
| The white arrows in the figure are NPTX1 and the arrow shows the transcriptional direction. The gray boxes are H3K27ac enriched fragments. E1 to E5 correspond to NPTX1-E1 to NPTX1-E5, respectively. 图 1 人(hg19)NPTX1及其两侧的H3K27ac富集片段和其他基因 Fig 1 Human (hg19) NPTX1 with its flanking H3K27ac enriched fragments and nearby genes |
5个富集片段的序列保守性较低 增强子具有一定的生物功能,其在进化过程中受到一定的约束,尤其是对生物发育有重要作用的增强子[34]。借助ECR浏览器,我们评估了5个富集片段在人、小鼠和斑马鱼之间的序列保守性。相对于记录在VISTA增强子数据库[30]中的保守性增强子,NPTX1-E1、NPTX1-E3、NPTX1-E4和NPTX1-E5的DNA序列在人(hg19)、小鼠(mm10)和斑马鱼(danRer7)之间的序列相似性较低(图 2)。NPTX1-E2在人和小鼠间序列相似性为77.7%,但在人和斑马鱼之间相似度低。由此可见5个富集片段在3个物种间的保守性较低。
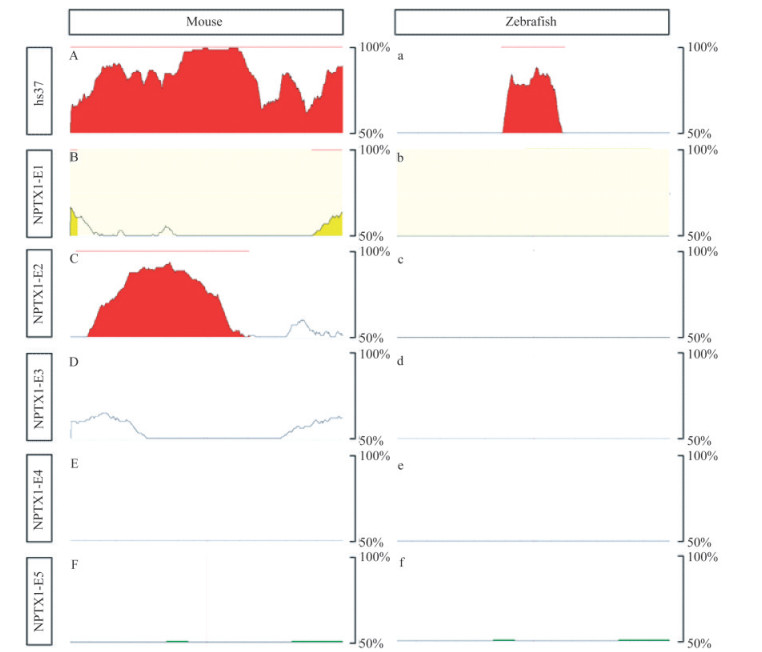
|
| The similarity of DNA sequences between human, mouse and zebrafish was compared by ECR browser. A-F are the results of DNA sequences comparing between human and mouse, and a-f are the results of DNA sequence comparing between human and zebrafish. A and a: A conserved enhancer, hs37, from the VISTA enhancer browser. The red indicates the intervals with more than 70% similarity. 图 2 不同DNA片段在人、小鼠和斑马鱼之间的序列保守性 Fig 2 Sequence conservation of different DNA fragments among human, mouse and zebrafish |
在人神经源细胞系SH-SY5Y中鉴定出1个增强子片段 通过PCR扩增,成功扩增出4个片段,命名为NPTX1-E1-P至NPTX1-E4-P,这些片段包含完整的H3K27ac富集片段及其两侧各500~600 bp的DNA序列。借助无缝克隆技术,成功将4个片段分别插入双荧光素酶验证载体中,在人神经源SH-SY5Y细胞系上检测增强子活性。实验结果显示,在SH-SY5Y细胞系中,NPTX1-E1-P的双荧光素酶相对活性比值明显高于阴性对照组(3.41±0.09 vs. 1.00±0.12,P<0.000 1),而另外3个片段的双荧光素酶活性比值则低于阴性对照组(图 3B),说明NPTX1-E1-P在SH-SY5Y细胞系内可明显上调最小启动子下游的基因表达水平。
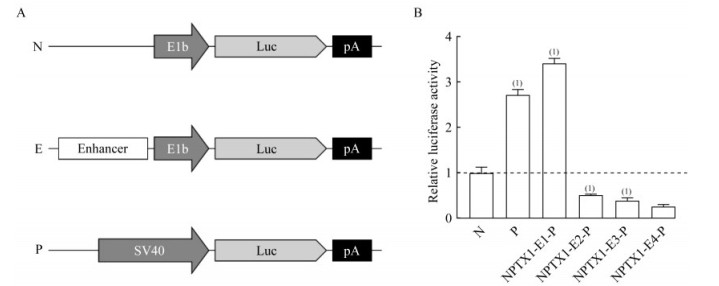
|
| A: Schematic diagram of the enhancer reporter plasmid and the control plasmid. N: Negative control plasmid pGL3-E1b, upstream of Luc contains only the minimal promoter E1b; E: Enhancer reporter plasmid pGL3-enhancer, the DNA fragment to be validated is inserted upstream of E1b; P: Positive control plasmid pGL3-SV40, upstream of Luc contains only the pan-expressing promoter SV40 promoter; E1b: Minimal promoter E1b; Luc: Luciferase; pA: Poly A. B: The relative dual luciferase activity of each assay plasmid was obtained by analyzing the enhancer activity of different fragments by the dual luciferase assay system, using the average of the dual luciferase activity ratio of the negative control plasmid as 1. vs. negative control (N), (1) P < 0.000 1. 图 3 鉴定NPTX1基因附近的增强子 Fig 3 Identification of enhancers near the NPTX1 gene |
NPTX1在不同组织内的增强子活性 为了探索NPTX1-E1-P在不同组织内的增强子活性是否会发生改变,我们进行了双荧光素酶实验,将在人肾源293T细胞上测得的双荧光素酶活性比值记为RT,在人神经源SH-SY5Y细胞上测得的双荧光素酶活性比值记为RS,用RT/RS表示293T细胞系中的相对增强子活性。实验结果显示(图 4),最小启动子载体pGL3-E1b(1.00±0.07 vs. 1.06±0.05,P=0.41)和泛表达启动子载体pGL3-SV40(1.00±0.04 vs. 0.99±0.03,P=0.98)在SH-SY5Y和293T细胞系中的rate1/RS差异无统计学意义,而NPTX1-E1-P在293T细胞中的双荧光素酶活性明显低于SH-SY5Y(0.60±0.05 vs. 1.00±0.05,P<0.000 1),说明NPTX1-E1-P在293T细胞系内的增强子活性明显降低。
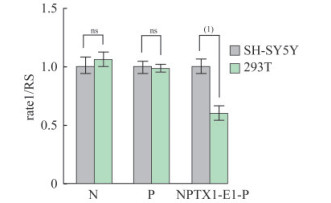
|
| N: Negative control plasmid pGL3-E1b; P: Positive control plasmid pGL3-SV40. RS: Relative dual luciferase activity of the corresponding plasmids obtained by assay on SH-SY5Y;rate1: Relative activity of dual luciferase in each well. ns: Not significant; (1) P < 0.000 1. 图 4 不同细胞系双荧光素酶活性检测 Fig 4 Different cell lines were assayed by dual luciferase activity |
我们在人NPTX1基因两侧识别并鉴定了1个序列保守程度较低的增强子NPTX1-E1-P,该增强子可在人神经来源的SH-SY5Y中上调报告基因的表达水平,且其增强子活性在人肾来源的293T细胞中降低。
NPTX1主要在中枢神经系统细胞中表达[2],在神经系统发育分化[1]、神经突触重塑[3]和神经元凋亡[4]等过程中有重要作用。然而,关于NPTX1的表达调控机制尚不明确。NPTX1的组织特异性表达部分受到增强子的调控,因而有必要鉴定可能调控NPTX1的增强子。在本研究中,增强子NPTX1-E1-P在人神经母细胞来源的细胞系中表现出明显的增强子活性,在人肾上皮细胞来源的细胞系中增强子活性下降,与NPTX1的表达模式有一定的相似之处,说明NPTX1-E1-P在NPTX1的表达调控中可能有一定的作用。
H3K27ac组蛋白标记是增强子活性的特征之一,常用于识别基因组中的增强子[16]。此种方式可以一次获得大量序列,且基本覆盖基因组中的增强子序列,但也囊括大量非增强子序列。在人类胚胎干细胞的研究中,仅有12%的H3K27ac富集片段具有增强子活性[35]。另一项研究显示,即使结合H3K27ac、p300、和RNA聚合酶Ⅱ的ChIP-seq数据,也只有不到10%的假定增强子片段具有增强子活性[36]。因此,尽管我们使用H3K27ac组蛋白标记在NPTX1附近寻找到5个富集片段,在人神经源SH-SY5Y细胞中验证出有增强子活性的却只有1个片段。
增强子具有一定的生物功能,其在进化过程中受到一定的约束,尤其是对生物发育有重要作用的增强子[34]。因此,寻找增强子待定序列的方式之一是寻找目标基因上下游的序列保守区域。Vista数据库记录了大量经过实验验证的保守性增强子[30],在其中搜索到NPTX1上下游100 kb范围内存在1个序列保守区mm2006(chr17:78,488,791-778,490,654,mm10),与我们鉴定的5个H3K27ac富集片段没有交集,且该区域在小鼠体内未检测到增强子活性。因此,我们的工作与Vista浏览器的工作互补,进一步完善了NPTX1附近的调控元件功能注释。
NPTX1的表达水平改变与多种肿瘤及其预后相关[8-10, 14-15],研究显示表达量改变与NPTX1启动子区甲基化水平改变相关[9-11],而其他疾病中NPTX1表达水平改变的机制尚不明确。大量全基因组关联分析(genome wide association study,GWAS)的研究显示,与疾病或特征相关联的变异富集在调控区,尤其是细胞特异性强的增强子区域[25, 37-38]。我们通过鉴定NPTX1附近的增强子,进一步完善了NPTX1附近的调控区功能注释,并探索了与该增强子结合的转录因子,为该区域突变的致病性评估和致病机制探索提供了一定的理论依据。
在本研究中,我们通过增强子与靶基因的距离,初步判断NPTX1-E1-P可能调控人NPTX1的表达。然而,NPTX1-E1-P两侧也有其他基因,距离最近的是ENDOV,其亦可能调控ENDOV的表达。此外,增强子与其靶基因的相互作用并非单纯的一对一的关系,NPTX1-E1-P也可能同时调控多个基因。其他增强子也可能跨越更远的距离(>100 kb)调控NPTX1的表达。HiC、3C等技术可以初步评估该增强子可能的靶基因,但是该技术分辨率较低,神经系统相关的公开数据库较少,未来相关数据公开后将进一步分析研究。亦可通过增强子敲除等技术,进一步确定NPTX1-E1-P的靶基因。
作者贡献声明 陈旭东 数据采集、统计和分析,论文构思、撰写和修订。张琦 数据审核,论文修订。张颖蓝,林佳 实验技术支持。王凤 生物信息学分析技术支持。桂怡婷,刘婷 图片绘制,数据统计和分析,论文修订。李强 项目管理,资金支持,论文修订。
利益冲突声明 所有作者均声明不存在利益冲突。
| [1] |
BOLES NC, HIRSCH SE, LE S, et al. NPTX1 regulates neural lineage specification from human pluripotent stem cells[J]. Cell Rep, 2014, 6(4): 724-736.
[DOI]
|
| [2] |
GUZELOGLU-KAYISLI O, BASAR M, SHAPIRO JP, et al. Long-acting progestin-only contraceptives enhance human endometrial stromal cell expressed neuronal pentraxin-1 and reactive oxygen species to promote endothelial cell apoptosis[J]. J Clin Endocrinol Metab, 2014, 99(10): E1957-E1966.
[DOI]
|
| [3] |
FIGUEIRO-SILVA J, GRUART A, CLAYTON KB, et al. Neuronal pentraxin 1 negatively regulates excitatory synapse density and synaptic plasticity[J]. J Neurosci, 2015, 35(14): 5504-5521.
[DOI]
|
| [4] |
ENGUITA M, DEGREGORIO-ROCASOLANO N, ABAD A, et al. Glycogen synthase kinase 3 activity mediates neuronal pentraxin 1 expression and cell death induced by potassium deprivation in cerebellar granule cells[J]. Mol Pharmacol, 2005, 67(4): 1237-1246.
[DOI]
|
| [5] |
COUTELIER M, JACOUPY M, JANER A, et al. NPTX1 mutations trigger endoplasmic reticulum stress and cause autosomal dominant cerebellar ataxia[J]. Brain, 2021, 145(4): 1519-1534.
|
| [6] |
DEPPE J, DEININGER N, LINGOR P, et al. A Novel NPTX1 de novo variant in a late-onset ataxia patient[J]. Mov Dis, 2022, 37(6): 1319-1321.
[DOI]
|
| [7] |
HELMCHEN C, KOCH PJ, GIRARD G, et al. NPTX1-related oculomotor apraxia: an intra-hemispheric disconnection disorder[J]. J Neurol, 2022, 269(7): 3931-3936.
[DOI]
|
| [8] |
LI H, YAN R, CHEN W, et al. Long non coding RNA SLC26A4-AS1 exerts antiangiogenic effects in human glioma by upregulating NPTX1 via NFKB1 transcriptional factor[J]. FEBS J, 2021, 288(1): 212-228.
[DOI]
|
| [9] |
HAGIHARA A, MIYAMOTO K, FURUTA J, et al. Identification of 27 5' CpG islands aberrantly methylated and 13 genes silenced in human pancreatic cancers[J]. Oncogene, 2004, 23(53): 8705-8710.
[DOI]
|
| [10] |
ZHOU C, QIN Y, XIE Z, et al. NPTX1 is a novel epigenetic regulation gene and associated with prognosis in lung cancer[J]. Biochem Biophys Res Commun, 2015, 458(2): 381-386.
[DOI]
|
| [11] |
MORI Y, OLARU AV, CHENG Y, et al. Novel candidate colorectal cancer biomarkers identified by methylation microarray-based scanning[J]. Endocr Relat Cancer, 2011, 18(4): 465-478.
[DOI]
|
| [12] |
YANG N, EIJSINK JJ, LENDVAI A, et al. Methylation markers for CCNA1 and C13ORF18 are strongly associated with high-grade cervical intraepithelial neoplasia and cervical cancer in cervical scrapings[J]. Cancer Epidemiol Biomarkers Prev, 2009, 18(11): 3000-3007.
[DOI]
|
| [13] |
ONGENAERT M, WISMAN GB, VOLDERS HH, et al. Discovery of DNA methylation markers in cervical cancer using relaxation ranking[J]. BMC Med Genomics, 2008, 1: 57.
[DOI]
|
| [14] |
YAN H, ZHENG C, LI Z, et al. NPTX1 promotes metastasis via integrin/FAK signaling in gastric cancer[J]. Cancer Manag Res, 2019, 11: 3237-51.
[DOI]
|
| [15] |
杜鑫, 于磊, 杨凌, 等. NPTX1在胸腺瘤患者中的表达及诊断价值[J]. 中国肺癌杂志, 2021, 24(1): 1-6. [CNKI]
|
| [16] |
BULGER M, GROUDINE M. Functional and mechanistic diversity of distal transcription enhancers[J]. Cell, 2011, 144(3): 327-339.
[DOI]
|
| [17] |
BUECKER C, WYSOCKA J. Enhancers as information integration hubs in development: lessons from genomics[J]. Trends Genet, 2012, 28(6): 276-284.
[DOI]
|
| [18] |
ONG CT, CORCES VG. Enhancers: emerging roles in cell fate specification[J]. EMBO Rep, 2012, 13(5): 423-430.
[DOI]
|
| [19] |
NATOLI G. Maintaining cell identity through global control of genomic organization[J]. Immunity, 2010, 33(1): 12-24.
[DOI]
|
| [20] |
SAGAI T, HOSOYA M, MIZUSHINAY, et al. Elimination of a long-range cis-regulatory module causes complete loss of limb-specific Shh expression and truncation of the mouse limb[J]. Development (Cambridge, England), 2005, 132(4): 797-803.
[DOI]
|
| [21] |
LEVINE M, TJIAN R. Transcription regulation and animal diversity[J]. Nature, 2003, 424(6945): 147-151.
[DOI]
|
| [22] |
BHATIA S, BENGANI H, FISH M, et al. Disruption of autoregulatory feedback by a mutation in a remote, ultraconserved PAX6 enhancer causes aniridia[J]. Am J Hum Genet, 2013, 93(6): 1126-1134.
[DOI]
|
| [23] |
SOLDNER F, STELZER Y, SHIVALILA CS, et al. Parkinson-associated risk variant in distal enhancer of α-synuclein modulates target gene expression[J]. Nature, 2016, 533(7601): 95-99.
[DOI]
|
| [24] |
WEEDON MN, CEBOLA I, PATCH AM, et al. Recessive mutations in a distal PTF1A enhancer cause isolated pancreatic agenesis[J]. Nat Genet, 2014, 46(1): 61-64.
[DOI]
|
| [25] |
HNISZ D, ABRAHAM BJ, LEE TI, et al. Super-enhancers in the control of cell identity and disease[J]. Cell, 2013, 155(4): 934-947.
[DOI]
|
| [26] |
OOI WF, XING M, XU C, et al. Epigenomic profiling of primary gastric adenocarcinoma reveals super-enhancer heterogeneity[J]. Nat Commun, 2016, 7: 12983.
[DOI]
|
| [27] |
PENNACCHIO LA, AHITUV N, MOSES AM, et al. In vivo enhancer analysis of human conserved non-coding sequences[J]. Nature, 2006, 444(7118): 499-502.
[DOI]
|
| [28] |
NOBREGA MA, OVCHARENKO I, AFZAL V, et al. Scanning human gene deserts for long-range enhancers[J]. Science (New York, NY), 2003, 302(5644): 413.
[DOI]
|
| [29] |
VISEL A, PRABHAKAR S, AKIYAMA JA, et al. Ultraconservation identifies a small subset of extremely constrained developmental enhancers[J]. Nat Genet, 2008, 40(2): 158-160.
[DOI]
|
| [30] |
VISEL A, MINOVITSKY S, DUBCHAK I, et al. VISTA Enhancer Browser--a database of tissue-specific human enhancers[J]. Nucleic Acids Res, 2007, 35(Database issue): D88-D92.
|
| [31] |
HEINTZMAN ND, STUART RK, HON G, et al. Distinct and predictive chromatin signatures of transcriptional promoters and enhancers in the human genome[J]. Nat Genet, 2007, 39(3): 311-318.
[DOI]
|
| [32] |
RADA-IGLESIAS A, BAJPAI R, SWIGUT T, et al. A unique chromatin signature uncovers early developmental enhancers in humans[J]. Nature, 2011, 470(7333): 279-283.
[DOI]
|
| [33] |
WANG F, ZHANG Y, WU F, et al. Functional assessment of heart-specific enhancers by integrating ChIP-seq data[J]. Pediatr Res, 2022, 92(5): 1332-1340.
[DOI]
|
| [34] |
WONG ES, ZHENG D, TAN SZ, et al. Deep conservation of the enhancer regulatory code in animals[J]. Science (New York, NY), 2020, 370(6517): eaax8137.
[DOI]
|
| [35] |
BARAKAT T S, HALBRITTER F, ZHANG M, et al. Functional dissection of the enhancer repertoire in human embryonic stem cells[J]. Cell Stem Cell, 2018, 23(2): 276-288.e8.
[DOI]
|
| [36] |
GASPERINI M, HILL AJ, MCFALINE-FIGUEROA JL, et al. A genome-wide framework for mapping gene regulation via cellular genetic screens[J]. Cell, 2019, 176(1-2): 377-390.e19.
[DOI]
|
| [37] |
KARCZEWSKI KJ, DUDLEY JT, KUKURBA KR, et al. Systematic functional regulatory assessment of disease-associated variants[J]. Proc Natl Acad Sci U S A, 2013, 110(23): 9607-9612.
[DOI]
|
| [38] |
CORRADIN O, SAIAKHOVA A, AKHTAR-ZAIDI B, et al. Combinatorial effects of multiple enhancer variants in linkage disequilibrium dictate levels of gene expression to confer susceptibility to common traits[J]. Genome Res, 2014, 24(1): 1-13.
[DOI]
|
 2023, Vol. 50
2023, Vol. 50


