2. 复旦大学肿瘤研究所 上海 200032
2. Fudan University Shanghai Cancer Institute, Shanghai 200032, China
RNA修饰在基因表达调控中扮演重要角色,哺乳动物中最丰富的mRNA内部修饰是m6A修饰。m6A修饰参与RNA的转录、加工、剪切、翻译和降解过程[1-4],研究显示m6A RNA甲基化与多种癌症的发生发展以及人类多种复杂疾病密切相关[5-7]。
在近年的研究中,最常用的鉴定m6A甲基化方法是m6A免疫沉淀测序(methylated RNA immunoprecipitation sequencing,MeRIP-Seq)和免疫沉淀定位法(m6A individual-nucleotide-resolution cross-linking and immunoprecipitation,miCLIP)[8-9]。然而,这些基于抗体的方法有一些显著的局限性,包括重复性不高[10]、分辨率较低、需要大量样本以及在不同条件下比较m6A甲基化的能力有限,无法精确检测m6A甲基化的变化。
为了在单核苷酸分辨率上检测和量化转录组中m6A的水平,本文根据参考文献[11]进行改进,建立了m6A特异性烯丙基化学标记测序(m6A-selective allyl chemical labeling and sequencing,m6A-SAC-seq)体系,并且保持较好的稳定性和重复性。该方法使用甲基转移酶MjDim1,替换常规的辅酶S-(5’-腺苷)-L-同型半胱氨酸(S-(5′-Adenosyl)-L-homocysteine,SAM)为烯丙基-S-(5’-腺苷)-L-同型半胱氨(allyl-S-(5′-Adenosyl)-L-homocysteine,Allyl-SAM),从而将RNA m6A加上烯丙基,成为am6A,通过I2处理将am6A进行环化,成为N1,N6环化A衍生物,HIV逆转录酶识别该环化产物并在逆转录时引入突变,构建文库,进行高通量测序后通过数据分析来鉴定m6A位点。
材料和方法合成Allyl-SAM 向100 mg SAM中加入1 mL乙酸、1 mL甲酸、52.0 mg高氯酸银和2.125 mL烯丙基溴,20 ℃搅拌8 h。加入20 mL 0.1%三氟乙酸,使反应淬灭。转移水相,用乙醚洗涤,使用反相高效液相色谱法(RP-HPLC)(型号:176803000,美国Waters公司)纯化Allyl-SAM,-80 ℃保存。
m6A RNA探针混合液的制备 设计和合成修饰含有m6A修饰的内参RNA探针。该内参探针由上海捷瑞生物工程有限公司合成,序列中含有NNm6ANN(N为A,U,C和G的混合物)的核心基序,以及特定的8个核苷酸序列构成的“条形码”,分别代表m6A含量为0、25%、50%、75%和100%。由含有m6A修饰的RNA探针和相同序列的未修饰的RNA探针按照特定比例混合而成(附件:表1)。
甲基转移酶MjDim1的制备 合成MjDim1并将其克隆到pET-His-SUMO质粒载体中,转化到T7感受态细胞(编号:C2566I,美国New England BioLabs公司)中,活化菌种过夜,将活化的菌种接种到灭菌的2×YT培养基中,37 ℃培养4 h,降温到16 ℃,继续培养2 h,加入终浓度为0.1 mmol/L的IPTG诱导表达,16 ℃培养16~18 h。收集细胞,在裂解缓冲液(1×PBS+150 mmol/L NaCl)中,用高压细菌破碎仪裂解细胞。使用镍柱亲和纯化后加Ulp1酶,4 ℃过夜,以去除SUMO标签。用洗脱缓冲液[含20 mmol/L Tris-HCl(pH=7.5)、150 mmol/L NaCl和20 mmol/L咪唑]洗脱MjDim1酶。使用AKTA蛋白纯化仪,经阴离子交换色谱(型号:Source15-Q,美国General Electri公司)和阳离子交换色谱(型号:Source15-S,美国General Electric公司)纯化,将蛋白梯度洗脱,采集蛋白峰并浓缩至1.2~2.0 mmol/L,加入终浓度为30%的甘油,于‒80 ℃分装保存。
MjDim1活性测试 将3 μL 20 mmol/L Allyl-SAM,0.6 μL 1 mol/L Tris HCl(pH=8.3)和3 μL纯水混匀,配置成中和Allyl-SAM。用1×结合/清洗缓冲液[含10 mmol/L Tris-HCl(pH=7.5)、1 mmol/L EDTA和2 mol/L NaCl],清洗并重悬磁珠(Dynabeads MyOne Streptavidin C1 beads)(美国Thermo Fisher Scientific公司),混匀后对照组和实验组每管分装50 μL。
在实验组中,4 μL中和Allyl-SAM、1 μL m6A RNA探针混合液(30 ng RNA)、4 μL RNA酶抑制剂(美国Thermo Fisher Scientific公司)、2 μL 10× MjDim1反应缓冲液[含400 mmol/L HEPES(pH=8.0)、400 mmol/L NH4Cl和40 mmol/L MgCl2]、4 μL MjDim1酶和7 μL纯水配置成20 μL反应体系,放入PCR仪中,50 ℃反应1 h。期间每隔20 min,在实验组中加入2 μL 10×MjDim1反应缓冲液、2 μL RNA酶抑制剂、8 μL纯水、4 μL Ally-SAM和4 μL MjDim1酶,50 ℃反应1 h。
在对照组中,将1 μL m6A RNA探针混合液(30 ng RNA)、5 μL RNA酶抑制剂、5 μL 10×MjDim1反应缓冲液和39 μL纯水配置成50 μL反应体系,50 ℃反应2 h。
每组加入1 μL 100 U/μL核酸酶(美国New England BioLabs公司)、2 μL 10×核酸酶缓冲液和14 μL纯水,37 ℃反应过夜。再加入2 μL 1 U/μL碱性磷酸酶(美国Thermo Fisher Scientific公司)和2.4 μL 10× CIP脱磷酸缓冲液,37 ℃反应2 h。使用LC-MS/MS质谱系统进行活性测试。
构建文库
提取样品mRNA 对于细胞系和新鲜冷冻的组织样本,用Trizol法或提取试剂盒纯化全转录组RNA。用Dynabeads mRNA DIRECT试剂盒(美国Thermo Fisher Scientific公司)提取mRNA,用于检测mRNA m6A修饰。
拆卸Poly A并连接接头片段 用oligo-dT与RNA样本的poly A尾巴进行退火,用RNase H(美国New England BioLabs公司)消化,去除poly A尾巴,然后用DNase Ⅰ(美国New England BioLabs公司)去除多余的oligo-dT,用RNA纯化试剂盒(美国Zymo Research公司)纯化。纯化后超声将RNA片段化,37 ℃下用PNK酶(美国New England BioLabs公司)末端修复RNA 30 min,释放3’羟基,以便下一步进行连接反应。在反应中加入m6A RNA探针混合液。使用截短型T4 RNA连接酶2(美国New England BioLabs公司),25 ℃下将RNA片段与3’接头(附件:表1)连接。在混合液中添加1 μL 5’去腺苷化酶(美国New England BioLabs公司),30 ℃反应1 h,去掉多余3’接头上的腺苷化修饰,添加1 μL RecJf(美国New England BioLabs公司),37 ℃孵育1 h,消化剩余的RNA接头片段。加入1 μL 50 μmol/L逆转录引物(附件:表1),75 ℃退火5 min,37 ℃退火15 min,25 ℃退火15 min。
标记m6A位点并进行逆转录 在反应溶液中加入15 μL磁珠纯化RNA。洗净磁珠,用6 μL H2O重悬,RNA在70 ℃ 30 s变性后4 ℃冷却,消除RNA二级结构。添加2 μL 10×MjDim1反应缓冲液(含2 μL RNA酶抑制剂、6 μL Allyl-SAM和4 μL 1.44 mmol/L MjDim1酶),50 ℃反应1 h。
取上清,加入4 μL H2O、1 μL 10×MjDim1反应缓冲液、1 μL RNA酶抑制剂、2 μL Allyl-SAM和2 μL酶,50 ℃孵育20 min。重复以上步骤7次,标记大多数m6A位点。用25 μL水清洗并重悬,在反应体系中加入1 μL 125 mmol/L I2,充分混合。室温暗反应1 h后,加入1 μL 40 mmol/L Na2S2SO3,使I2淬灭。用9 μL纯水清洗并重悬,加入2 μL 10×AMV-RT缓冲液(美国New England BioLabs公司)、2 μL 10 mmol/L dNTP 2、2 μL 25 mmol/L MgCl2、1.25 μL 0.1 mol/L DTT、2 μL RNA酶抑制剂、2 μL HIV-RT酶(美国Worthington Biochemical公司),37 ℃进行3 h的逆转录反应(图 1)。用8 μL纯水清洗并重悬。

|
| m6A-SAC-seq utilizes MjDim1 and Allyl-SAM as a co-factor to convert m6A to am6A, followed by cyclization upon I2 treatment. Mutations are introduced upon reverse transcription at m6A sites. We could identify m6A sites by specific A to C/T mutation locations and the stoichiometry could be quantified by addition of spike-in calibration probes. 图 1 m6A-SAC-seq实验原理图 Fig 1 Schematic diagram of m6A-SAC-seq |
连接cDNA 3'接头片段和构建文库 将1 μL RNase H缓冲液和1 μL RNase H加入逆转录产物中,37 ℃恒温反应30 min。清洗反应液体,用不含核酸酶的H2O重悬,95 ℃反应10 min洗脱cDNA。用DNA纯化试剂盒(型号:D4030,美国Zymo Research公司)纯化到10 μL。向cDNA溶液中添加2 μL 10×T4 RNA连接酶缓冲液、2 μL 10 mmol/L ATP、10 μL 50% PEG8000、1 μL 50 μmol/L cDNA 3’接头片段(附件:表1)和1 μL T4 RNA连接酶1(美国New England BioLabs公司),25 ℃反应过夜(12 h)。用DNA纯化试剂盒纯化cDNA,用21 μL H2O洗脱cDNA。
取1 μL cDNA溶液用于qPCR检测,取15 μL cDNA溶液用作建立文库。qPCR反应体系:1.25 μL 10 μmol/L Universal PCR引物(美国New England BioLabs公司),12.5 μL 10 μmol/L PCR Index引物,1 μL cDNA溶液,1 μL 25×SYBR Green Ⅰ,12.5 μL 2×NEB Next Ultra Ⅱ Q5 Master Mix(美国New England BioLabs公司),加纯水补至25 μL。PCR反应条件:98 ℃ 30 s;98 ℃ 10 s,65 ℃ 75 s,40个循环;65 ℃ 5 min;恢复至4 ℃。通过qPCR的ΔCt值确定最佳建库周期数。
用于建立文库的50 μL PCR反应体系:5 μL 10 μmol/L Universal PCR引物,5 μL 10 μmol/L PCR Index引物,15 μL纯化后的cDNA溶液,25 μL 2×NEB Next Ultra Ⅱ Q5 Master Mix。PCR反应条件:98 ℃ 30 s;98 ℃ 10 s,65 ℃ 75 s,循环数由qPCR结果决定;65 ℃ 5 min;恢复至4 ℃。
使用AMPure XP磁珠(型号:A63880,美国Beckman公司)纯化DNA文库,按照Illumina HiSeq X Ten with paired-end 2×150 bp标准(美国Illumina公司)进行测序。
数据处理
原始序列数据的准备和质量控制 从UCSC基因组浏览器(https://hgdownload.soe.ucsc.edu/goldenPath/hg38/bigZips/)下载人类hg38参考基因组和转录组文件,从UCSC基因组数据库(https://genome.ucsc.edu/cgi-bin/hgTables)下载ncbiRefSeq GTF文件,建立参考基因组(附件:数据1)。从GitHub(https://github.com/CTLife/m6A-SAC-seq)下载脚本,通过FASTQC检查原始FASTQ文件的质量(附件:数据2)。
通过内参探针的标准曲线来换算m6A含量 根据内参探针的m6A含量和测序得到的突变率计算标准曲线(附件:数据3)。换算得到转录组中m6A的含量,检测每个样本的碱基突变率(附件:数据4)。
预处理和序列比对 使用Trimmomatic拆卸低质量的接头和碱基,删除无接头的序列(附件:数据5)。将序列比对到参考基因组,质检后删除低质量序列(附件:数据6)。
突变调用和m6A位点识别 通过SAMtools和VarScan调用每个BAM文件的突变(附件:数据7)。从VarScan的输出中,通过确定最大背景突变率、P值、最小突变率和覆盖率参数来识别m6A位点(附件:数据8)。
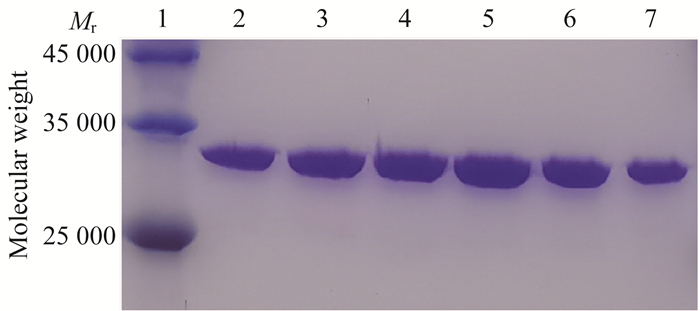
结果甲基转移酶MjDim1的制备 用HPLC法纯化MjDim1蛋白,经过离子交换柱Source-15 Q柱后,使用Source-15 S柱纯化,收集MjDim1蛋白峰(图 2)。SDS-PAGE电泳凝胶显示的纯化蛋白的条带大小符合MjDim1的相对分子质量(32 000,图 3)。最终得到蛋白浓度为1.44 mmol/L的MjDim1酶。

|
| 图 2 MjDim1酶的Source 15-S离子交换纯化曲线 Fig 2 Source 15-S ion exchange purification curve of MjDim1 enzyme |
|
| MjDim1 was purified by Source 15-S resin. Lane 1:A bclonal RM19001 Protein marker; Lane 2-7: Purified MjDim1 enzyme. 图 3 纯化MjDim1酶的SDS-PAGE凝胶电泳照片 Fig 3 SDS-PAGE gel electrophoresis map of purified MjDim1 enzyme |
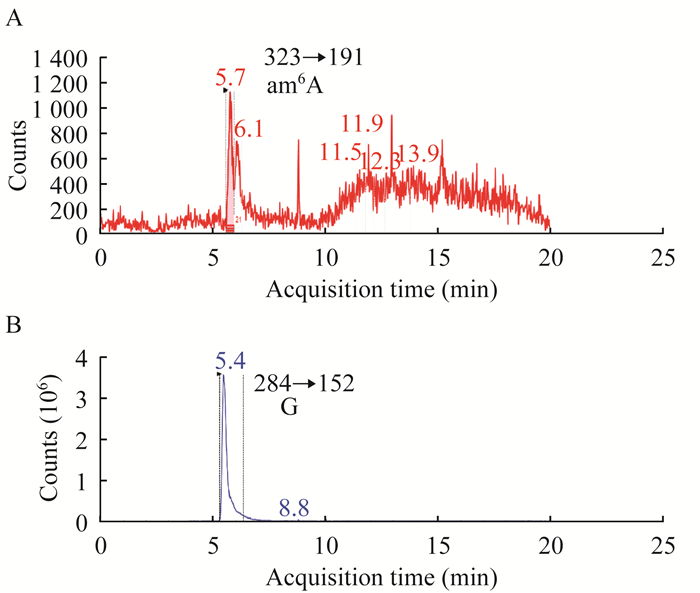
MjDim1活性测试结果 m6A探针经过MjDim1酶在20 μL反应体系中进行烯丙基标记后,生成am6A。am6A的保留时间约为5.5 min(图 4)。统计峰面积得到碱基含量,计算m6A的转换率。m6A的转换率计算公式为:转换率={1-[(Aream6A,treated/AreaG,treated)/(Aream6A,control/AreaG,control)]}×100%。
|
| A: UHPLC-MS/MS quantitation of the m6A probe after allyl labeling using the fresh MjDim1 enzyme. The acquisition time of am6A (N6-allyl, N6-methyladenosine) is around 5.5 min. B: UHPLC-MS/MS quantitation of G after using the fresh MjDim1 enzyme as control. 图 4 m6A探针的质谱结果检测MjDim1活性 Fig 4 Results of MjDim1 activity measured by UHPLC-MS/MS quantitation of the m6A probe |
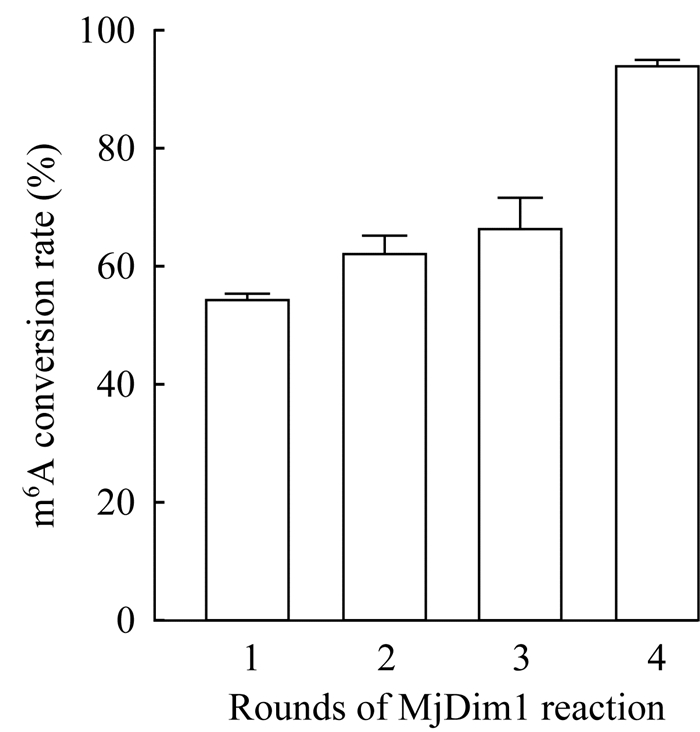
经过MjDim1酶处理不同次数后的m6A转换率随MjDim1酶处理次数增加而上升(图 5),经过4轮MjDim1酶处理,活性检测探针中含有100% m6A的转换率为92.8%(> 90%),说明MjDim1酶的活性处于较高水平,可以在样品反应中使用。
|
| RNA probes with m6A are treated with MjDim1 and Allyl-SAM cofactor for multiple rounds labeling. The conversion rates of m6A to am6A are calculated from three independent biological replicates. 图 5 经MjDim1酶处理不同次数后的m6A转换率 Fig 5 Results of m6A conversion rate after different rounds of treatments with MjDim1 enzyme |
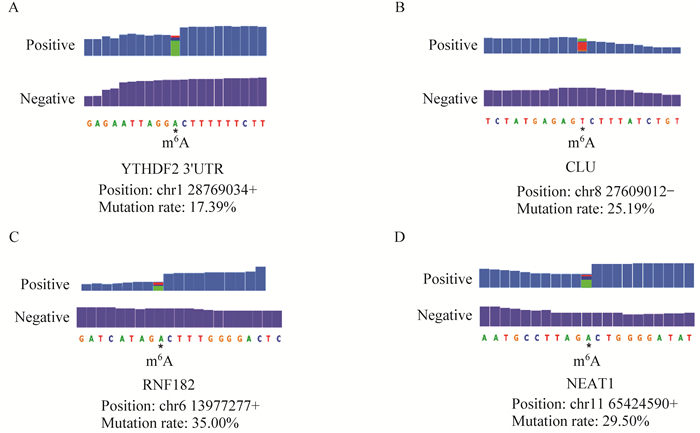
测序分析结果 经过m6A-SAC-seq处理后,逆转录酶会在m6A位点引入突变,但不会在附近的未修饰位点产生突变,可以通过特定的A突变成U或者C的位点来识别m6A位点。在HeLa细胞系中总共检测出10 958个m6A位点(图 6)。以其中4个m6A位点为例,除m6A位点以外,m6A位点附近碱基均未产生突变,并且可以得到该m6A位点的突变率(图 7)。

|
| A: Numbers of m6A sites between biological repeats identified by m6A-SAC-seq using the same RNA sample from HeLa cells; B: Calibration curve for each GGACU motif is generated by linear regression. Goodness of fit (R2) of linear regression was also shown. 图 6 m6A-SAC-seq识别m6A位点并测量m6A含量 Fig 6 Identification of m6A site and measurement of m6A fraction using m6A-SAC-seq |
|
| 图 7 使用m6A-SAC-seq后m6A位点及其附近碱基的突变率 Fig 7 Misincorporation rate of specific m6A sites and the adjacent bases with m6A-SAC-seq |
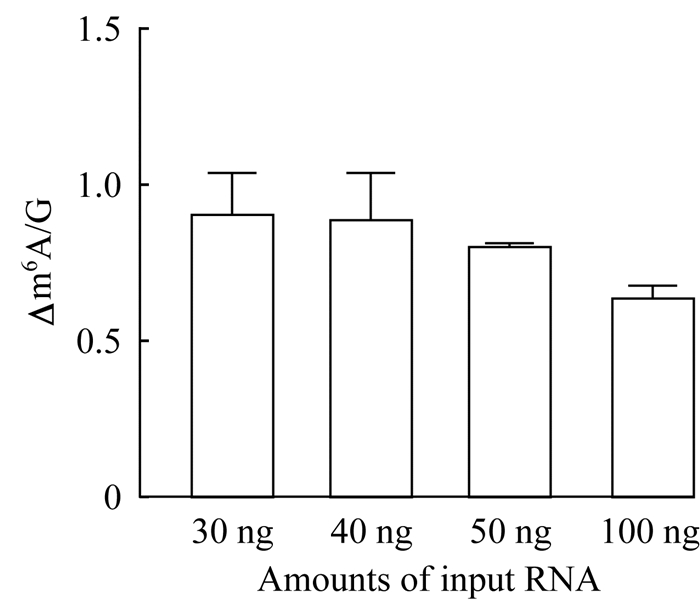
通过LC-MS/MS检测不同RNA起始量的条件下,使用MjDim1标记HeLa mRNA m6A效率(图 8)。RNA起始量在30~100 ng,检测结果的差异不显著。RNA起始量低于30 ng,构建文库需要的PCR循环数比较多,测序的冗余度也会比较高。RNA起始量 > 100 ng,MjDim1酶无法充分标记RNA样本中的m6A。
|
| 图 8 m6A-SAC-seq使用不同RNA起始量的m6A检测效率 Fig 8 Conversion rate of m6A site using different amounts of input RNA by m6A-SAC-seq |
m6A-SAC-seq还可以通过校准探针来量化突变率,从而得到该位点m6A的含量。Chr1:28769034+的YTHDF2 3’UTR上的m6A位点经过m6A-SAC-seq处理后的突变率是17.39%(图 7)。序列中腺嘌呤的突变率和线性相关性可以采用线性回归模型为:Y=aX+b。其中Y为观察到的突变率,X为m6A含量比例,得到相关系数R2=0.9726(图 6B)。经过标准曲线对照后,可得到该位点m6A含量均在25%以上。综上,m6A-SAC-seq仅需30 ng的mRNA样本就可以实现m6A位点在单核苷酸分辨率上的检测。
讨论m6A是真核生物中最常见的一种RNA修饰,富集在终止密码子附近的3'非翻译区[12]。近年来,越来越多的研究发现m6A与癌症相关,能促进癌细胞增殖,并对放疗或化疗产生耐药性[13-15]。m6A甲基化在哺乳动物发育过程中发挥着重要作用,包括胚胎发育、神经发生、性别决定等[16-18]。m6A-SAC-seq具有以下优势。
单核苷酸分辨率,检测精度高 目前普遍使用的MeRIP-Seq技术利用特异性抗体选取携带m6A修饰的RNA片段的免疫沉淀来检测m6A位点,但MeRIP-seq不能达到单碱基分辨率。
m6A-SAC-seq则能通过特异性甲基转移酶,将样品mRNA上的m6A标记上烯丙基形成am6A,通过I2处理生成N1,N6环化的A的衍生物,通过逆转录引入突变,测序后能在单碱基分辨率水平检测整个转录组中的m6A位点,并对其进行定量。相比于依赖抗体的检测方法,m6A-SAC-seq的分辨率高,并且可以精确定量。
送检样本量要求低,检测样本多样 miCLIP技术是目前常用的达到单核苷酸分辨率水平的检测方法,可通过在结合位点交联抗体,在逆转录过程中诱导突变再测序。但是该方法需要更复杂的操作和更多的测序材料,并且在检测峰值和峰值变化方面的可重复性不高。
相比之下,m6A-SAC-seq只需约30 ng mRNA或去除了rRNA的总RNA,突破了样本的取样量瓶颈。通过RNA内参探针绘制标准曲线,可以准确比较不同条件下的m6A含量水平,增加了临床疾病样本取样来源的灵活性。
检测步骤简便,重复性良好 PA-m6A-seq[19]和m6A-CLIP[20]等多种改良的方法提高了m6A位点的检测精度,但上述方法都依赖于m6A特异性抗体,灵敏性、重复性和准确性相对较差。
m6A-SAC-seq中建立文库的操作都在磁珠固相上进行,方便进行多次清洗纯化,减少污染概率。m6A-SAC-seq对m6A的标记依赖于酶的特异性识别,比抗体拥有更好的灵敏性、重复性和准确度。
然而,m6A-SAC-seq技术也存在不足和局限性:由于MjDim1酶偏向于Gm6AC基序,对于Am6AC基序活性较低,未来拟通过改造目前版本的MjDim1甲基转移酶,以期减少其对特定碱基序列检测时的偏好性。
本文描述了m6A-SAC-seq技术的实验操作和数据分析流程,m6A-SAC-seq拥有对各种癌细胞以及临床样本中全转录组m6A进行定位和定量的潜力。我们认为m6A-SAC-seq将有助于更多RNA m6A功能方面的研究,并且在临床上对特定基因的m6A修饰含量进行定量检测,为癌症的早期诊断、治疗和预后提供新的研究策略。
作者贡献声明 孟瀚哲 实验操作,数据统计和分析,论文撰写。何维志 论文指导和修订。胡璐璐 研究构思和设计,论文指导和修订。
利益冲突声明 所有作者均声明不存在利益冲突。
| [1] |
WANG X, ZHAO BS, ROUNDTREE IA, et al. N(6)-methyladenosine modulates messenger RNA translation efficiency[J]. Cell, 2015, 161(6): 1388-1399.
[DOI]
|
| [2] |
WANG Y, LI Y, TOTH JI, et al. N6-methyladenosine modification destabilizes developmental regulators in embryonic stem cells[J]. Nat Cell Biol, 2014, 16(2): 191-198.
[DOI]
|
| [3] |
HUANG H, WENG H, CHEN J. The biogenesis and precise control of RNA m6A methylation[J]. Trends Genet, 2020, 36(1): 44-52.
[DOI]
|
| [4] |
WANG X, LU Z, GOMEZ A, et al. N6-methyladenosine-dependent regulation of messenger RNA stability[J]. Nature, 2014, 505(7481): 117-120.
[DOI]
|
| [5] |
TONG J, FLAVELL RA, LI HB. RNA m6A modification and its function in diseases[J]. Front Med, 2018, 12(4): 481-489.
[DOI]
|
| [6] |
SUN T, WU R, MING L. The role of m6A RNA methylation in cancer[J]. Biomed Pharmacother, 2019, 112: 108613.
[DOI]
|
| [7] |
CUI Q, SHI H, YE P, et al. m6A RNA methylation regulates the self-renewal and tumorigenesis of glioblastoma stem cells[J]. Cell Rep, 2017, 18(11): 2622-2634.
[DOI]
|
| [8] |
MEYER KD, SALETORE Y, ZUMBO P, et al. Comprehensive analysis of mRNA methylation reveals enrichment in 3' UTRs and near stop codons[J]. Cell, 2012, 149(7): 1635-1646.
[DOI]
|
| [9] |
LINDER B, GROZHIK AV, OLARERIN-GEORGE AO, et al. Single-nucleotide-resolution mapping of m6A and m6Am throughout the transcriptome[J]. Nat Methods, 2015, 12(8): 767-772.
[DOI]
|
| [10] |
MCINTYRE A, GOKHALE NS, CERCHIETTI L, et al. Limits in the detection of m6A changes using MeRIP/m6A-seq[J]. Sci Rep, 2020, 10(1): 6590.
[DOI]
|
| [11] |
HU LL, LIU S, PENG Y, et al. m6A RNA modifications are measured at single-base resolution across the mammalian transcriptome[J]. Nat Biotechnol, 2022, 40(8): 1210-1219.
[DOI]
|
| [12] |
KE S, ALEMU EA, MERTENS C, et al. A majority of m6A residues are in the last exons, allowing the potential for 3' UTR regulation[J]. Genes Dev, 2015, 29(19): 2037-2053.
[DOI]
|
| [13] |
YANG B, WANG JQ, TAN Y, et al. RNA methylation and cancer treatment[J]. Pharmacol Res, 2021, 174: 105937.
[DOI]
|
| [14] |
HUANG H, CUI X, QIN X, et al. Analysis and identification of m6A RNA methylation regulators in metastatic osteosarcoma[J]. Mol Ther Nucleic Acids, 2022, 27: 577-592.
[DOI]
|
| [15] |
DAI D, WANG H, ZHU L, et al. N6-methyladenosine links RNA metabolism to cancer progression[J]. Cell Death Dis, 2018, 9(2): 124.
[DOI]
|
| [16] |
NIU Y, ZHAO X, WU YS, et al. N6-methyl-adenosine (m6A) in RNA: an old modification with a novel epigenetic function[J]. Genomics Proteomics Bioinformatics, 2013, 11(1): 8-17.
[DOI]
|
| [17] |
PAN Y, MA P, LIU Y, et al. Multiple functions of m6A RNA methylation in cancer[J]. J Hematol Oncol, 2018, 11(1): 48.
[DOI]
|
| [18] |
PEER E, RECHAVI G, DOMINISSINI D. Epitranscriptomics: regulation of mRNA metabolism through modifications[J]. Curr Opin Chem Biol, 2017, 41: 93-98.
[DOI]
|
| [19] |
CHEN K, LUO G Z, HE C. High-resolution mapping of N6-methyladenosine in transcriptome and genome using a photo-crosslinking-assisted strategy[J]. Methods Enzymol, 2015, 560: 161-185.
|
| [20] |
MOLINIE B, WANG J, LIM KS, et al. m6A-LAIC-seq reveals the census and complexity of the m6A epitranscriptome[J]. Nat Methods, 2016, 13(8): 692-698.
[DOI]
|
 2023, Vol. 50
2023, Vol. 50


