2. 复旦大学生殖与发育研究院 上海 200011
2. Institute of Reproduction and Development, Fudan University, Shanghai 200011, China
慢性肾脏病(chronic kidney disease,CKD)是一种全球流行病,影响着全世界超过10%的人口[1]。肾间质纤维化是CKD进展的共同最终途径,但目前治疗手段仍有限[2]。表观遗传学修饰尤其是DNA甲基化在肾脏纤维化和CKD进展中起重要作用[3-5]。RASAL1基因是一种Ras活性调节基因,其编码蛋白属于Ras GTP酶活化蛋白家族(Ras GTPase-activating protein,Ras GAP)中的GAP1家族[6]。RASAL1能将GTP水解为GDP而使Ras蛋白失活,从而参与细胞的增殖、分化、凋亡。RASAL1低表达在多种恶性肿瘤中的作用已得到广泛研究,而甲基化是RASAL1基因表达调控的重要机制[7]。近年来,RASAL1在脏器纤维化中的作用亦引起重视。既往研究通过肾纤维化的多种实验动物模型、肾活检的小队列人群、人肾脏成纤维细胞的体外培养等,证实RASAL1甲基化及低表达与肾脏纤维化有关[8-11]。这一作用有待于更大范围人群、不同种族尤其是亚洲人群、不同病理类型肾病的验证以及长时间的随访研究。本研究拟通过肾活检的单中心队列,经过平均6.9年的随访,进一步从患者层面验证RASAL1甲基化在肾脏纤维化及CKD进展中的作用。
资料和方法研究设计 本研究为单中心回顾性队列研究,纳入2012年10月至2013年1月在复旦大学附属中山医院肾内科行肾活检病理检查的CKD患者,临床和病理资料完整,临床随访至2021年12月。本研究获得复旦大学附属中山医院伦理委员会的批准(批准号:B2022-041R),入组患者均签署知情同意书。CKD定义为肾脏损伤或肾小球滤过率(glomerular filtration rate,GFR) < 60 mL·min‒1·1.73m‒2持续超过3个月。排除标准:合并恶性肿瘤、活动性感染、3个月内使用过糖皮质激素或免疫抑制剂、吸烟的患者。
一般资料 记录患者肾穿时性别、年龄、肾脏基础疾病及相关疾病(如高血压、糖尿病、自身免疫病、肿瘤等)病史。实验室指标包括血常规、尿常规、肝肾功能、电解质、24 h尿蛋白定量。根据CKD-EPI 2009公式计算估算肾小球滤过率(eGFR)。eGFR=141×min(SCr/κ,1)α×max(SCr/κ,1)‒1.209×0.993Age×1.021(女性)。其中,SCr表示血肌酐(mg/dL),κ=0.7(女性)或0.9(男性),α=‒0.329(女性)或‒0.411(男性)。
病理损伤指标 记录研究对象的肾活检病理诊断。肾小管间质各项病理参数参照Katafuchi评分标准[12],包括有无间质炎性细胞浸润、间质纤维化和肾小管萎缩。根据盲法原则,由一位病理医师独立阅片,阅片人对患者临床资料不知情。
RASAL1基因甲基化检测
标本留取 肾组织来源于肾活检常规病理检查后的剩余组织,外周血单个核细胞(peripheral blood mononuclear cell,PBMC)来源于常规实验室检查后剩余的血细胞部分。
主要步骤 使用DNA提取试剂盒[天根生化科技(北京)有限公司]进行DNA抽提。CpG岛选取及引物设计:RASAL1 PCR引物,F:5'-GGGGG-TATAGATTATGTTTTTTAAGTTTAA-3';R:5'-CAAAAACCTCCCCACAATCC-3'(生物素化)。RASAL1焦磷酸测序引物5'-AGGAAGGGGGTT-TTTTTTTAT-3'。引物由上海生工生物合成。使用Bisul DREAM-Methylation Universal试剂盒(上海奇悟生物科技有限公司)对DNA进行亚硫酸盐处理,使用QIAGEN PyroMark PCR试剂盒(德国QIAGEN公司)对目的片段进行扩增。焦磷酸测序:按照PyroMark Q96测序仪(德国QIAGEN公司)操作说明进行目标序列的甲基化分析。CpG位点的甲基化程度表示为甲基化的胞嘧啶占所有胞嘧啶的比例。分别检测7个CpG岛位点的甲基化水平(+326~+364),这些位点的超甲基化与基因沉默有关[7]。RASAL1的甲基化水平取7个位点的平均值。
随访及结局 记录患者随访过程中的尿蛋白、肾功能、用药情况(是否使用激素/免疫抑制剂)。主要终点:终末期肾病(end stage kidney disease,ESKD,即eGFR < 15 mL·min‒1·1.73 m‒2或肾脏替代治疗;eGFR较基线值下降超过40%。次要终点:eGFR较基线值下降超过20%。33例患者平均随访83(25~103)个月,无死亡病例,无主要终点事件,6例患者达到次要终点。
统计学方法 采用SPSS 20.0统计软件进行统计学处理。计量资料以x±s(正态分布)或M(P25,P75)(非正态分布)表示,计数资料以绝对例数或百分比(%)表示。组间计量资料比较采用t检验,计数资料比较采用χ2检验。采用Spearman分析行单因素相关分析。采用ROC曲线下面积(AUC)评估肾组织RASAL1甲基化水平对肾功能减退的诊断价值,肾组织RASLA1甲基化率的临界值为1.93%。RASAL1甲基化率呈非正态分布,进行自然对数转换后偏态得到纠正,分析中采用对数转换值。采用Kaplan-Meier曲线分析高甲基化组和低甲基化组患者无事件肾脏存活率。P < 0.05为差异有统计学意义。
结果患者基本资料 33例CKD患者,男性18例,女性15例,年龄(39.3±15.2)岁。肾活检病理类型包括IgA肾病14例(42.4%),膜性肾病6例(18.2%),微小病变病5例(15.1%),局灶节段性肾小球硬化3例(9.1%),局灶节段增生性肾炎3例(9.1%),系膜增生性肾炎2例(6.1%)。合并高血压者11例(33.3%),合并糖尿病者1例(3.0%)。患者平均血肌酐(78.6±19.8)μmol/L,eGFR(97.1±19.9)mL·min‒1·1.73m‒2。CKD1期22例(66.7%),2期10例(30.3%),3期1例(3.0%)。分别对患者肾组织及PBMC RASAL1甲基化进行检测,检测7个位点。肾组织各位点RASAL1甲基化率平均值为1.57%(1.14%,2.29%)(表 1)。
| [M(P25, P75)] | |||||||||||||||||||||||||||||
| Position | Renal tissue(%mC) | PBMC(%mC) | |||||||||||||||||||||||||||
| 1 | 0(0,2.00) | 0(0,0) | |||||||||||||||||||||||||||
| 2 | 4.00(3.00,5.50) | 2.00(2.00,2.00) | |||||||||||||||||||||||||||
| 3 | 0(0,0) | 0(0,0) | |||||||||||||||||||||||||||
| 4 | 2.00(2.00,3.00) | 0(0,2.00) | |||||||||||||||||||||||||||
| 5 | 2.00(2.00,2.00) | 0(0,0) | |||||||||||||||||||||||||||
| 6 | 2.00(2.00,2.00) | 0(0,2.00) | |||||||||||||||||||||||||||
| 7 | 0(0,2.00) | 0(0,0) | |||||||||||||||||||||||||||
| Average | 1.57(1.14,2.29) | 0.57(0.57,0.64) | |||||||||||||||||||||||||||
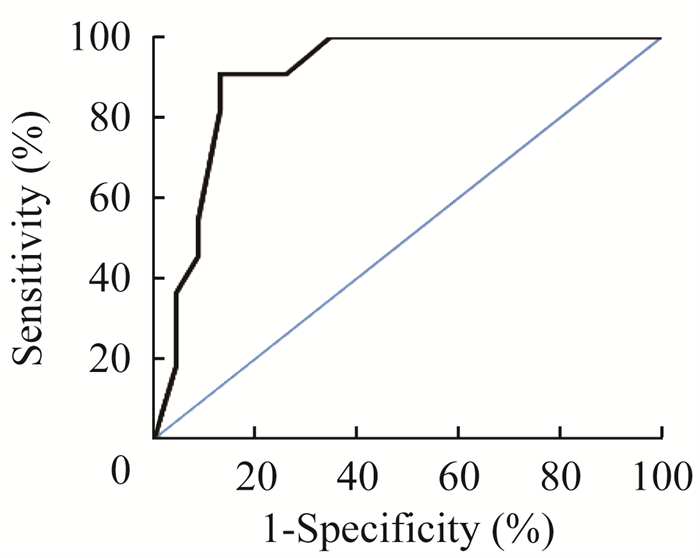
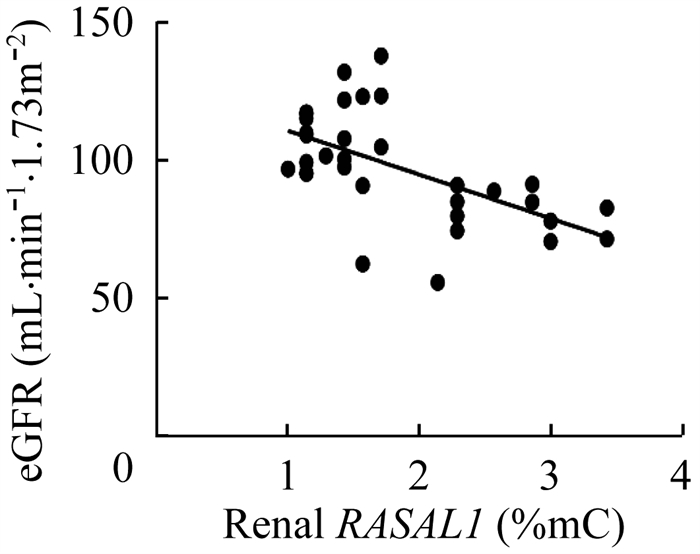
肾组织RASAL1甲基化与eGFR的相关性 将患者分为肾功能正常组(eGFR≥90 mL·min‒1·1.73 m‒2)和肾功能减退组(eGFR < 90 mL·min‒1·1.73 m‒2),肾功能减退组患者肾组织RASAL1甲基化率显著高于肾功能正常组[2.57%(2.29%,3.00%)vs. 1.43%(1.14%,1.61%),t=‒6.577,P < 0.01]。肾组织RASAL1甲基化率评价患者肾功能减退的AUC为0.942(P < 0.01),肾组织RASAL1甲基化率为临界值1.93%时,预测肾功能减退的灵敏度为91%,特异性为87%(图 1)。Spearman相关性分析提示,肾组织RASAL1甲基化率与eGFR负相关(ρ=‒0.598,P < 0.01)(图 2)。
|
| 图 1 肾组织RASAL1甲基化率预测基线肾功能减退的ROC曲线 Fig 1 ROC curve of RASAL1 methylation level in renal tissue to predict renal function impairment |
|
| 图 2 肾组织RASAL1甲基化率与基线eGFR的相关性 Fig 2 Correlation between RASAL1 methylation level in renal tissue and eGFR |
患者外周血PBMC RASAL1甲基化率 33例患者PBMC各位点的RASAL1甲基化率平均值为0.57%(0.57%,0.64%)。Spearman相关性分析提示,PBMC与肾组织RASAL1甲基化率呈正相关(ρ=0.413,P < 0.05)。PBMC RASAL1甲基化率与eGFR负相关(ρ=‒0.590,P < 0.01)。
肾组织RASAL1甲基化与CKD患者肾脏病理损伤的相关性以1.93%为临界值,高甲基化组患者肾间质纤维化比例明显高于低甲基化组患者(P < 0.05,表 2)。
| [n(%)] | |||||||||||||||||||||||||||||
| Index | Hypomethylation group(n=21) | Hypermethylation group(n=12) | χ2/P | ||||||||||||||||||||||||||
| Interstitial fibrosis | 6.303/0.012 | ||||||||||||||||||||||||||||
| Without | 13(61.9) | 2(16.7) | |||||||||||||||||||||||||||
| With | 8(38.1) | 10(83.3) | |||||||||||||||||||||||||||
| Tubular atrophy | 0.171/0.679 | ||||||||||||||||||||||||||||
| Without | 1(4.8) | 1(8.3) | |||||||||||||||||||||||||||
| With | 20(95.2) | 11(91.7) | |||||||||||||||||||||||||||
| Interstitial cell infiltration | 1.229/0.268 | ||||||||||||||||||||||||||||
| Without | 5(23.8) | 1(8.3) | |||||||||||||||||||||||||||
| With | 16(76.2) | 11(91.7) | |||||||||||||||||||||||||||
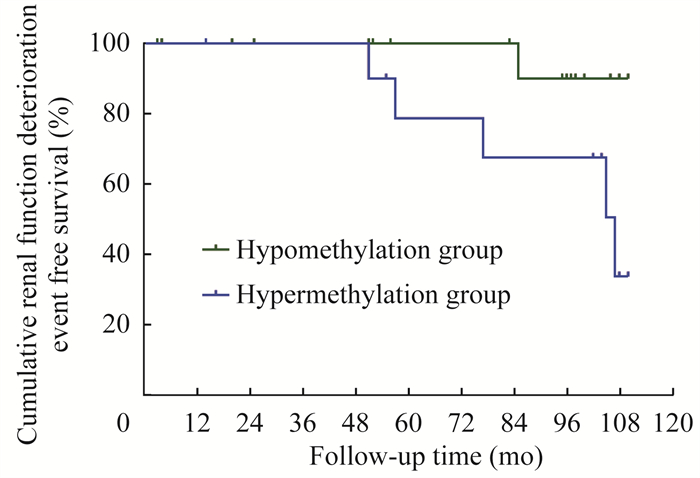
肾组织RASAL1甲基化对CKD患者预后的影响 33例患者平均随访83(25~103)个月,无死亡病例,无主要终点事件,6例患者随访过程中达到次要终点(eGFR较基线下降超过20%)。肾组织RASAL1低甲基化组(n=21)和高甲基化组(n=12)患者的基线和预后资料见表 3。两组分别有1例(n=21)和5例(n=12)的患者达到次要终点(P < 0.01)。Kaplan-Meier曲线分析提示,高甲基化组患者无恶化事件肾脏存活率明显低于低甲基化组患者,但差异无统计学意义(P=0.064,图 3)。
| [x±s, M(P25, P75) or n(%)] | |||||||||||||||||||||||||||||
| Parameters | All (n=33) | Hypomethylation group(n=21) | Hypermethylation group(n=12) | Z,t or χ2 | P | ||||||||||||||||||||||||
| Age(y) | 39.3±15.2 | 36.4±13.4 | 44.2±17.5 | -1.416(2) | 0.167 | ||||||||||||||||||||||||
| Male gender | 18(54.5) | 10(47.6) | 8(66.7) | 1.117(3) | 0.290 | ||||||||||||||||||||||||
| Hypertension | 11(33.3) | 5(23.8) | 6(50.0) | 2.357(3) | 0.125 | ||||||||||||||||||||||||
| Diabetes mellitus | 1(3.0) | 0(0) | 1(8.3) | 1.695(3) | 0.193 | ||||||||||||||||||||||||
| Serum creatinine(μmol/L) | 78.6±19.8 | 70.3±17.6 | 93.0±15.1 | ‒3.744(2) | 0.001 | ||||||||||||||||||||||||
| Serum uric acid(μmol/L) | 380.5±103.5 | 382.8±124.8 | 376.6±53.0 | 0.162(2) | 0.872 | ||||||||||||||||||||||||
| eGFR(mL·min‒1·1.73 m‒2) | 97.1±19.9 | 107.8±16.5 | 79.7±10.3 | 5.318(2) | < 0.001 | ||||||||||||||||||||||||
| Proteinuria(g/24 h) | 1.7(0.8,4.2) | 1.7(0.9,5.8) | 2.4(0.9,3.7) | ‒0.146(2) | 0.885a | ||||||||||||||||||||||||
| Follow up time(mo) | 83(25,103) | 83(22,98) | 90(51,106) | ‒1.087(1) | 0.291 | ||||||||||||||||||||||||
| Glucocorticoid/immunosuppressants | 19(57.6) | 12(57.1) | 7(58.3) | 0.004(3) | 0.947 | ||||||||||||||||||||||||
| Death | 0 | 0 | 0 | / | / | ||||||||||||||||||||||||
| Primary endpoint | 0 | 0 | 0 | / | / | ||||||||||||||||||||||||
| Secondary endpoint | 6(18.2) | 1(4.8) | 5(41.7) | 6.991(3) | 0.008 | ||||||||||||||||||||||||
| (1) Z value;(2) t test;(3) χ2 test. a Data were ln transformed. | |||||||||||||||||||||||||||||
|
| 图 3 CKD患者肾组织RASAL1甲基化水平与肾脏预后的关系(Kaplan-Meier曲线) Fig 3 RASAL1 methylation level in renal tissue and renal prognosis in patients with CKD (Kaplan-Meier curve) |
近年来,表观遗传修饰包括DNA甲基化、羟甲基化、组蛋白修饰等在肾脏纤维化及CKD进展中的作用引起重视,可能作为潜在的治疗靶点[3, 13-15]。既往研究发现RASAL1异常甲基化及低表达参与了包括肾纤维化在内的多种脏器纤维化过程[16-17]。本研究纳入了33例行肾活检的CKD患者,排除了吸烟、恶性肿瘤等可能会影响DNA甲基化水平的情况[18],采用焦磷酸测序法检测肾组织RASAL1基因启动子甲基化水平,横断面分析中,Spearman相关分析提示肾组织RASAL1甲基化率与eGFR呈负相关(P < 0.01)。肾组织RASAL1甲基化率预测基线肾功能减退的AUC为0.942(P < 0.01)。肾脏病理损伤指标分析提示,肾组织RASAL1高甲基化组患者肾间质纤维化比例明显高于低甲基化组患者(83.3% vs. 38.1%,P < 0.05)。提示肾组织RASAL1甲基化与肾间质纤维化及肾功能减退相关,与既往研究结果一致。
既往实验研究表明,RASAL1甲基化是肾纤维化的重要机制。Bechtel等[8]通过活检肾脏分离出人成纤维细胞,体外培养后发现12个甲基化水平差异显著的纤维化相关基因,并通过体内外实验证实,RASAL1的高甲基化可导致RASAL1表达持续性下调,促进成纤维细胞持续活化和肾纤维化。后续研究进一步在肾纤维化的多种动物模型中验证了RASAL1异常甲基化的作用,如单侧输尿管梗阻、叶酸肾病、糖尿病肾病、5/6肾切除、肾缺血再灌注模型等[9-11]。但在CKD患者中,特别是亚洲人群,RASAL1甲基化对肾间质纤维化及慢性肾病进展的作用研究尚少。Tampe等[9]曾通过肾活检的小队列人群(共6例),证实肾间质纤维化的患者存在RASAL1甲基化及mRNA低表达。这一作用有待于更大范围人群、不同种族人群、不同病理类型肾病的验证以及长时间的随访研究。本研究的优势在于纳入了33例CKD患者检测肾组织RASAL1甲基化水平,较以往研究病例数明显增加,经过平均6.9年的随访发现,肾组织RASAL1高甲基化组患者发生eGFR进一步下降的概率高于低甲基化组(P < 0.01)。从患者层面进一步证实了肾组织RASAL1甲基化与肾间质纤维化及肾功能减退相关。目前生存分析的结果无统计学意义,后续进一步扩大样本量,有望得到更有说服力的结论。
既往研究发现,循环中游离RASAL1基因甲基化片段水平与肾脏RASAL1甲基化水平及肾脏纤维化程度正相关,但样本量较小(9例)[10]。另有研究发现,外周血PBMC的RASAL1甲基化水平与镉(具有肾毒性的重金属)污染地区人群的eGFR下降及肾间质小管损伤相关,但无肾组织甲基化数据[19]。本研究同时检测了患者肾组织及PBMC的RASAL1甲基化水平,结果显示:PBMC的RASAL1甲基化水平与肾组织正相关(ρ=0.413,P < 0.05)。评价肾组织病理损伤及肾脏预后方面,肾组织RASAL1甲基化优于PBMC。可能与本研究纳入的CKD患者病情较轻,PBMC的RASAL1甲基化水平普遍偏低有关。总之,DNA甲基化用于肾纤维化预测的无创标志物还有待进一步研究。
在肾小球硬化程度方面,我们发现肾组织RASAL1高甲基化组患者肾活检病理所示肾小球硬化比例高于低甲基化组[0.15(0.07,0.37)vs. 0.05(0,0.08),P=0.036],提示RASAL1甲基化与肾小球硬化存在一定的相关性。但既往实验研究中,RASAL1对肾脏纤维化的影响主要关注于肾小管及间质[11, 17],RASAL1在肾小球硬化中的作用及机制有待进一步研究。
本研究存在以下不足:(1)总样本量偏小,纳入患者以CKD1~2期为主,未纳入肾功能明显减退的患者;(2)缺少肾组织RASAL1转录及表达的数据。
表观遗传改变是可逆的,因此有重要的治疗靶点前景。既往实验研究发现骨形态发生蛋白7、肼苯哒嗪等可通过逆转RASAL1甲基化改善肾纤维化[9, 11]。最近有研究发现基于高保真CRISPR/ Cas9技术的基因特异性双加氧酶,可通过靶向消除RASAL1和Klotho的异常甲基化,改善肾纤维化[20]。因此,RASAL1甲基化在肾脏病领域有广泛的研究前景和重要的临床意义。
综上所述,CKD患者肾脏RASAL1基因甲基化与肾间质纤维化和基线eGFR下降相关。肾脏RASAL1甲基化与随访过程中eGFR进一步下降也存在相关趋势,但未见统计学意义。后续研究中将进一步扩大样本量,分析不同疾病类型、疾病各个阶段RASAL1甲基化的作用。
作者贡献声明 张笛 数据整理和分析,论文撰写。张函 研究构思,数据分析,论文修订。陈静,张晨,沈子妍 数据收集和分析,文献调研。章晓燕 研究构思,论文指导。
利益冲突声明 所有作者均声明不存在利益冲突。
| [1] |
WANG F, HE K, WANG J, et al. Prevalence and risk factors for CKD: a comparison between the adult populations in China and the United States[J]. Kidney Int Rep, 2018, 3(5): 1135-1143.
[DOI]
|
| [2] |
杨梦凡, 范桢亮, 尹日平, 等. 肾小管间质纤维化发病机制的研究进展[J]. 中国中西医结合肾病杂志, 2021, 22(2): 186-188. [CNKI]
|
| [3] |
TAMPE B, ZEISBERG M. Chromatin dynamics at the core of kidney fibrosis[J]. Matrix Biol, 2018, 68-69: 194-229.
[DOI]
|
| [4] |
刘婉婷, 钱诗蕾, 袁小澎. 肾纤维化的表观遗传学研究进展[J]. 广东医学, 2019, 40(9): 1342-1344. [DOI]
|
| [5] |
陈林娜, 张鹏, 王汉民. DNA甲基化与糖尿病肾病的研究进展[J]. 医学研究杂志, 2019, 48(12): 11-13. [CNKI]
|
| [6] |
SOT B, BEHRMANN E, RAUNSER S, et al. Ras GTPase activating (RasGAP) activity of the dual specificity GAP protein rasal requires colocalization and C2 domain binding to lipid membranes[J]. Proc Natl Acad Sci, 2013, 110(1): 111-116.
[DOI]
|
| [7] |
JIN H, WANG X, YING J, et al. Epigenetic silencing of a Ca2+-regulated Ras GTPase-activating protein RASAL defines a new mechanism of Ras activation in human cancers[J]. Proc Natl Acad Sci, 2007, 104(30): 12353-12358.
[DOI]
|
| [8] |
BECHTEL W, MCGOOHAN S, ZEISBERG EM, et al. Methylation determines fibroblast activation and fibrogenesis in the kidney[J]. Nat Med, 2010, 16(5): 544-550.
[DOI]
|
| [9] |
TAMPE B, TAMPE D, MULLER CA, et al. Tet3-mediated hydroxymethylation of epigenetically silenced genes contributes to bone morphogenic protein 7-induced reversal of kidney fibrosis[J]. J Am Soc Nephrol, 2014, 25(5): 905-912.
[DOI]
|
| [10] |
TAMPE B, TAMPE D, ZEISBERG EM, et al. Induction of tet3-dependent epigenetic remodeling by low-dose hydralazine attenuates progression of chronic kidney disease[J]. EBioMedicine, 2015, 2(1): 19-36.
[DOI]
|
| [11] |
TAMPE B, STEINLE U, TAMPE D, et al. Low-dose hydralazine prevents fibrosis in a murine model of acute kidney injury-to-chronic kidney disease progression[J]. Kidney Int, 2017, 91(1): 157-176.
[DOI]
|
| [12] |
KATAFUCHI R, KIYOSHI Y, OH Y, et al. Glomerular score as a prognosticator in IgA nephropathy: its usefulness and limitation[J]. Clin Nephrol, 1998, 49(1): 1-8.
|
| [13] |
KATO M, NATARAJAN R. Epigenetics and epigenomics in diabetic kidney disease and metabolic memory[J]. Nat Rev Nephrol, 2019, 15(6): 327-345.
[DOI]
|
| [14] |
王圆圆, 毛彦稳, 张小欢, 等. 组蛋白去乙酰化酶抑制剂SAHA对糖尿病大鼠肾间质纤维化的影响及机制[J]. 中华肾脏病杂志, 2017, 33(12): 929-934. |
| [15] |
顾玉露, 陈静, 张函, 等. H2S通过诱导Klotho基因启动子去甲基化改善单侧输尿管梗阻(UUO)小鼠肾脏纤维化[J]. 复旦学报(医学版), 2019, 46(3): 294-301. [URI]
|
| [16] |
LIU X, GAO S, XU H. lncRNAPCAT29 inhibits pulmonary fibrosis via the TGFbeta1regulated RASAL1/ERK1/2 signal pathway[J]. Mol Med Rep, 2018, 17(6): 7781-7788.
|
| [17] |
MAO Y. Hypermethylation of RASAL1: a key for renal fibrosis[J]. EBioMedicine, 2015, 2(1): 7-8.
|
| [18] |
SU SF, HO H, LI JH, et al. DNA methylome and transcriptome landscapes of cancer-associated fibroblasts reveal a smoking-associated malignancy index[J]. J Clin Invest, 2021, 131(16): e139552.
|
| [19] |
ZHANG C, LIANG Y, LEI L, et al. Hypermethylations of RASAL1 and KLOTHO is associated with renal dysfunction in a Chinese population environmentally exposed to cadmium[J]. Toxicol Appl Pharmacol, 2013, 271(1): 78-85.
|
| [20] |
XU X, TAN X, TAMPE B, et al. High-fidelity CRISPR/Cas9-based gene-specific hydroxymethylation rescues gene expression and attenuates renal fibrosis[J]. Nat Commun, 2018, 9(1): 3509.
|
 2023, Vol. 50
2023, Vol. 50


