2. 济宁医学院 2018 级临床医学五年制 济宁 272067;
3. 济宁医学院临床医学院外科学总论教研室 济宁 272067
2. Five-year Program of Clinical Medicine, Grade of 2018, School of Clinical Medicine, Jining Medical University, Jining 272067, Shandong Province, China;
3. Department of Pandect Surgury, School of Clinical Medicine, Jining Medical University, Jining 272067, Shandong Province, China
卵巢癌是女性癌症死亡的第五大原因,通常晚期才被发现[1]。流行病学研究表明美国约80%的卵巢癌患者存在局部或远处转移[2]。影响卵巢癌患者预后的主要因素是分期水平,Ⅰ期患者的5年生存率为70%,而Ⅳ期患者的5年生存率仅为8% [3]。因此,识别卵巢癌患者预后的生物标志物和潜在的调节因子具有重要意义。近年来,卵巢癌的靶向治疗和免疫治疗为患者提供了新方向,其目的是通过激活T细胞,调节T细胞浸润水平来延长患者生存时间[4],同时有研究显示免疫治疗是刺激机体对肿瘤的免疫应答[5]。因此,进一步明确肿瘤细胞与免疫细胞之间的相互作用及分子机制,可能为卵巢癌患者的免疫治疗提供新的靶点。
羧基谷氨酸蛋白(matrix gamma linolenic acid protein,MGP)是在骨中发现的一种内源性小蛋白,由软骨细胞分泌到细胞外基质中,在心脏、肾脏中均有表达。MGP通过与羟基磷灰石中的晶核结合,发挥钙化抑制剂的功能,从而阻止晶体生长。MGP也是重要的局部血管钙化抑制剂,可以接受两种转录后修饰:维生素K-dependentγ-谷氨酸羧化和丝氨酸磷酸化[6]。已发现乳腺癌细胞系表达MGP,在乳腺癌MCF-7细胞中降低MGP表达,可导致肿瘤细胞增殖和侵袭能力增强。MGP在其他类型肿瘤中也有表达,如结肠癌、卵巢癌。我们应用生物信息学方法分析了MGP基因在卵巢癌组织中的表达情况,并发现MPG高表达与患者预后差密切相关;同时分析了MGP表达水平与卵巢癌免疫微环境的关系,为进一步研究其在卵巢癌发生发展中的作用和机制提供有价值的依据。
材料和方法TCGA数据库分析 进入TCGA网站(https://www.cancer.gov/about-nci/organization/ccg/research/structural-genomics/tcga),点击Access TCGA Data,进入数据下载页面;点击Repository,在Cases选项中选择ovary,在Program选项中选择TCGA,在Project选项中选择TCGA-OV,在Files选项中选择transcriptome profiling,在Data Type选项中选择Gene Expression Quantification,在Experimental Strategy选项中选择RNA-seq,在Workflow Type选项中选择HTSeq-FPKM,最后点击Add All Files to Cart,进入Cart页面,在Download选项中下载Manifest、Cart、Metadata和Clinical文件。
GTEX数据库分析 基因型组织表达(Genotype-Tissue Expression,GTEX)是一个大型人类组织mRNA数据集,为识别与基因表达变化相关的遗传变异提供参考。通过UCSC Xena官网(https://xena.ucsc.edu/)进入GTEX数据下载页面,下载gene expression RNAseq转录组TOIL RSEM fpkm格式数据和GTEX phenotype临床信息。
TIMER网站分析 进入TIMER官网(https://cistrome.Shinyapps.io/timer)可查看到7个模块。“Diff Exp”基因差异表达模块通过箱线图显示基因在TCGA肿瘤组织和癌旁正常组织中的分布情况,并使用Wilcoxon检验对差异表达进行统计学分析。“Survival”生存模块构建COX比例风险模型,协变量包括年龄、性别、种族、肿瘤分期和基因表达。通过Kaplan-Meier绘制生存曲线,通过log-rank检验比较两组生存曲线的P值。该COX模型由R包“survival”中的函数coxph()拟合;系数coef为回归系数,HR为风险比,95%CI_l和95%CI_u显示上下95%CI。“Mutation”突变模块通过箱线图显示不同基因突变状态下免疫细胞浸润水平的分布,并使用双边Wilcoxon秩和检验进行统计分析。“Correlation”相关性模块通过散点图绘制目标基因在肿瘤中的分布情况,并通过Spearman的rho值评估统计学意义,还提供了以肿瘤纯度或年龄为条件的部分相关选项。
基因集富集分析 GSEA方法是一组具有共同生物学功能、染色体位置或调节通路的基因[7]。本研究根据MGP基因表达水平将卵巢癌样本分为高表达组和低表达组。使用GSEA_4.0.1版本软件,在Gene sets database设置中分别选择c2.cp.kegg.v7.4.symbols.gmt[Curated]和c2.cp.biocarta.v7.4.symbols.gmt[Curated],设置选项中Number of permutations选择1000,Phenotype labes选择h_versus_l。
统计学分析 采用R 3.5.1软件进行统计分析。通过limma包处理转录组数据,通过Wilcoxon检验分析两组间差异。测量数据用x±s表示,计数数据用百分比(%)表示,P < 0.05为差异有统计学意义。
结果MGP在不同类型人类肿瘤中的mRNA表达水平 MGP mRNA在各种肿瘤与邻近正常组织间的表达差异如图 1A所示,在膀胱尿路上皮癌、乳腺癌、结肠癌中MGP表达均低于癌旁组织。由于TCGA数据库中无正常卵巢组织数据,我们结合GTEX数据库发现,MGP在正常卵巢组织中高表达,而在卵巢癌组织中低表达(图 1B)。
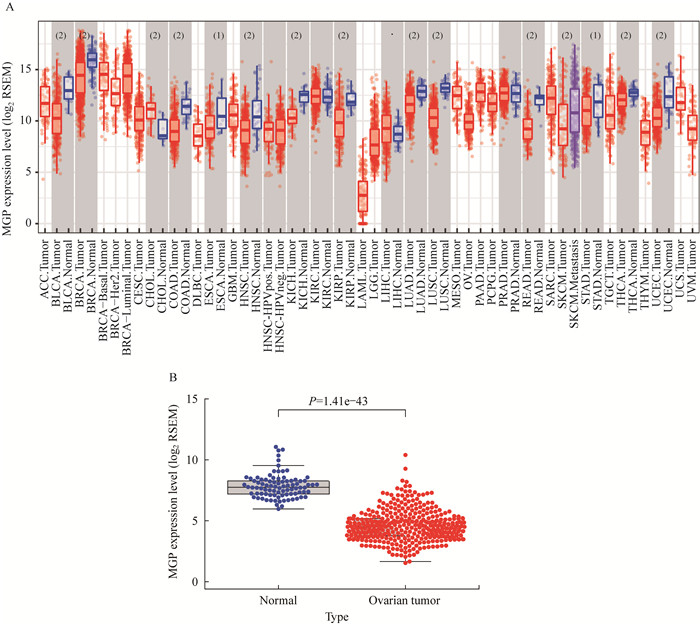
|
| A: MGP mRNA expression in various tumor tissues and adjacent tissues; B: MGP mRNA expression in ovarian cancer and normal ovarian tissue. (1)P < 0.05; (2)P < 0.001. 图 1 MGP在泛癌组织中与正常组织中的表达情况 Fig 1 Expression level of MGP in pan-cancer tissues and normal tissues |
GSEA分析MGP基因在卵巢癌中富集的信号通路 以KEGG基因集作为GSEA分析的参考基因集,MGP高表达组与细胞黏附分子、细胞因子受体相互作用、血管平滑肌收缩、肠道免疫球蛋白IgA、自然杀伤细胞介导细胞毒性等信号通路相关(图 2A)。以BIOCARTA基因集作为GSEA分析的参考基因集,MGP高表达组与CCR5通路、NKT通路、IL-17通路、Th1/Th2等通路有关(图 2B)。表明MGP可能通过上述生物学过程促进卵巢癌的发生发展,特别是与淋巴细胞和细胞因子密切相关。
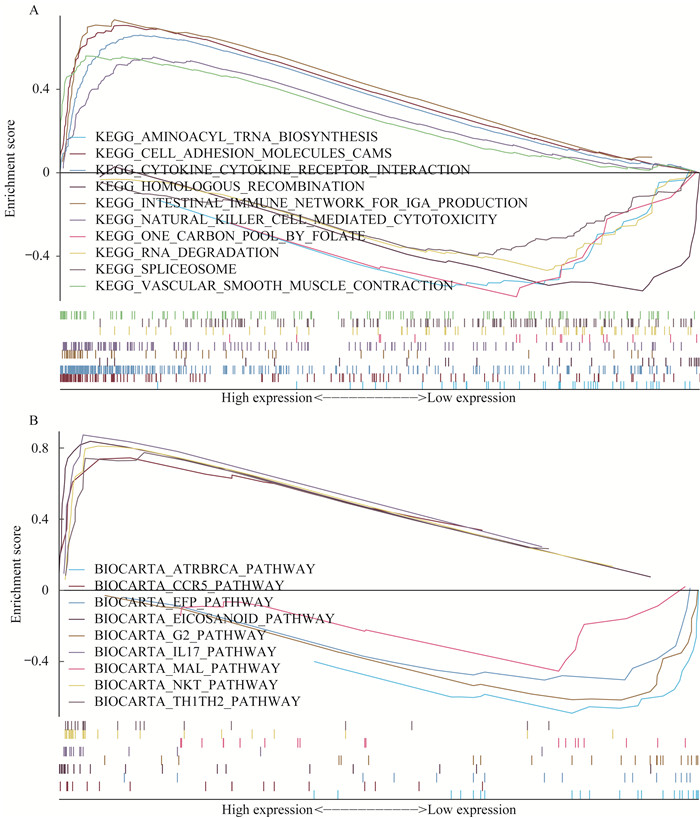
|
| A: MGP enrichment analysis with KEGG gene set as reference gene set; B: MGP enrichment analysis with BIOCARTA gene set as reference gene set. 图 2 GSEA分析MGP基因在卵巢癌中富集的信号通路 Fig 2 GSEA analysis on the enriched signal pathway of MGP gene in ovarian cancer |
MGP与卵巢癌免疫细胞浸润的关系 通过TIMER网站分析MGP表达与卵巢癌微环境中不同类型免疫细胞的相关性:MGP表达水平与CD8+T细胞、巨噬细胞、中性粒细胞呈正相关,而与肿瘤纯度呈负相关(P < 0.05,图 3A)。通过分析MGP基因拷贝数发现,MGP基因缺失与B细胞、CD8+ T细胞、CD4+ T细胞、树突状细胞的浸润水平密切相关(图 3B)。
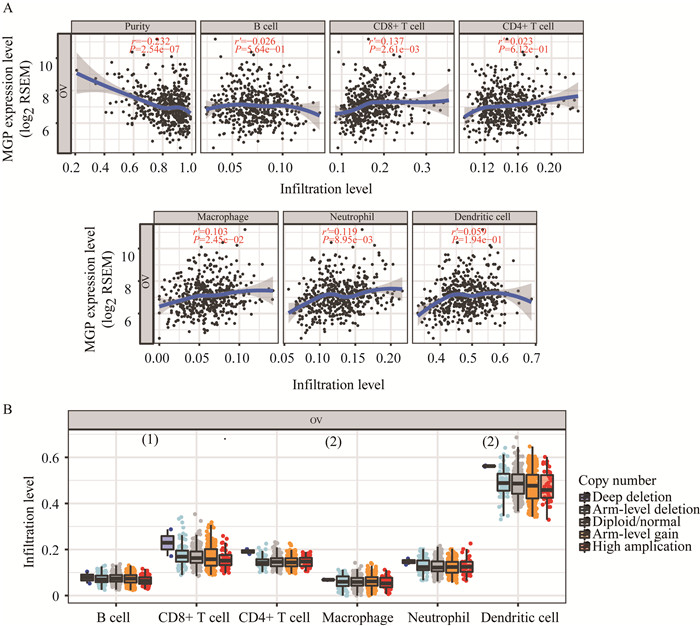
|
| A: Relationship between lymphocyte infiltration and MGP expression; B: Lymphocyte infiltration was correlated with MGP gene mutation.r: Correlation; r': Partial correlation. (1)P < 0.05; (2)P < 0.001. 图 3 MGP表达水平和基因突变与免疫浸润的关系 Fig 3 Relationship between MGP expression level, gene mutation and immune infiltration |
MGP和免疫细胞浸润水平与卵巢癌患者预后的关系 通过TIMER网站生存分析模块,绘制免疫细胞浸润和基因的Kaplan-Meier图,通过log-rank检验比较两组生存曲线的P值。结果显示,卵巢癌患者中CD4+ T细胞、巨噬细胞浸润水平和MGP表达水平与患者预后密切相关(P < 0.05,图 4)。高水平CD4+ T细胞、低水平巨噬细胞和低水平MGP提示卵巢癌患者预后好。
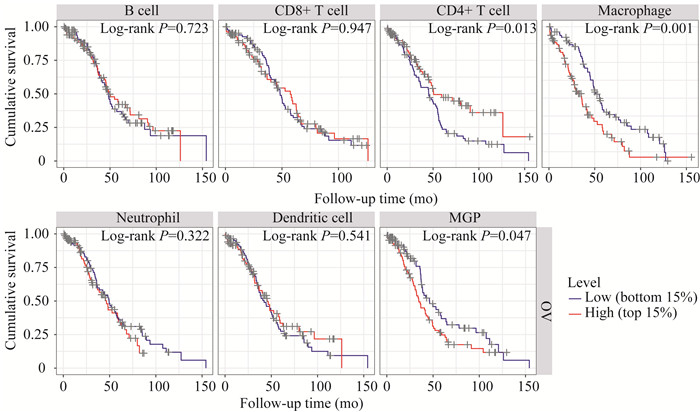
|
| 图 4 MGP和免疫细胞浸润水平与卵巢癌患者预后的关系 Fig 4 Relationship between the level of MGP and immune cell infiltration and the prognosis of patients with ovarian cancer |
MGP是卵巢癌患者的独立预后因子 通过构建COX回归模型发现:患者年龄,巨噬细胞、中性粒细胞浸润水平,MGP表达水平与患者预后呈正相关,而CD8、CD4 T细胞与患者预后呈负相关(P < 0.05,表 1),即MGP高表达组患者预后更差。
| Factors | Coefficient | HR | 95%CI | P |
| Age | 0.024 | 1.025 | 1.014-1.035 | < 0.001 |
| B cell | 1.328 | 3.773 | 0.008-1.806e+03 | 0.673 |
| CD8+ T cell | -4.607 | 0.010 | 0-0.432 | 0.017 |
| CD4+ T cell | -14.249 | 0 | 0-0.001 | < 0.001 |
| Macrophage | 7.772 | 2 373.156 | 9.521-5.915e+05 | 0.006 |
| Neutrophil | 11.264 | 77 956.004 | 16.414-3.702e+08 | 0.009 |
| Dendritic | -1.801 | 0.165 | 0.002-17.299 | 0.448 |
| MGP | 0.127 | 1.136 | 1.011-1.275 | 0.032 |
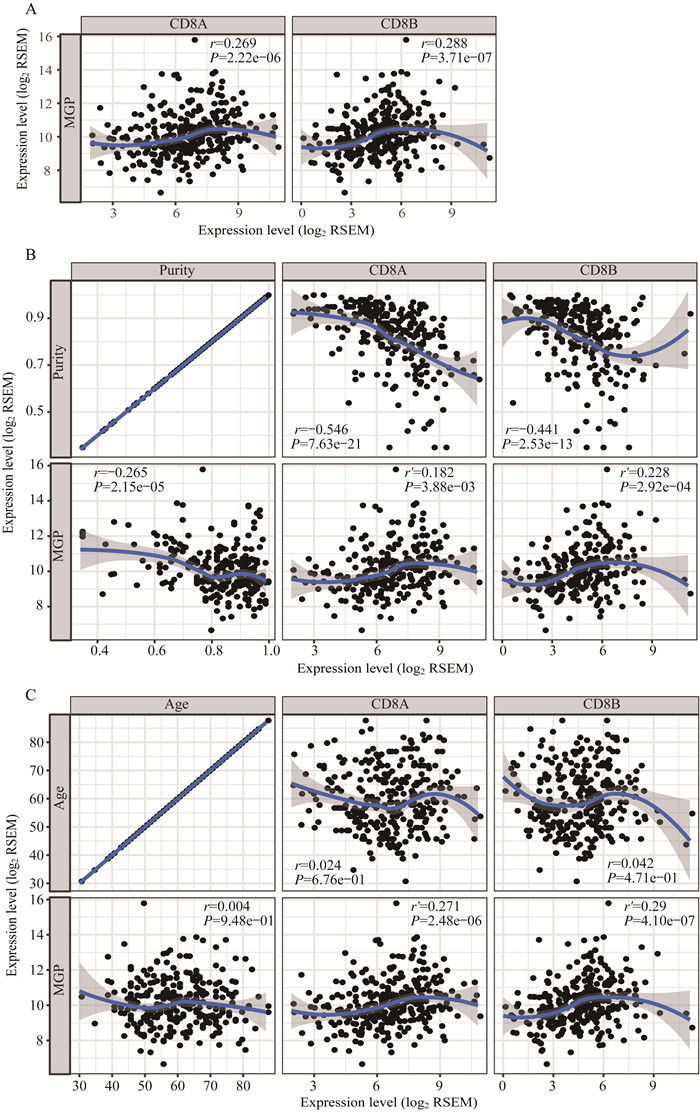
MGP表达与免疫细胞相关基因的关系 通过分析TIMER数据库相关性模块,使用Spearman的rho值评估统计差异并绘制散点图,同时提供了以肿瘤纯度或年龄为条件的部分相关选项。结果显示,MGP与大多数免疫相关基因的相关性显著,经过肿瘤纯度或年龄调整后差异仍有统计学意义(P < 0.05,图 5,表 2),图 5仅展示MGP与CD8A和CD8B基因的相关性。
|
| A: Correlation analysis of MGP with CD8A and CD8B;B: Correlation analysis of MGP with CD8A and CD8B adjusted by tumor purity; C: Correlation analysis of MGP with CD8A and CD8B adjusted by age. r: Correlation; r ': Partial correlation. 图 5 MGP与CD8基因表达的相关性分析 Fig 5 Correlation analysis of MGP and CD8 gene expression |
| Description(Gene markers) | None | Purity | Age | |||||||||
| Correlation | P | Correlation | P | Partial correlation | Partial P | Correlation | P | Partial correlation | Partial P | |||
| CD8+ T cell | ||||||||||||
| CD8A | 0.269 | 2.22e-06 | -0.546 | 7.63e-21 | 0.182 | 3.88e-03 | 0.024 | 6.76e-01 | 0.271 | 2.48e-06 | ||
| CD8B | 0.288 | 3.71e-07 | -0.441 | 2.53e-13 | 0.228 | 2.92e-04 | 0.042 | 4.71e-01 | 0.290 | 4.10e-07 | ||
| T cell(general) | ||||||||||||
| CD3D | 0.327 | 5.26e-09 | -0.573 | 3.02e-23 | 0.226 | 3.16e-04 | -0.005 | 9.37e-01 | 0.333 | 4.84e-09 | ||
| CD3E | 0.287 | 4.28e-07 | -0.612 | 4.35e-27 | 0.161 | 1.11e-02 | -0.027 | 6.41e-01 | 0.292 | 3.30e-07 | ||
| CD2 | 0.274 | 1.26e-06 | -0.604 | 3.20e-26 | 0.147 | 2.02e-02 | -0.009 | 8.77e-01 | 0.278 | 1.26e-06 | ||
| B cell | ||||||||||||
| CD19 | 0.042 | 4.63e-01 | -0.078 | 2.20e-01 | 0.014 | 8.24e-01 | 0.016 | 7.89e-01 | 0.050 | 3.96e-01 | ||
| CD79A | 0.310 | 3.51e-08 | -0.476 | 1.45e-45 | 0.244 | 1.03e-04 | 0.172 | 3.11e-03 | 0.315 | 3.55e-08 | ||
| Monocyte | ||||||||||||
| CD86 | 0.249 | 1.25e-05 | -0.615 | 2.11e-27 | 0.107 | 9.27e-02 | -0.036 | 5.43e-01 | 0.255 | 9.61e-06 | ||
| CD115(CSF1R) | 0.254 | 7.98e-06 | -0.602 | 5.06e-26 | 0.090 | 1.57e-01 | -0.057 | 3.32e-01 | 0.260 | 6.30e-06 | ||
| TAM | ||||||||||||
| CCL2 | 0.263 | 3.80e-06 | -0.506 | 1.14e-17 | 0.144 | 2.28e-02 | -0.027 | 6.39e-01 | 0.257 | 8.21e-06 | ||
| CD68 | 0.257 | 6.20e-06 | -0.600 | 8.44e-26 | 0.101 | 1.12e-01 | -0.021 | 7.17e-01 | 0.264 | 4.58e-06 | ||
| IL10 | 0.286 | 4.58e-07 | -0.442 | 2.35e-13 | 0.165 | 9.13e-03 | 0.044 | 4.47e-01 | 0.276 | 1.54e-06 | ||
| M1 macrophage | ||||||||||||
| INOS(NOS2) | 0.063 | 2.75e-01 | -0.150 | 1.77e-02 | 0.001 | 9.91e-01 | -0.076 | 1.90e-01 | 0.053 | 3.65e-01 | ||
| IRF5 | 0.066 | 2.53e-01 | -0.287 | 3.88e-06 | -0.004 | 9.51e-01 | -0.089 | 1.29e-01 | 0.079 | 1.77e-01 | ||
| COX2(PTGS2) | 0.128 | 2.57e-02 | -0.333 | 6.61e-08 | 0.112 | 7.79e-02 | 0.084 | 1.48e-01 | 0.125 | 3.23e-02 | ||
| M2 macrophage | ||||||||||||
| CD163 | 0.240 | 2.59e-05 | -0.57 | 6.25e-23 | 0.076 | 2.34e-01 | 0.036 | 5.33e-01 | 0.239 | 3.36e-05 | ||
| VSIG4 | 0.317 | 2.06e-08 | -0.582 | 4.91e-24 | 0.174 | 5.77e-03 | 0.051 | 3.81e-01 | 0.322 | 1.63e-08 | ||
| MS4A4A | 0.344 | 1.02e-09 | -0.603 | 3.45e-26 | 0.205 | 1.15e-03 | 0.038 | 5.14e-01 | 0.347 | 1.01e-09 | ||
| Neutrophils | ||||||||||||
| CD66b(CEACAM8) | 0.055 | 3.37e-01 | 0.030 | 6.42e-01 | 0.090 | 1.55e-01 | 0.039 | 5.10e-01 | 0.05 | 3.93e-01 | ||
| CD11b(ITGAM) | 0.192 | 7.79e-04 | -0.585 | 2.40e-24 | 0.022 | 7.30e-01 | -0.009 | 8.76e-01 | 0.199 | 6.09e-04 | ||
| CCR7 | 0.316 | 2.23e-08 | -0.543 | 1.32e-20 | 0.190 | 2.64e-03 | -0.075 | 1.96e-01 | 0.323 | 1.51e-08 | ||
| Natural killer cell | ||||||||||||
| KIR2DL1 | 0.076 | 1.84e-01 | -0.244 | 9.85e-05 | 0.011 | 8.64e-01 | 0.019 | 7.52e-01 | 0.088 | 1.31e-01 | ||
| KIR2DL3 | 0.024 | 6.84e-01 | -0.180 | 4.39e-03 | -0.027 | 6.76e-01 | -0.180 | 1.89e-03 | 0.033 | 5.79e-01 | ||
| KIR2DL4 | 0.044 | 4.50e-01 | -0.340 | 3.48-08 | -0.060 | 3.44e-01 | -0.114 | 5.00e-02 | 0.049 | 4.05e-01 | ||
| KIR3DL1 | 0.075 | 1.93e-01 | -0.328 | 1.10e-07 | -0.020 | 7.59e-01 | 0.029 | 6.19e-01 | 0.077 | 1.86e-01 | ||
| KIR3DL2 | 0.089 | 1.23e-01 | -0.270 | 1.49e-05 | -0.001 | 9.88e-01 | -0.066 | 2.57e-01 | 0.094 | 1.08e-01 | ||
| KIR3DL3 | -0.041 | 4.75e-01 | -0.111 | 7.91e-02 | -0.082 | 2.00e-01 | -0.101 | 8.40e-02 | -0.044 | 4.49e-01 | ||
| KIR2DS4 | -0.007 | 9.07e-01 | -0.204 | 1.18e-03 | -0.090 | 1.58e-01 | -0.106 | 6.90e-02 | 0.003 | 9.58e-01 | ||
| Dendritic cell | ||||||||||||
| HLA-DPB1 | 0.288 | 3.75e-07 | -0.476 | 1.54e-15 | 0.179 | 4.60e-03 | -0.129 | 2.66e-02 | 0.293 | 3.05e-07 | ||
| HLA-DQB1 | 0.174 | 2.34e-03 | -0.372 | 1.33e-09 | 0.074 | 2.42-01 | -0.065 | 2.64e-01 | 0.183 | 1.64e-03 | ||
| HLA-DRA | 0.254 | 8.29e-06 | -0.407 | 2.12e-11 | 0.157 | 1.29e-02 | -0.111 | 5.65e-02 | 0.256 | 9.08e-06 | ||
| HLA-DPA1 | 0.244 | 1.79e-05 | -0.458 | 2.14e-14 | 0.141 | 2.64e-02 | -0.125 | 3.14e-02 | 0.247 | 1.80e-05 | ||
| BDCA-1(CD1C) | 0.327 | 5.65e-09 | -0.442 | 2.34e-13 | 0.222 | 4.12e-04 | -0.046 | 4.32e-01 | 0.330 | 6.65e-09 | ||
| BDCA-4(NRP1) | 0.219 | 1.27e-04 | -0.473 | 2.28e-15 | 0.042 | 5.09e-01 | -0.020 | 7.34e-01 | 0.222 | 1.24e-04 | ||
| CD11c(ITGAX) | 0.137 | 1.71e-02 | -0.579 | 9.12e-24 | -0.028 | 6.58e-01 | -0.048 | 4.12e-01 | 0.143 | 1.39e-02 | ||
| Th1 | ||||||||||||
| T-bet(TBX21) | 0.218 | 1.31e-04 | -0.551 | 3.04e-21 | 0.088 | 1.68e-01 | -0.048 | 4.15e-01 | 0.222 | 1.27e-04 | ||
| STAT4 | 0.192 | 7.75e-04 | -0.549 | 4.06e-21 | 0.059 | 3.54e-01 | -0.039 | 5.08e-01 | 0.196 | 7.46e-04 | ||
| STAT1 | -0.101 | 7.96e-02 | -0.134 | 3.40e-02 | -0.121 | 5.74e-02 | -0.032 | 5.83e-01 | -0.101 | 8.37e-02 | ||
| IFN-γ(IFNG) | 0.163 | 4.40e-03 | -0.442 | 2.37e-13 | 0.070 | 2.71e-01 | -0.004 | 9.40e-01 | 0.162 | 5.29e-03 | ||
| TNF-α(TNF) | 0.031 | 5.87e-01 | -0.205 | 1.12e-03 | -0.052 | 4.17e-01 | 0.014 | 8.11e-01 | 0.024 | 6.81e-01 | ||
| Th2 | ||||||||||||
| GATA3 | 0.196 | 6.09e-04 | -0.477 | 1.32e-15 | 0.097 | 1.25e-01 | 0.133 | 5.16e-02 | 0.207 | 3.53e-04 | ||
| STAT6 | -0.074 | 2.00e-01 | -0.073 | 2.50e-01 | -0.084 | 1.84e-01 | -0.046 | 4.36e-01 | -0.060 | 3.03e-01 | ||
| STAT5A | 0.093 | 1.06e-01 | -0.193 | 2.17e-03 | 0.058 | 3.59e-01 | -0.043 | 4.64e-01 | 0.084 | 1.51e-01 | ||
| IL13 | 0.021 | 7.22e-01 | -0.090 | 1.54e-01 | 0.015 | 8.13e-01 | 0.030 | 6.09e-01 | 0.004 | 9.47e-01 | ||
| Tfh | ||||||||||||
| BCL6 | -0.010 | 8.69e-01 | 0.076 | 2.31e-01 | 0.046 | 4.67e-01 | -0.090 | 1.23e-01 | -0.009 | 8.76e-01 | ||
| IL21 | 0.050 | 3.85e-01 | -0.079 | 2.15e-01 | 0.022 | 7.32e-01 | 0.052 | 3.73e-01 | 0.052 | 3.78e-01 | ||
| Th17 | ||||||||||||
| STAT3 | 0.126 | 2.85e-01 | -0.259 | 3.37e-05 | 0.040 | 5.27e-01 | 0.032 | 5.85e-01 | 0.114 | 5.05e-02 | ||
| IL17A | 0.022 | 6.97e-01 | -0.141 | 2.56e-02 | -0.051 | 4.25e-01 | -0.017 | 7.65e-01 | 0.016 | 7.89e-01 | ||
| Treg | ||||||||||||
| FOXP3 | 0.106 | 6.44e-02 | -0.505 | 1.39e-17 | -0.060 | 3.44e-01 | -0.004 | 9.51e-01 | 0.108 | 6.38e-02 | ||
| CCR8 | 0.018 | 7.61e-01 | -0.319 | 2.64e-07 | -0.086 | 1.75e-01 | 0.054 | 3.55e-01 | 0.029 | 6.22e-01 | ||
| STAT5B | 0.070 | 2.26e-01 | -0.115 | 6.98e-02 | 0.053 | 4.09e-01 | 0.041 | 4.78e-01 | 0.051 | 3.86e-01 | ||
| TGFβ(TGFβ1) | 0.312 | 3.55e-08 | -0.669 | 8.29e-34 | 0.172 | 6.45e-03 | 0.038 | 5.19e-01 | 0.325 | 1.13e-08 | ||
| T cell exhaustion | ||||||||||||
| PD-1(PDCD1) | 0.279 | 8.16e-07 | -0.497 | 5.21e-17 | 0.183 | 3.68e-03 | -0.008 | 8.91e-01 | 0.272 | 2.25e-06 | ||
| CTLA4 | 0.203 | 3.82e-04 | -0.516 | 2.16e-08 | 0.065 | 3.10e-01 | 0.022 | 7.02e-01 | 0.207 | 3.50e-04 | ||
| LAG3 | 0.141 | 1.39e-02 | -0.355 | 8.08e-09 | 0.070 | 2.69e-01 | -0.012 | 8.38e-01 | 0.142 | 1.47e-02 | ||
| TIM-3(HAVCR2) | 0.249 | 1.28e-05 | -0.631 | 3.28e-29 | 0.095 | 1.35e-01 | -0.032 | 5.78e-01 | 0.256 | 9.11e-06 | ||
| GZMB | 0.229 | 5.64e-05 | -0.436 | 4.91e-13 | 0.085 | 1.80e-01 | -0.063 | 2.84e-01 | 0.227 | 8.69e-05 | ||
卵巢癌分期对患者5年生存率有重要影响。研究表明,MGP在病理性血管生成和肿瘤发生中发挥重要作用。在胶质母细胞瘤、宫颈癌和皮肤癌中均发现MGP表达水平升高,且与肿瘤进展呈正相关[8-9]。与正常组织中相比,MGP在多种实体瘤中表达下调。但是,我们在生存分析中发现MGP高表达组的卵巢癌患者预后更差,同时COX风险比例模型显示MGP为高风险因素。
通过GSEA方法分析MGP基因在卵巢癌组织中的富集情况,结果显示MGP富集于细胞黏附分子等信号通路。研究表明,细胞黏附分子PCDH18的低表达与头颈鳞癌的整体生存较差相关,而MPG通过调节这一信号通路,导致患者预后不良[10]。MGP同时富集于细胞因子受体信号通路、血管平滑肌收缩信号通路、CCR5信号通路、NK7信号通路等,因此我们推测MGP可能通过这些信号通路调控卵巢癌的发生发展。
CD8可特异性标记细胞毒性T细胞。细胞毒性T细胞是一种在体内识别肿瘤抗原并分泌针对效应细胞的细胞因子的寡克隆T细胞群,并与免疫浸润有关,因此在肿瘤清除中起积极作用。PD-L1和CD8+ T细胞联合诱导凋亡,并与过表达PDK1的卵巢癌细胞共培养可导致PD-L1表达增加,而PDK1的缺失则产生相反的作用[11]。我们通过GSEA分析MGP信号转导途径,其中几条与免疫密切相关,包括biocarta_NKT_信号通路、biocarta_TH1TH2_信号通路和biocarta_IL17_信号通路等,这些通路在MGP高表达组中均有富集。通过分析mRNA的表达,肿瘤微环境中存在抗肿瘤和免疫抑制因子,卵巢癌组织中CD4+T细胞浸润水平对生存率有影响,Kaplan-Meier生存曲线有显著差异[12]。噬菌体在宿主对肿瘤的防御中起积极作用,免疫细胞浸润影响卵巢癌的发生发展,尤其是T淋巴细胞的浸润[13]。一些淋巴细胞促进肿瘤的发展,而另一些淋巴细胞则抑制肿瘤的发展。但两者之间的机制尚不清楚,还需要进一步研究。
使用消炎药可能降低卵巢癌的风险。阿司匹林等非甾体抗炎药可能通过限制巨噬细胞的分化,发挥M2型巨噬细胞的免疫抑制作用,从而降低卵巢癌患者的风险。我们的数据显示卵巢癌患者与巨噬细胞浸润水平显著相关,提示MGP可能与肿瘤相关巨噬细胞的极化有关。MGP影响肿瘤血管生成,MGP的表达水平与肿瘤血管形成有关。本研究显示MGP表达增加与CD4+ T细胞和CD8+ T细胞浸润呈正相关。同时卵巢癌中CD8+ T细胞、CD4+ T细胞浸润水平与中性粒细胞和MGP表达均显著相关。因此,我们推测MGP有激活CD4+T细胞和CD8+ T细胞的潜力。此外,MGP表达与T细胞和B细胞标记基因的相关性提示MGP与卵巢癌肿瘤的免疫调节有关。首先,T细胞基因标记(如CCR7和CD3E),辅助T细胞Th1标记(如STAT4、INF-γ和INF-α)和Th2标记(如GATA-3、STAT6、STAT5A和IL13)均与MGP表达显著相关。这些结果揭示了MGP在免疫调节中的作用。MGP有激活巨噬细胞、中性粒细胞和树突状细胞的潜力。MGP表达与KIR3DL1、KIR3DL2、CD68、CD115、IL-10、HLA-DPB1等巨噬细胞和树突状细胞基因标志物的表达呈正相关。这些相关性提示了MGP调控卵巢癌免疫细胞功能的潜在分子机制。
本文首次证实MGP在卵巢癌预后中具有重要作用,同时证实MGP基因高表达与患者预后差密切相关。MGP表达与卵巢癌免疫细胞浸润水平密切相关,并调控多种免疫相关信号通路。在未来的研究中,需要进一步研究MGP在卵巢癌细胞中的功能,并对其分子机制进行深入探讨。
作者贡献声明 崔洪全 数据分析,论文撰写。张潇艺,陈彬 数据下载和整理。冯其贞 论文审核和修订。
利益冲突声明 所有作者均声明不存在利益冲突。
| [1] |
SUN H, CAO D, MA X, et al. Identification of a prognostic signature associated with DNA repair genes in ovarian cancer[J]. Front Genet, 2019, 10: 839.
[DOI]
|
| [2] |
ZHANG D, BAI B, XI Y, et al. Is aspirin use associated with a decreased risk of ovarian cancer? A systematic review and meta-analysis of observational studies with dose-response analysis[J]. Gynecol Oncol, 2016, 142(2): 368-377.
[DOI]
|
| [3] |
AHMED FY, WILTSHAW E, A'HERN RP, et al. Natural history and prognosis of untreated stage I epithelial ovarian carcinoma[J]. J Clin Oncol, 1996, 14(11): 2968-2975.
[DOI]
|
| [4] |
ZHANG G, LIU C, BAI H, et al. Combinatorial therapy of immune checkpoint and cancer pathways provides a novel perspective on ovarian cancer treatment[J]. Oncol Lett, 2019, 17(3): 2583-2591.
[URI]
|
| [5] |
POSTOW MA, CALLAHAN MK, WOLCHOK JD. Immune checkpoint blockade in cancer therapy[J]. J Clin Oncol, 2015, 33(17): 1974-1982.
[DOI]
|
| [6] |
CRANENBURG EC, KOOS R, SCHURGERS LJ, et al. Characterisation and potential diagnostic value of circulating matrix Gla protein (MGP) species[J]. Thromb Haemost, 2010, 104(4): 811-822.
[URI]
|
| [7] |
CHEN Y, MILLER C, MOSHER R, et al. Identification of cervical cancer markers by cDNA and tissue microarrays[J]. Cancer Res, 2003, 63(8): 1927-1935.
[URI]
|
| [8] |
HWANG WT, ADAMS SF, TAHIROVIC E, et al. Prognostic significance of tumor-infiltrating T cells in ovarian cancer: a meta-analysis[J]. Gynecol Oncol, 2012, 124(2): 192-198.
[DOI]
|
| [9] |
WANG JJ, SIU MK, JIANG YX, et al. Aberrant upregulation of PDK1 in ovarian cancer cells impairs CD8+ T cell function and survival through elevation of PD-L1[J]. Oncoimmunology, 2019, 8(11): e1659092.
[DOI]
|
| [10] |
MLYNSKA A, SALCIUNIENE G, ZILIONYTE K, et al. Chemokine profiling in serum from patients with ovarian cancer reveals candidate biomarkers for recurrence and immune infiltration[J]. Oncol Rep, 2019, 41(2): 1238-1252.
[URI]
|
| [11] |
BAANDRUP L, FABER MT, CHRISTENSEN J, et al. Nonsteroidal anti-inflammatory drugs and risk of ovarian cancer: systematic review and meta-analysis of observational studies[J]. Acta Obstet Gynecol Scand, 2013, 92(3): 245-255.
[DOI]
|
| [12] |
PERES LC, CAMACHO F, ABBOTT SE, et al. Analgesic medication use and risk of epithelial ovarian cancer in African American women[J]. Br J Cancer, 2016, 114(7): 819-825.
[DOI]
|
| [13] |
BARNARD ME, HECHT JL, RICE MS, et al. Anti-inflammatory drug use and ovarian cancer risk by COX1/COX2 expression and infiltration of tumor-associated macrophages[J]. Cancer Epidemiol Biomarkers Prev, 2018, 27(12): 1509-1517.
[DOI]
|
 2022, Vol. 49
2022, Vol. 49


