皮肤表面定植的细菌、真菌和病毒组成了皮肤共生微生物组,又称皮肤菌群[1]。皮肤菌群和宿主相互作用,共同抵御外来病原体、维持皮肤屏障、调节皮肤免疫[2-3]。以往皮肤菌群研究主要采用高通量测序的方法,揭示皮肤菌群的组成、分布和代谢功能等特征,探索菌群与疾病的相关性,但存在测序深度不够[4]、菌株水平信息缺乏、菌群功能研究难以深入等问题[5]。为弥补高通量测序的局限性,同时得益于微生物培养技术的快速发展,培养组学技术逐渐被用于探索人体共生菌群的组成和功能。培养组学已经成功培养了许多此前未被分离鉴定的菌种,揭示了宏基因组学研究所发现的未知菌群的详细分类和具体功能[6],在皮肤病研究领域显示出良好前景。本文就菌群研究方法的沿革、测序技术的局限性、培养组学的技术路线和技术进展、生理状态下人皮肤菌群培养组学研究、培养组学在皮肤病研究中的应用等方面进行综述。
菌群研究方法的沿革及培养组学的优势 微生物培养的起源可以追溯到19世纪中叶,传统微生物研究通常使用少数几种经验性的固体、液体培养基,直接观察不同细菌在特定培养基中的生理行为和表型特征[7],并结合染色试验[8],作为微生物种属鉴定的金标准。20世纪70年代初,细菌培养开始被用于肠道菌群组成和功能的研究[9],厌氧培养技术的进步推动了肠道菌群研究的进展[10]。尽管早期的微生物培养发现了许多重要的人体共生菌种,但大多数共生细菌仍然未能被成功培养。
进入21世纪,高通量测序技术快速拓展了人们对人体共生菌群的认识,超过80%的以往无法被培养的细菌种类通过测序被发现[11],测序技术迅速取代了传统培养方法,成为了共生菌群研究的主流方法。但测序技术在实现高灵敏度的同时,存在一些缺陷和偏倚。例如环境微生物的污染、宿主测序信息的干扰、取样方法的局限、DNA提取过程中的试剂污染等,降低了测序结果的准确性和可信度[12-13]。
由于测序方法的局限性,以及细菌培养方法的重大革新,培养组学重新受到了微生物学研究的重视。培养组学是指基于多样化的培养条件,分离培养得到尽可能多的菌株,并结合基质辅助激光解吸电离飞行时间质谱(matrix-assisted laser desorption/ionization time-of-flight mass spectrometry,MALDI-TOF MS)和16S rRNA测序技术,鉴定细菌种类并深入研究其功能的研究方法[14]。MALDI-TOF MS技术于2009年被首次报道用于培养组学属和种水平的精确鉴定[15],这一技术与传统菌种鉴定技术相比更快速、经济而准确,极大推动了培养组学的发展[16]。且与传统微生物培养相比,培养组学的培养条件更全面,可以得到更多的可培养菌,包括在以往研究中被认为“不可被培养”的物种[17]。培养组学可以确定宏基因组学发现的不能归入已知微生物物种基因序列的准确种属[18-19]。基于测序的方法通常只能进行相关性分析,无法确定微生物分类单元与功能之间的因果关系,培养组学还可以揭示单个菌种和菌株的详细信息,深入解释测序的结果[20],与宏基因组学研究形成了良好的互补作用。
2012年,Lagier等[21]首次发表了基于培养组学的肠道菌群研究,使用212种不同的培养条件,从粪便样本中分离出了174种此前未被报道的肠道细菌,其中包括31个新物种。截至2018年,培养组学显著增加了培养得到的与人类相关的细菌物种数量,可培养的人类相关细菌数量从2012年首个培养组学研究前的2172个物种上升到了2671个[22]。运用多样化的细菌培养条件,联合培养组和宏基因组等多组学分析的方法,可以更好地揭示人体共生菌群的全貌,探究菌株水平的改变对疾病的影响,为研究宿主-菌群相互作用提供了更广阔的视角[23]。
培养组学的技术路线及技术进展 培养组学的基本策略是用尽可能全面的培养条件,培养得到尽可能多的菌种[24]。培养组学的第一步是收集样本,并根据研究需要设计尽可能全面的培养条件,分别培养所得样本中的细菌。为了能在复杂的共生菌群中分离纯化特定的菌株,需要设置不同温度、pH及气体环境,添加选择性抑制剂,并使用含有特殊碳源、微量元素、生长因子的专用培养基在低浓度下促进苛养菌的生长[25-26]。此外,还需要厌氧罐、GasPak系统和厌氧室等系统用于厌氧菌的分离培养。在不同培养条件的固体培养基中获得单菌落后,对单菌落进行传代分离培养,再进行后续菌种鉴定。
基于培养组学的细菌物种鉴定通常使用MALDI-TOF MS和16S rRNA测序两种方法。16S rRNA测序成本高、耗时长,限制了其在高通量培养研究中的应用。MALDI-TOF MS鉴定快速、经济、准确,但其依赖于不断更新的细菌蛋白质数据库,受限于已被分离鉴定的细菌种类,部分无法被MALDI-TOF MS鉴定的细菌仍然需要通过16S rRNA测序进行鉴定。如果16S rRNA测序结果与最接近的已知菌株的相似度小于98.65%,则该菌株可能是一个新物种,这一新物种的发现可以被全基因组测序证实[27]。此外,全基因组测序还可以进一步得到菌株水平的基因组成和功能特征。通过对菌株全基因组序列的功能注释,以及耐药基因、毒力因子、代谢产物特征等关键功能基因的分析,可以预测特定菌株的功能特点,并据此开展后续的功能验证。此外,培养组学可以与宏基因组、代谢组、蛋白组等多组学研究相结合,深化对培养得到细菌的功能认识。皮肤菌群培养组学的技术路线如图 1所示。
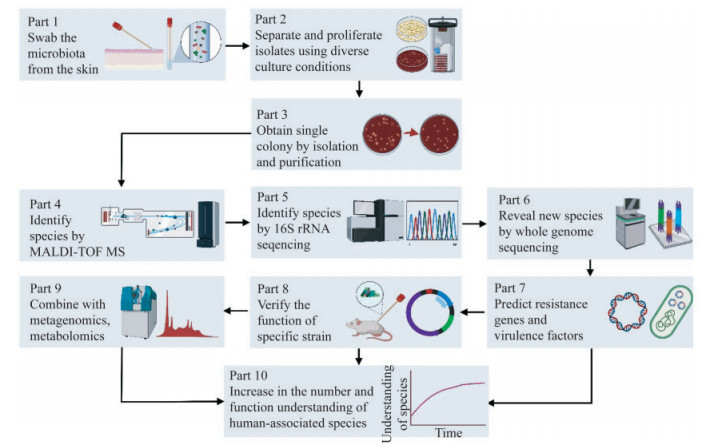
|
| Part 1: The first step of skin microbiota culturomics is to swab the microbiota from skin; Part 2: Separate and proliferate isolates using diverse culture conditions to culture as many bacteria as possible, including low concentrations of fastidious microorganisms; Part 3: Then obtain a single colony by isolation and purification; Part 4: Identifying species by MALDI-TOF mass spectrometry is an important feature of culturomics, which relies on a constantly updated database of bacterial proteins; Part 5: If identification fails, the isolate is identified by 16S rRNA sequencing; Part 6: When there is < 98.65% similarity to the closest known strain, the whole-genome sequencing could confirm the new species; Part 7: The whole-genome sequencing analysis can predict resistance genes, virulence factors, and other genetic traits of the species; Part 8: The function of specific strain could be verified by mouse and cell experiments; Part 9: Culturomics could be combined with metagenomics, metabolomics, and other multi-omics studies; Part 10: Culturomics could increase the number and function understanding of human-associated species. 图 1 皮肤菌群培养组学的技术路线 Fig 1 The technical route of skin microbiota culturomics |
目前培养组学采用的细菌培养方法存在养出率低、培养工作量大等限制[28],一些新技术和新方法的出现正在解决这些问题。Whelan等[18]采用平板覆盖算法(plate coverage algorithm,PLCA),对肺囊性纤维化痰标本进行富集培养。与直接测序分析相比,富集培养后进行的宏基因组学分析可以实现更丰富的分类学多样性、更优质的宏基因组组装、更长的重叠群以及更全面的功能注释。Cross等[29]采用了一种“反向基因组学”方法,使用抗体来预测膜蛋白,并从一个特定的分类单元中对微生物进行分类和培养。该方法已成功从环境样本、口腔菌群样本、唾液样本中定向富集和培养得到了常规方法未能培养的菌种。Fenn等[30]将粪便样本中的缓慢生长细菌与快速生长细菌进行共培养,发现大肠埃希菌(Escherichia coli)的存在允许粪杆菌属(Faecalibacterium)、嗜胆菌属(Bilophila)、拟杆菌属(Bacteroides)、萨特氏菌属(Suterella)、戈登氏杆菌属(Gordonibacter)的几种细菌通过甲基萘醌(Menaquinone)生物合成途径生长。此外,使用半透膜分离的双层软琼脂模拟共生细菌间的体外交流,也可以分离以前未被培养的物种[31]。
生理状态下人皮肤菌群培养组学研究 既往生理状态下皮肤菌群多样性研究主要基于高通量测序分析[32],近年来皮肤菌群培养组学丰富了测序所得的结果。Shami等[33]的研究分析了80名健康沙特人3个部位可培养需氧皮肤菌群的多样性,发现足部需氧菌多样性最高,手部其次,头皮需氧菌多样性最低。其中微球菌属(Micrococcus)和凝固酶阴性葡萄球菌(coagulase-negative Staphylococcus,CNS)是最主要的皮肤可培养须氧菌。老年人样本中培养得到了鲍曼不动杆菌(Acinetobacter baumannii)、粪肠球菌(Enterococcus faecalis)和肺炎克雷伯菌(Klebsiella pneumoniae),而在年轻人样本中培养得到了铜绿假单胞菌(Pseudomonas aeruginosa),推测这些细菌可能来源于医院相关的环境微生物。皮肤菌群的培养须排除环境微生物的干扰,但有时环境微生物和共生菌群并不能严格区分,环境微生物在病理情况下可能干扰皮肤共生菌群的组成。
2020年Microbiology首次报道了基于严格意义上培养组学设计的生理状态下皮肤菌群研究。Timm等[34]采集了17名22~45岁健康人的皮肤菌群样本,选取前额、前臂、肘窝等3个部位,设置了12种不同的分离培养条件,共得到842个菌株,分别属于38个不同的细菌属。研究发现了人类微生物组计划未报道的12株类芽孢杆菌属(Paenibacillus)、8株玫瑰单胞菌属(Roseomonas)和7株迪茨氏菌属(Dietzia)的细菌。研究还发现皮肤菌群碳源利用模式具有多样性,系统发育相近的种属碳源利用模式具有相似性,但同属的细菌碳源利用模式不尽相同。这一发现有助于解释健康人皮肤菌群多样性,也说明皮肤菌群研究中关注物种和菌株水平差异的必要性。
培养组学在鉴定皮肤菌群的多样性和个体特异性、部位特异性中具有独特优势。利用培养组学可以发现低丰度的物种以及鉴定与16S rRNA测序不同的菌种,也可以作为高通量测序的重要互补实验技术,完善对人体皮肤菌群组成的认识。
培养组学在皮肤疾病研究中的应用 随着高通量测序技术的发展,人们逐渐认识了整体皮肤菌群多样性和单个皮肤菌株改变对皮肤疾病的影响[35]。近年来培养组学与测序技术互相补充,进一步推动了疾病状态下的皮肤菌群研究。
Langan等[36]同时应用培养组学和16S rRNA测序,揭示了银屑病患者治疗过程中皮肤菌群的变化:银屑病皮损处可培养需氧菌中厚壁菌门(Firmicutes)的丰度增加,放线菌门(Actinobacteria)的丰度降低;皮损处皮肤菌群多样性降低,而在银屑病系统治疗期间皮肤菌群多样性部分恢复。
皮肤菌群的改变参与了皮肤慢性创伤的恢复过程[37],但皮肤菌群在慢性创伤恢复中的具体改变以及个体特异性的菌群组成变化尚未明确[38]。Oates等[40]培养了26例皮肤慢性创伤患者的皮肤菌群,发现伤口部位的皮肤菌群多样性高于对照侧,而分离出已知病原体伤口和未感染伤口的皮肤菌群多样性没有显著差异[39]。一项纳入2 963例慢性创伤患者的16S rRNA测序分析表明,慢性创伤处皮肤菌群多样性显著升高,葡萄球菌属(Staphylococcus)和假单胞菌属(Pseudomonas)的比例升高,患者的人种特征和创伤类型不会影响慢性创伤皮肤的菌群组成。培养结果与测序方法所得的皮肤菌群多样性结果相符,培养组学可以揭示特定菌株在维持慢性创伤皮肤菌群多样性中的作用。
通常认为皮肤局部抗生素和其他抗皮肤致病菌治疗措施可能降低皮肤菌群多样性[41],干扰正常的皮肤共生菌群组成,并可能导致特应性皮炎等炎症性皮肤病的发生[42]。Burnham等[43]通过培养组学技术分析了7例ICU患者和8例局部应用去金黄色葡萄球菌(Staphylococcus aureus)定植治疗的健康居民的皮肤菌群。研究运用多种培养条件,共得到174株分属于158个不同种的皮肤细菌。健康人治疗后皮肤金黄色葡萄球菌的丰度降低,皮肤菌群多样性和丰度没有显著改变;而ICU患者在外用抗菌治疗后,皮肤菌群的多样性和丰度均降低。该研究表明局部抗菌疗法不会对健康人的皮肤菌群造成重大干扰,仍然可以保留其皮肤的“有益”共生菌;而重症患者的皮肤菌群更脆弱,需要谨慎使用抗生素,警惕细菌耐药性的产生以及皮肤菌群稳态的破坏。
结语 培养组学方法的不断改进与完善,为皮肤菌群培养组学在皮肤病研究中的应用提供了更广阔的空间。培养组学与高通量测序技术的结合将庞大的菌群测序数据与精确的物种分类和功能研究相结合,从而更好地了解皮肤菌群的组成和功能,并为后续通过调节皮肤菌群来预防和治疗皮肤疾病提供依据。在解决了培养、分离、纯化、鉴定细菌的问题之后,如何建立特定菌株与皮肤疾病的因果关系是目前皮肤菌群培养组学研究需要解决的重要问题。构建合适的动物模型进行体内实验[44],运用现有的体外实验技术证明候选病原体或保护性微生物在特定疾病中的作用[45],将是皮肤菌群培养组学研究方向。
作者贡献声明 郁天泽 文献收集和整理,综述构思和撰写,图表绘制(使用BioRender.com绘制)。裘卓琼 综述构思和修订。李巍 综述构思、修订和审校。
利益冲突声明 所有作者均声明不存在利益冲突。
| [1] |
LOOMIS KH, WU SK, ERNLUND A, et al. A mixed community of skin microbiome representatives influences cutaneous processes more than individual members[J]. Microbiome, 2021, 9(1): 22.
[DOI]
|
| [2] |
BAURECHT H, RUHLEMANN MC, RODRIGUEZ E, et al. Epidermal lipid composition, barrier integrity, and eczematous inflammation are associated with skin microbiome configuration[J]. J Allergy Clin Immunol, 2018, 141(5): 1668-1676.
[DOI]
|
| [3] |
OH J, BYRD AL, PARK M, et al. Temporal stability of the human skin microbiome[J]. Cell, 2016, 165(4): 854-866.
[DOI]
|
| [4] |
MCKINZIE PB, BISHOP ME. A streamlined and high-throughput error-corrected next-generation sequencing method for low variant allele frequency quantitation[J]. Toxicol Sci, 2020, 173(1): 77-85.
|
| [5] |
CALLAHAN BJ, GRINEVICH D, THAKUR S, et al. Ultra-accurate microbial amplicon sequencing with synthetic long reads[J]. Microbiome, 2021, 9(1): 130.
[DOI]
|
| [6] |
LAGIER JC, KHELAIFIA S, ALOU MT, et al. Culture of previously uncultured members of the human gut microbiota by culturomics[J]. Nat Microbiol, 2016, 1: 16203.
[DOI]
|
| [7] |
BASCOMB S, MANAFI M. Use of enzyme tests in characterization and identification of aerobic and facultatively anaerobic gram-positive cocci[J]. Clin Microbiol Rev, 1998, 11(2): 318-340.
[DOI]
|
| [8] |
CARLONE GM, VALADEZ MJ, PICKETT MJ. Methods for distinguishing gram-positive from gram-negative bacteria[J]. J Clin Microbiol, 1982, 16(6): 1157-1159.
[DOI]
|
| [9] |
MOORE WE, HOLDEMAN LV. Human fecal flora: the normal flora of 20 Japanese-Hawaiians[J]. Appl Microbiol, 1974, 27(5): 961-979.
[DOI]
|
| [10] |
GRACEY M, BURKE V, OSHIN A, et al. Bacteria, bile salts, and intestinal monosaccharide malabsorption[J]. Gut, 1971, 12(9): 683-692.
[DOI]
|
| [11] |
ALMEIDA A, MITCHELL AL, BOLAND M, et al. A new genomic blueprint of the human gut microbiota[J]. Nature, 2019, 568(7753): 499-504.
[DOI]
|
| [12] |
FEERO WG. Bioinformatics, sequencing accuracy, and the credibility of clinical genomics[J]. JAMA, 2020, 324(19): 1945-1947.
[DOI]
|
| [13] |
LOUCA S, DOEBELI M, PARFREY LW. Correcting for 16S rRNA gene copy numbers in microbiome surveys remains an unsolved problem[J]. Microbiome, 2018, 6(1): 41.
[DOI]
|
| [14] |
DIAKITE A, DUBOURG G, DIONE N, et al. Optimization and standardization of the culturomics technique for human microbiome exploration[J]. Sci Rep, 2020, 10(1): 9674.
[DOI]
|
| [15] |
SENG P, DRANCOURT M, GOURIET F, et al. Ongoing revolution in bacteriology: routine identification of bacteria by matrix-assisted laser desorption ionization time-of-flight mass spectrometry[J]. Clin Infect Dis, 2009, 49(4): 543-551.
[DOI]
|
| [16] |
GREGORY D, CHAUDET H, LAGIER JC, et al. How mass spectrometric approaches applied to bacterial identification have revolutionized the study of human gut microbiota[J]. Expert Rev Proteomics, 2018, 15(3): 217-229.
[DOI]
|
| [17] |
BROWNE HP, FORSTER SC, ANONYE BO, et al. Culturing of 'unculturable' human microbiota reveals novel taxa and extensive sporulation[J]. Nature, 2016, 533(7604): 543-546.
[DOI]
|
| [18] |
WHELAN FJ, WADDELL B, SYED SA, et al. Culture-enriched metagenomic sequencing enables in-depth profiling of the cystic fibrosis lung microbiota[J]. Nat Microbiol, 2020, 5(2): 379-390.
[DOI]
|
| [19] |
GOVIL T, PASTE M, SAMANTA D, et al. Metagenomics and culture dependent insights into the distribution of firmicutes across two different sample types located in the Black Hills Region of South Dakota, USA[J]. Microorganisms, 2021, 9(1): 113.
[DOI]
|
| [20] |
MARTINY AC. High proportions of bacteria are culturable across major biomes[J]. ISME J, 2019, 13(8): 2125-2128.
[DOI]
|
| [21] |
LAGIER JC, ARMOUGOM F, MILLION M, et al. Microbial culturomics: paradigm shift in the human gut microbiome study[J]. Clin Microbiol Infect, 2012, 18(12): 1185-1193.
[DOI]
|
| [22] |
LAGIER JC, DUBOURG G, MILLION M, et al. Culturing the human microbiota and culturomics[J]. Nat Rev Microbiol, 2018, 16: 540-550.
[DOI]
|
| [23] |
TIDJANI ALOU M, MILLION M, TRAORE SI, et al. Gut bacteria missing in severe acute malnutrition, can we identify potential probiotics by culturomics?[J]. Front Microbiol, 2017, 8: 899.
[DOI]
|
| [24] |
CARINI P. A "cultural" renaissance: genomics breathes new life into an old craft[J]. mSystems, 2019, 4(3): e00092-19.
|
| [25] |
LAGIER JC, EDOUARD S, PAGNIER I, et al. Current and past strategies for bacterial culture in clinical microbiology[J]. Clin Microbiol Rev, 2015, 28(1): 208-236.
[DOI]
|
| [26] |
ECALE F, HOUARI AEL, CRAPART S, et al. In vitro sensitivity of 30 anaerobic bacterial strains of the human intestinal core microbiota to antibiotics: culture and LC-MS/MS approaches[J]. Anaerobe, 2021, 67: 102314.
[DOI]
|
| [27] |
ABDALLAH RA, BEYE M, DIOP A, et al. The impact of culturomics on taxonomy in clinical microbiology[J]. Antonie Van Leeuwenhoek, 2017, 110(10): 1327-1337.
[DOI]
|
| [28] |
DIAKITE A, DUBOURG G, RAOULT D. Updating the repertoire of cultured bacteria from the human being[J]. Microb Pathog, 2021, 150: 104698.
[DOI]
|
| [29] |
CROSS KL, CAMPBELL JH, BALACHANDRAN M, et al. Targeted isolation and cultivation of uncultivated bacteria by reverse genomics[J]. Nat Biotechnol, 2019, 37(11): 1314-1321.
[DOI]
|
| [30] |
FENN K, STRANDWITZ P, STEWART EJ, et al. Quinones are growth factors for the human gut microbiota[J]. Microbiome, 2017, 5(1): 161.
[DOI]
|
| [31] |
TANAKA Y, BENNO Y. Application of a single-colony coculture technique to the isolation of hitherto unculturable gut bacteria[J]. Microbiol Immunol, 2015, 59(2): 63-70.
[DOI]
|
| [32] |
LI Z, XIA J, JIANG L, et al. Characterization of the human skin resistome and identification of two microbiota cutotypes[J]. Microbiome, 2021, 9(1): 47.
[DOI]
|
| [33] |
SHAMI A, AL-MIJALLI S, PONGCHAIKUL P, et al. The prevalence of the culturable human skin aerobic bacteria in Riyadh, Saudi Arabia[J]. BMC Microbiol, 2019, 19(1): 189.
[DOI]
|
| [34] |
TIMM CM, LOOMIS K, STONE W, et al. Isolation and characterization of diverse microbial representatives from the human skin microbiome[J]. Microbiome, 2020, 8(1): 58.
[DOI]
|
| [35] |
TOMIDA S, NGUYEN L, CHIU BH, et al. Pan-genome and comparative genome analyses of propionibacterium acnes reveal its genomic diversity in the healthy and diseased human skin microbiome[J]. mBio, 2013, 4(3): e00003-13.
|
| [36] |
LANGAN EA, KUNSTNER A, MIODOVNIK M, et al. Combined culture and metagenomic analyses reveal significant shifts in the composition of the cutaneous microbiome in psoriasis[J]. Br J Dermatol, 2019, 181(6): 1254-1264.
[DOI]
|
| [37] |
LIU SH, HUANG YC, CHEN LY, et al. The skin microbiome of wound scars and unaffected skin in patients with moderate to severe burns in the subacute phase[J]. Wound Repair Regen, 2018, 26(2): 182-191.
[DOI]
|
| [38] |
TIPTON CD, WOLCOTT RD, SANFORD NE, et al. Patient genetics is linked to chronic wound microbiome composition and healing[J]. PLoS Pathog, 2020, 16(6): e1008511.
[DOI]
|
| [39] |
OATES A, BOWLING FL, BOULTON AJ, et al. Molecular and culture-based assessment of the microbial diversity of diabetic chronic foot wounds and contralateral skin sites[J]. J Clin Microbiol, 2012, 50(7): 2263-2271.
[DOI]
|
| [40] |
WOLCOTT RD, HANSON JD, REES EJ, et al. Analysis of the chronic wound microbiota of 2, 963 patients by 16S rDNA pyrosequencing[J]. Wound Repair Regen, 2016, 24(1): 163-174.
[DOI]
|
| [41] |
LIMA KM, DAVIS RR, LIU SY, et al. Longitudinal profiling of the burn patient cutaneous and gastrointestinal microbiota: a pilot study[J]. Sci Rep, 2021, 11(1): 10667.
[DOI]
|
| [42] |
NAKATSUJI T, CHEN TH, NARALA S, et al. Antimicrobials from human skin commensal bacteria protect against Staphylococcus aureus and are deficient in atopic dermatitis[J]. Sci Transl Med, 2017, 9(378): eaah4680.
[DOI]
|
| [43] |
BURNHAM CA, HOGAN PG, WALLACE MA, et al. Topical decolonization does not eradicate the skin microbiota of community-dwelling or hospitalized adults[J]. Antimicrob Agents Chemother, 2016, 60(12): 7303-7312.
[DOI]
|
| [44] |
ZAGATO E, POZZI C, BERTOCCHI A, et al. Endogenous murine microbiota member Faecalibaculum rodentium and its human homologue protect from intestinal tumour growth[J]. Nat Microbiol, 2020, 5(3): 511-524.
[DOI]
|
| [45] |
MYLES IA, EARLAND NJ, ANDERSON ED, et al. First-in-human topical microbiome transplantation with Roseomonas mucosa for atopic dermatitis[J]. JCI Insight, 2018, 3(9): e120608.
[DOI]
|
 2022, Vol. 49
2022, Vol. 49


