2. 复旦大学中西医结合研究院 上海 200040
2. Institutes of Integrative Medicine, Fudan University, Shanghai 200040, China
哮喘是一种异质性疾病,转录组测序常用来研究哮喘的发病机制及药物干预哮喘的疗效。由于哮喘的异质性,传统基于血液或组织样本的转录组测序方法获得的是细胞群体的宏观结果,呈现的是同一基因在所有细胞中的平均表达水平,不能呈现同一基因在单个细胞之间存在的差异以及其在疾病发病机制中的作用,因此不能完全揭示异质性哮喘的发病机制及药物疗效。单细胞转录组测序(single-cell RNA sequencing,scRNA-seq)是在单细胞水平对全转录组进行扩增与测序的一项新技术,能够获得每个细胞的mRNA表达信息,发现不同的细胞亚群以及相关信息,揭示细胞间基因表达的异质性并鉴定稀有细胞类型。scRNA-seq能够绘制肺组织细胞图谱,进行细胞谱系追踪,多角度、多层次和直观认识哮喘的发病机制,为深入研究和理解哮喘发病机制提供新的技术手段。本文对scRNA-seq技术在哮喘研究中的应用做一概述,以期为哮喘这一异质性疾病发病机制的深入研究提供思路。
哮喘概述 哮喘影响各年龄段的健康,患病率逐年上升,尤其是儿童患病率。据WHO统计,目前全球约有3亿人罹患哮喘,哮喘在不同国家的患病率为1%~18%,每年因哮喘死亡的人数高达25万,对个人、家庭和社会造成沉重的负担。全球哮喘防治创议(Global Initiative for Asthma,GINA)指出,哮喘是一种异质性疾病,以慢性气道炎症为主要特征[1]。哮喘的发病机制尚未完全明确,基因和环境的相互作用,参与哮喘发病的病理进展。哮喘的病理过程由多种细胞和细胞组分共同参与,包括肥大细胞、嗜酸性粒细胞(eosinophils,EOS)、T淋巴细胞、B淋巴细胞、巨噬细胞、中性粒细胞(neutrophils,NEU)、NK细胞和上皮细胞等,这些细胞与炎症细胞和外部环境相互作用,导致患者出现慢性气道炎症、可逆性气流受限、气道高反应性和气道重塑。
哮喘具有不同的疾病过程。可根据患者的病史、临床表现、病理生理学特征、治疗反馈以及预后差异分为不同的哮喘表型[2-3]。T淋巴细胞在哮喘慢性炎症中具有重要作用,2型辅助性T细胞(type 2 helper T cells,Th2)免疫应答增强(2型免疫反应)被认为是一种重要的哮喘炎症表型[4]。当初次接触过敏原,上皮细胞监测到过敏原,释放促炎因子,促进肺部树突状细胞的募集和活化,树突状细胞摄取过敏原,将过敏原转运至回流淋巴结,诱导过敏原特异性Th2细胞活化。当再次接触相同过敏原时,Th2效应细胞被刺激活化,分泌IL-4、IL-5和IL-13等2型炎症因子,引起过敏性气道炎症、肺部EOS增多、杯状细胞增生等炎症变化。大量关于哮喘的发病机制以及药效学研究均以Th2细胞作为关注的重点。NEU增多及Th17免疫应答增强也被认为是一种重要的哮喘炎症表型,参与重症哮喘的病理进展[5-10]。此外,临床上根据诱导痰中炎症细胞比例将哮喘分为EOS型、NEU型、混合粒细胞型和粒细胞缺乏型炎症表型。近年来,一些新的T细胞亚型及其在哮喘中的作用逐渐被认识,包括调节性T细胞(regulatory T,Treg)、滤泡辅助T细胞(follicular T helper,Tfh)、Th3、Th17、Th22和Th9细胞等,由此可见哮喘表型的多样性和发病机制的复杂性。
scRNA-seq概述 scRNA-seq是以单个细胞为单位,通过将组织或体液样本中的细胞群分离成单个细胞,进行全转录组扩增和高通量测序,获得相应数据并进行信息分析的技术,也是目前应用最广泛的单细胞测序方法。scRNA-seq能够获得每个细胞同一基因的转录组表达数据,准确呈现基因表达量在不同细胞间的差异,进而在单细胞水平重新认识各种组织器官及其在疾病发病机制中的作用,尤其对异质性疾病的研究具有不可替代的优势。2009年,Tang等[11]首次将scRNA-seq用于小鼠胚叶细胞的研究,开创了第一个哺乳动物scRNA-seq方法。此后相继出现改良和优化的scRNA-seq技术[12-17]。2017年,商业化的10×Genomics技术问世[18],此后相继开发的Seq-Well技术[19]、Microwell-seq测序平台[20]、SPLit-seq技术[21]和Slide-Seq技术[22]使大规模单细胞测序相关研究得以开展。相较于传统转录组测序技术,scRNA-seq能够深入研究细胞类型、细胞功能、细胞亚群、细胞亚群的异质性和细胞谱系等[23],被广泛应用于生命科学和医学研究的多个领域。scRNA-seq的基本流程包括单细胞制备、单细胞文库构建与测序和数据分析(图 1)。
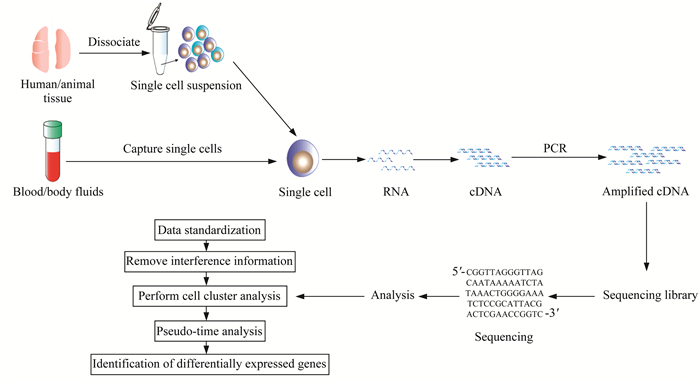
|
| 图 1 单细胞转录组测序流程图 Fig 1 Flow chart of scRNA-seq |
单细胞制备 单细胞制备是scRNA-seq的第一步,直接影响后续的建库、转录组测序和数据分析。常用的单细胞制备方法包括低通量单细胞制备方法(如有限稀释法、显微操作法、激光捕获显微切割分离法)和高通量单细胞分选方法(如流式细胞法和微流控技术)。有限稀释法通过对细胞悬液进行梯度稀释,至适宜浓度获得单细胞悬液。显微操作法需要将制备的单细胞悬液稀释至适当浓度,在光学显微镜下挑选目的细胞,并利用口吸管将细胞吸出,获得后续测序所需的单细胞。激光捕获显微切割分离法需要将组织样本固定至热塑膜涂片上,在倒置显微镜下使用高度聚焦的激光束切割待分选的目的细胞。流式细胞法分选目的细胞,首先对目的细胞进行荧光标记,利用流式细胞仪分选单个活细胞,该技术通量高、特异性强,是最常用的单细胞制备方法之一。微流控技术分选法利用含有与细胞大小一致的反应室的微流控芯片,进而获得单个活细胞,除了具有分选活细胞的功能外,该技术还可以进行后续单细胞基因组或转录测序,具有高通量和高效率的双重优势。
单细胞文库构建与测序 获得单细胞之后,需要对单个细胞进行转录组扩增,构建RNA文库以满足后续测序需求。全转录组扩增的方法主要有基于PCR的方法(多重扩增方法)和基于体外转录的全转录组方法(使用条形码的多重线性扩增)。根据合成第2条cDNA链方法的不同,PCR扩增法分为末端加尾法、模板转换法和随机引物法。体外转录扩增法以线性扩增法代替PCR指数扩增,避免了PCR指数扩增带来的实验误差。体外转录扩增法从单细胞逆转录反应开始,使用特殊的引物逆转录为cDNA,在T7RNA聚合酶的作用下以上述cDNA为模板,转录得到RNA并建立RNA文库[12]。
单细胞测序包括先扩增序列再测序的第二代高通量测序和直接测序的第三代高通量测序。第二代高通量测序具有测序成本较低和测序效率较高的特点,包括Illumina公司的Solexa聚合酶合成测序技术、ABI公司的寡聚物连接检测测序和Roche公司的焦磷酸测序技术。第三代高通量测序基于单分子测序技术,不依赖PCR扩增技术,能够避免PCR扩增产生的误差。
数据分析 scRNA-seq数据分析包括一系列操作步骤,由于结果包含海量信息,首先需要对数据进行标准化处理。标准化的原始数据处理流程和数据处理包可从公开数据库下载。在常规分析之前,需要移除干扰信息(如接头序列、连接序列和标签序列等),对剩余的序列信息进行分析。首先,通过基础流程Cell Ranger将原始数据拆分成FASTQ文件[4, 24],通过质控除去不符合要求的数据,再用标签复读、计算机对相应数据量化并生成基因表达矩阵。接着,利用Seurat软件进行细胞聚类分析[25],先后通过主成分分析(principal component analysis,PCA)进行线性降维[26]和t-分布随机邻域嵌入(t-distributed stochastic neighbor embedding,t-SNE)算法进行非线性降维[27],呈现可视化结果。然后,利用monocle软件进行伪时间分析。最后,寻找每个聚类中显著表达的基因进行高度差异表达基因的鉴定。scRNA-seq数据分析主要是对测序样本进行异质性分析,分析细胞内的基因变化、对细胞进行分群(通过已知的marker基因)、发现新的细胞类型以及分析不同细胞群体之间的相互作用。
scRNA-seq在哮喘研究中的应用 scRNA-seq是近年来迅速发展的生命科学前沿技术,能够在单细胞水平揭示细胞的基因表达状态,反映细胞间的异质性,为研究异质性疾病提供了新的研究思路和技术手段。通过分离肺组织单细胞,进行转录组测序分析,有助于准确理解细胞分化、信号转导等生物学过程及相关的基因调控网络,深入阐明哮喘的发病机制。scRNA-seq已应用于哮喘的研究中,并取得一定进展。已发表的相关研究主要对肺组织、气道、淋巴结细胞进行研究及分群,揭示不同细胞群体及群体之间的相互作用在哮喘发病中的作用。
肺部结构细胞和免疫细胞的相互作用对于维持肺稳态至关重要。Vieira Braga等[28]对正常人和哮喘患者肺组织的上气道、下气道和肺实质进行单细胞转录组学研究,选取通过鼻刷、支气管活检、肺切除和肺移植获得的正常受试者和哮喘患者的样本,对肺组织的结构细胞和定居在肺组织的炎症细胞及其相互作用进行研究。针对上皮细胞谱系,在上气道、下气道和肺实质中发现了至少10种上皮细胞,包括基底细胞、club细胞、纤毛细胞、杯状细胞Ⅰ型肺泡细胞、Ⅱ型肺泡细胞和ionocyte等;每种基底细胞、杯状细胞和纤毛上皮细胞又可以细分为2种表达不同marker基因的细胞状态。针对免疫细胞,在上气道、下气道和肺实质中发现了髓样细胞,包括巨噬细胞、NEU、树突状细胞和肥大细胞;同时发现了淋巴样细胞,包括T细胞、NK细胞和B细胞。针对基质细胞,在上气道、下气道和肺实质中发现了血管内皮细胞、成纤维细胞、肌成纤维细胞、平滑肌细胞和淋巴管内皮细胞。结构细胞和炎症细胞的转录组学表型和细胞比例均存在差异;同时鉴定出一种新的定居在肺组织的CD4+ T细胞亚群,该亚群同时具有循环记忆细胞和组织定居记忆细胞的特征,许多疾病相关基因具有高度细胞类型特异性表达模式。该研究在哮喘患者肺部发现了一种新的上皮细胞状态——黏液纤毛细胞。这些黏液纤毛细胞代表了一种纤毛细胞的过渡状态,其参与哮喘这类慢性病的黏液细胞增生。此外,还发现了其他哮喘相关的改变,包括杯状细胞、上皮内肥大细胞、气道壁病理性效应Th2细胞数量增加等。通过分析正常受试者和哮喘患者的细胞间通讯发现,伴随与Th2细胞相互作用明显增加,结构细胞通讯明显减少,该研究对上皮细胞的变化产生了新的见解,发现免疫细胞和结构细胞之间的通讯模式改变是哮喘性气道炎症的发病基础。
微生物低剂量暴露、呼吸道病毒感染及空气污染物都是哮喘发病的危险因素,但是这些危险因素和宿主2型免疫紊乱易感性之间的发病机制仍不明确。Radermecker等[29]利用scRNA-seq技术对来自于载体对照、低剂量及高剂量LPS暴露合并HDM致敏和激发诱导的哮喘模型小鼠肺组织的NEU进行深入研究,结果显示NEU共分为6群,其中载体对照的肺组织中,NEU主要为1个细胞群体(cluster 0);低剂量及高剂量LPS暴露合并HDM致敏和激发诱导的哮喘模型小鼠肺组织中,NEU分为5群(cluster 1~5)。cluster 1仅出现在低剂量LPS暴露合并HDM致敏和激发诱导的哮喘模型小鼠肺组织中;cluster 2/3/5出现在高剂量LPS暴露合并HDM致敏和激发诱导的哮喘模型小鼠肺组织中;cluster 4平均出现在低剂量及高剂量LPS暴露合并HDM致敏和激发诱导的哮喘模型小鼠肺组织中。分别对这6群NEU进行差异表达基因分析,明确了共同的、剂量非依赖的LPS诱导的基因表达特征,具体表现为:与NEU分群cluster 0相比,NEU分群cluster 1~5具有共同表达上调的转录本。对低剂量及高剂量LPS暴露诱导的肺组织特异性NEU分群(低剂量:cluster 1,高剂量:cluster 2/3/5)的基因表达特征进行鉴定,Cxcr4和Lamp-1转录本(其编码的蛋白质可以通过流式细胞术检测到)在低剂量LPS暴露特异性的NEU分群cluster 1中显著上调。高剂量LPS暴露特异性的NEU分群cluster 2/3/5中包含25个转录本,基因本体论分析(gene ontology analysis)显示这25个转录本富集在补体受体介导的信号通路及1型或2型干扰素反应等生物过程。低剂量LPS暴露特异性的NEU分群cluster 1中包含97个转录本,分别富集在内质网应激、活性氧合成、氧化应激和ERK1/2信号级联反应,这些通路均参与NET形成和分泌。通过scRNA-seq分析明确,在低剂量与高剂量LPS暴露诱导的炎症环境中,肺部NEU存在明显差异。
Th2细胞在哮喘发病中具有重要作用,但是对哮喘病理进展中Th2细胞的转录谱特征仍不清楚。Tibbitt等[30]对HDM诱导的哮喘模型中纵隔淋巴结、肺组织和气道中Th细胞进行scRNA-seq研究。HDM诱导的小鼠哮喘模型中,CD4+ T细胞扩增了120余倍。气道中出现大量Foxp3+Treg、Th1、Th2和Th17细胞亚群,而肺组织和淋巴结中较少出现Th相关细胞亚群。该研究利用SMART-Seq2平台对气道中的CD3+CD4+CD44+Th细胞进行scRNA-seq,通过两次独立实验,在每个细胞中检测到2 000~2 500个基因表达,在所有细胞中共检测到超过12 000个基因,其中1 971个基因明显差异表达。进一步分析显示,这些CD3+CD4+CD44+Th细胞共分为6个亚群,均表达Cd4、Cd3e、Cd3g、Cd3d和Cd44。Th细胞亚群cluster 1最像典型的Treg细胞,表达Foxp3、Ctla4、Il10、Folr4、Il2ra和Klrg1 mRNA;Th细胞亚群cluster 4具有Th2细胞表型,表达Il1rl1(ST2)、Gata3、Il13、Il5、Il17rb、Ltb4r1和Ccr8 mRNA。Pparg是Th2细胞介导免疫应答所必需的基因,在Th2细胞亚群中特异性表达。此外,在Th细胞亚群cluster 4中发现了一系列高表达基因(Igfbp7、Plac8、Gclc、Serpinb6a、Fgl2、Vdr、Hlf),此前并未报道这些基因与Th2免疫应答相关。Th细胞亚群cluster 5特征性表达Cxcr3、Ccl5和Ms4a4b mRNA,这群细胞同时富集Tbx21和Ifng mRNA,提示这群细胞具有Th1细胞特征。Th细胞亚群cluster 2高表达Tcf7、S1pr1、Tmem176a和Tmem176b mRNA,具有活化和记忆表型。Th细胞亚群cluster 3缺少明确的差异表达基因,可能是细胞的混合表型。Th细胞亚群cluster 6显著表达Ifit3、Isg15、Isg20、Mx1、Stat1和Stat2,说明这群细胞对Ⅰ型干扰素具有应答反应。但是Th细胞亚群cluster 6并未显著表达Ⅰ型干扰素受体Ifnar1和Ifnar2,说明不具有对这类细胞因子作出反应的倾向性。研究人员提出假设,Ⅰ型干扰素应答反应可能是由HDM提取物中的toll样受体配体介导的。进一步通过野生型、Tlr4-/-和Myd88-/-小鼠开展相关研究,结果显示,对Ⅰ型干扰素的应答反应并不局限于特定的Th细胞亚群,对Ⅰ型干扰素应答的幼稚CD4+ T细胞、Th1、Th2、Treg细胞的转录谱特征。显著富集到气道的Th2细胞特征性基因包括Cd200r1、Il6、Plac8和Igfbp7;Th2细胞内差异表达的基因显著富集到脂代谢相关通路,抑制关键通路明确了糖脂代谢通路在Th2细胞中的作用,进一步明确了细胞代谢对过敏性疾病中病理性Th2细胞分化的作用。
结语 自2009年scRNA-seq技术问世以来,已广泛用于各种疾病发病机制的研究,尤其是异质性疾病。目前基于scRNA-seq技术在哮喘方面的研究结果,揭示出肺组织不同的细胞群体及其相互作用在哮喘的发病机制中具有重要作用。目前针对肺组织的scRNA-seq还有待进一步提升。首先,肺组织单细胞悬液制备过程复杂,对肺组织样本中某些细胞群体的分选提出了挑战,部分细胞群体死亡率过高,造成数据失真或损失。构成肺组织的各类细胞对不同的消化酶的反应条件不同,为了尽可能获得肺组织的全部细胞种类,需要对肺组织的酶消化方法进行实验性探索。其次,现有scRNA-seq技术主要针对含有poly-A尾的mRNA,不能检测在哮喘发病中具有重要作用但无poly-A尾的microRNA和lncRNA等,这也是scRNA-seq技术需要完善的重要方面。最新研究显示,单细胞组学研究已由转录组学向多组学延伸,未来的单细胞多组学测序技术将更全面和准确揭示多种类型肺部细胞及其亚型在哮喘发病中的具体作用机制。
作者贡献声明 魏颖 论文构思、撰写和修改。吾尼且木·吐拉克,滕方舟 文献整理。汤蔚峰 文献整理,制图。董竞成 论文审定。
利益冲突声明 所有作者均声明不存在利益冲突。
| [1] |
GLOBAL INITIATIVE FOR ASTHMA. GINA Global Strategy for Asthma Management and Prevention[EB/OL]. (2020-04-06)[2020-09-15]. http://www.ginasthma.org.
|
| [2] |
CHUPP GL, KAUR R, MAINARDI A. New therapies for emerging endotypes of asthma[J]. Annu Rev Med, 2020, 71: 289-302.
[DOI]
|
| [3] |
FUHLBRIGGE AL, Castro M. Precision medicine in asthma-using phenotypes to understand endotypes that lead us to new therapeutic options[J]. J Allergy Clin Immunol Pract, 2020, 8(2): 496-497.
[DOI]
|
| [4] |
LLOYD CM, SNELGROVE RJ. Type 2 immunity: expanding our view[J]. Sci Immunol, 2018, 3(25): 1-11.
[URI]
|
| [5] |
EKSTEDT S, STENBERG H, TUFVESSON E, et al. The potential role of CD16(high) CD62L (dim) neutrophils in the allergic asthma[J]. Allergy, 2019, 74(11): 2265-2268.
[DOI]
|
| [6] |
KRISHNAMOORTHY N, DOUDA DN, BRUGGE-MANN TR, et al. Neutrophil cytoplasts induce TH17 differentiation and skew inflammation toward neutrophilia in severe asthma[J]. Sci Immunol, 2018, 3(26): 1-13.
[URI]
|
| [7] |
LACHOWICZ-SCROGGINS ME, DUNICAN EM, CHARBIT AR, et al. Extracellular DNA, neutrophil extracellular traps, and inflammasome activation in severe asthma[J]. Am J Respir Crit Care Med, 2019, 199(9): 1076-1085.
[DOI]
|
| [8] |
NG LG, OSTUNI R, HIDALGO A. Heterogeneity of neutrophils[J]. Nat Rev Immunol, 2019, 19(4): 255-265.
[DOI]
|
| [9] |
PAPAYANNOPOULOS V. Neutrophil extracellular traps in immunity and disease[J]. Nat Rev Immunol, 2018, 18(2): 134-147.
[DOI]
|
| [10] |
RADERMECKER C, LOUIS R, BUREAU F, et al. Role of neutrophils in allergic asthma[J]. Curr Opin Immunol, 2018, 54: 28-34.
[DOI]
|
| [11] |
TANG F, BARBACIORU C, WANG Y, et al. mRNA-Seq whole-transcriptome analysis of a single cell[J]. Nat Methods, 2009, 6(5): 377-382.
[DOI]
|
| [12] |
HASHIMSHONY T, WAGNER F, SHER N, et al. CEL-Seq: single-cell RNA-Seq by multiplexed linear amplification[J]. Cell Rep, 2012, 2(3): 666-673.
[DOI]
|
| [13] |
ISLAM S, KJALLQUIST U, MOLINER A, et al. Characterization of the single-cell transcriptional landscape by highly multiplex RNA-seq[J]. Genome Res, 2011, 21(7): 1160-1167.
[DOI]
|
| [14] |
JAITIN DA, KENIGSBERG E, KEREN-SHAUL H, et al. Massively parallel single-cell RNA-seq for marker-free decomposition of tissues into cell types[J]. Science, 2014, 343(6172): 776-779.
[DOI]
|
| [15] |
KLEIN AM, MAZUTIS L, AKARTUNA I, et al. Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells[J]. Cell, 2015, 161(5): 1187-1201.
[DOI]
|
| [16] |
PICELLI S, BJORKLUND AK, FARIDANI OR, et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells[J]. Nat Methods, 2013, 10(11): 1096-1098.
[DOI]
|
| [17] |
RAMSKOLD D, LUO S, WANG YC, et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells[J]. Nat Biotechnol, 2012, 30(8): 777-782.
[DOI]
|
| [18] |
ZHENG GX, TERRY JM, BELGRADER P, et al. Massively parallel digital transcriptional profiling of single cells[J]. Nat Commun, 2017, 8: 14049.
[DOI]
|
| [19] |
GIERAHN TM, WADSWORTH MH 2ND, HUGHES TK, et al. Seq-Well: portable, low-cost RNA sequencing of single cells at high throughput[J]. Nat Methods, 2017, 14(4): 395-398.
[DOI]
|
| [20] |
HAN X, WANG R, ZHOU Y, et al. Mapping the mouse cell atlas by microwell-Seq[J]. Cell, 2018, 172(5): 1091-1107.
[DOI]
|
| [21] |
ROSENBERG AB, ROCO CM, MUSCAT RA, et al. Single-cell profiling of the developing mouse brain and spinal cord with split-pool barcoding[J]. Science, 2018, 360(6385): 176-182.
[DOI]
|
| [22] |
RODRIQUES SG, STICKELS RR, GOEVA A, et al. Slide-seq: a scalable technology for measuring genome-wide expression at high spatial resolution[J]. Science, 2019, 363(6434): 1463-1467.
[DOI]
|
| [23] |
PAPALEXI E, SATIJA R. Single-cell RNA sequencing to explore immune cell heterogeneity[J]. Nat Rev Immunol, 2018, 18(1): 35-45.
[DOI]
|
| [24] |
WOLF FA, ANGERER P, THEIS FJ. SCANPY: large-scale single-cell gene expression data analysis[J]. Genome Biol, 2018, 19(1): 15.
[DOI]
|
| [25] |
SATIJA R, FARRELL JA, GENNERT D, et al. Spatial reconstruction of single-cell gene expression data[J]. Nat Biotechnol, 2015, 33(5): 495-502.
[DOI]
|
| [26] |
TREUTLEIN B, BROWNFIELD DG, WU AR, et al. Reconstructing lineage hierarchies of the distal lung epithelium using single-cell RNA-seq[J]. Nature, 2014, 509(7500): 371-375.
[DOI]
|
| [27] |
SAADATPOUR A, GUO G, ORKIN SH, et al. Characterizing heterogeneity in leukemic cells using single-cell gene expression analysis[J]. Genome Biol, 2014, 15(12): 525.
[DOI]
|
| [28] |
VIEIRA BRAGA FA, KAR G, BERG M, et al. A cellular census of human lungs identifies novel cell states in health and in asthma[J]. Nat Med, 2019, 25(7): 1153-1163.
[DOI]
|
| [29] |
RADERMECKER C, SABATEL C, VANWINGE C, et al. Locally instructed CXCR4(hi) neutrophils trigger environment-driven allergic asthma through the release of neutrophil extracellular traps[J]. Nat Immunol, 2019, 20(11): 1444-1455.
[DOI]
|
| [30] |
TIBBITT CA, STARK JM, MARTENS L, et al. Single-cell RNA sequencing of the T helper cell response to house dust mites defines a distinct gene expression signature in airway Th2 cells[J]. Immunity, 2019, 51(1): 169-184.
[DOI]
|
 2021, Vol. 48
2021, Vol. 48


