溶组织内阿米巴(Entamoeba histolytica,Eh)是一种主要寄生在人体肠道的厌氧原虫,可引起严重的阿米巴性结肠炎和肠外脓肿。阿米巴病的严重性在全球寄生原虫病中居第二位,致死率仅次于疟疾。据估计,全世界每年约有5~10万人死于阿米巴病,5 000万人出现Eh新感染。Eh感染人体的过程与一系列阿米巴毒力因子蛋白质相关。具有感染性的Eh包囊经口摄入人体,随后通过胃和小肠,在回肠末端或结肠转变为具有致病性的滋养体。滋养体通过细胞表面的半乳糖/乙酰氨基半乳糖可抑制性凝集素(galactose/N-acetyl galactosamine-inhibitable lectin,Gal/GalNAc lectin)等物质黏附于宿主肠上皮细胞,随后释放阿米巴穿孔素(amoebapore)和半胱氨酸蛋白酶(cysteine proteases,CP)等毒力因子,通过接触依赖性细胞溶解作用使与之接触的细胞发生坏死或凋亡,亦或通过胞啃作用直接损伤宿主细胞,从而形成烧瓶样的肠道溃疡[1-4]。
结构生物学(structural biology)是一门通过交叉融合生物、化学、物理、数学和计算机等多领域知识来研究生物大分子的结构及其动态变化,进而阐明生命现象的学科,这门学科的出现与发展为药物设计、疫苗开发和蛋白质分子性能改造等应用方向奠定了理论和技术基础[5-6]。结构生物学的起源可以追溯到20世纪50年代Waston和Crick解析出DNA双螺旋结构[7]。随着技术的不断发展,结构生物学在核酸和蛋白质结构研究中发挥越来越重要的作用。蛋白质结构坐标数据库(Protein Data Bank,PDB,http://www.rcsb.org/pdb)中收录已解析的蛋白质三维结构数目从1989年的300余个增至156 101个(截至2019年9月18日)。结构生物学技术也已运用到现代生物学和医学研究的各个领域,包括人体寄生虫学研究领域。在人体寄生原虫Eh中,已经解析出150余种虫体蛋白质的三维结构,为研究阿米巴代谢活动、致病机制以及研发药物和疫苗等提供了重要信息。
结构生物学技术发展概况 目前结构生物学的主要研究方法包括X射线晶体学(X-ray macromolecular crystallography)、核磁共振波谱学(nuclear magnetic resonance spectroscopy)和冷冻电子显微学(cryo-electron microscopy)。
X射线晶体学 1957年,英国科学家Kendrew等[8]首次将X射线晶体学方法运用于蛋白质三维结构的解析,即获得分辨率为6Å的抹香鲸肌红蛋白晶体结构。之后,Kendrew等[9]将抹香鲸肌红蛋白晶体结构的分辨率提高到2Å,并获得1962年诺贝尔化学奖。X射线晶体学的基本原理是利用X射线对高纯度蛋白质晶体进行衍射,通过对衍射数据的分析处理来获得近原子分辨率的蛋白质大分子三维结构。经过半个多世纪的理论发展和硬件设备升级,X射线晶体学已经成为结构生物学研究中最主要的研究手段之一[10-11]。
目前PDB数据库收录的蛋白质三维结构中有89.1%(139 132个,截至2019年9月18日)是通过X射线晶体学方法解析得到的。然而,X射线晶体学的应用仍存在一定的局限,即必须先获得可衍射的蛋白质晶体才能进行结构探测,而在实验室获得蛋白质结晶往往是一个非常耗时且具有挑战性的工作,特别是膜蛋白和超大分子量蛋白质复合物通常难以结晶,往往需要通过诱变和高通量筛选来获得构象稳定的蛋白质样品[5, 12-13]。因此,一些重要的蛋白质分子难以通过X射线晶体学方法获得三维结构信息,需要寻求其他的解决方法。
核磁共振波谱学 20世纪50年代,核磁共振技术与X射线衍射方法几乎同时开始应用于结构生物学研究领域。1957年,Saunders等[14]首次成功运用核磁共振波谱学方法解析蛋白质结构即核糖核酸酶的三维结构。多维光谱学、量子计算和高通量核磁共振技术的发展,为核磁共振波谱学提供了理论和技术支撑[15-18]。目前PDB数据库收录的蛋白质三维结构中有8.1%(12 657个,截至2019年9月18日)是通过核磁共振波谱学方法解析得到的。
核磁共振波谱学通常用于解析溶液中蛋白质的三维结构、动力学特征以及蛋白质间相互作用。与X射线晶体学相比,核磁共振波谱技术解析出的蛋白质结构更接近于生理状态。然而,该技术常用于解析不易结晶的小分子量蛋白质的结构,对于大分子量蛋白质复合体或难溶于水的蛋白质往往不易得到理想的结果。在21世纪之前,通过核磁共振技术解析结构的蛋白质分子量几乎都不超过25 000。随着技术发展,特别是脉冲序列技术的突破,通过核磁共振解析的最大蛋白质分子量突破1MDa,因此核磁共振波谱学依然具有很好的应用前景[19-20]。
冷冻电子显微学 冷冻电子显微学,又称冷冻电镜技术,是一项从20世纪70年代发展起来的新兴技术,现已成功运用于细胞生物学和结构生物学研究。2013年,由于直接电子相机的应用,冷冻电镜技术成功解析高分辨率膜蛋白三维结构,即分辨率为3.4 Å的哺乳动物细胞膜蛋白辣椒素受体的三维结构[21]。在此之后,越来越多的膜蛋白和分子量较大的蛋白质及其复合物通过冷冻电镜技术解析出三维结构。目前PDB数据库收录的蛋白质三维结构中有2.4%(3 755个,截至2019年9月18日)是通过冷冻电镜技术解析得到的。
冷冻电镜技术的基本原理是将细胞或生物大分子样品快速冷冻,然后利用透射电子显微镜收集冷冻样品的图像,后续通过一系列数据处理方法来重构冷冻样品的三维结构。基本分析方法包括单颗粒三维重构法、电子晶体学方法和电子断层扫描成像技术等,其中单颗粒三维重构法是解析蛋白质分子三维结构最常用的方法。单颗粒三维重构法是根据蛋白质分子的不同取向,将电镜视野中数以百万计的单个蛋白质分子颗粒图像进行分类和平均,提高图像信噪比,从而解析得到高分辨率的蛋白质分子三维结构[22]。在冷冻电镜技术中,蛋白质样品通常无需结晶,只需要很少量蛋白质溶液(如在单颗粒三维重构法中只需3 μL左右浓度为1~10 μg/μL的蛋白质溶液)通过快速冷冻即可进行图像收集和处理,可以尽量保持样品的原始状态,得到更有意义的结构分析[23-25]。
结构生物学在Eh致病机制研究中的应用 目前,PDB数据库中收录的Eh蛋白质三维结构共计156个,其中有143个是通过X射线晶体学方法解析的,另外13个是通过核磁共振波谱学技术解析的,暂无通过冷冻电镜技术解析的结构。在已解析的Eh蛋白质结构中有39个是结构蛋白,包括肌动蛋白、肌球蛋白、钙调蛋白等,还有117个是非结构蛋白,主要是参与阿米巴细胞代谢的各类酶。Eh致病过程相关的重要蛋白质当中,三维结构得到解析的有阿米巴穿孔素A和半胱氨酸蛋白酶抑制剂。
阿米巴穿孔素A 在Eh几种重要的毒力因子蛋白质中,最先解析出三维结构的是阿米巴穿孔素A。阿米巴穿孔素是一类存在于滋养体细胞质颗粒中的小分子蛋白家族,包括阿米巴穿孔素A、B和C。当阿米巴滋养体与靶细胞或组织接触时,滋养体可以分泌出阿米巴穿孔素,使得靶细胞表面形成离子通道,破坏靶细胞膜屏障,使靶细胞或组织发生损害[26-27]。早在1982年,科学家就已经发现阿米巴穿孔素,并对其功能进行研究[28]。1992年,Leippe等[29]利用阿米巴穿孔素A的互补脱氧核糖核酸(complementary deoxyribonucleic acid,cDNA)序列,推导出阿米巴穿孔素A的氨基酸序列(即一级结构)是由77个氨基酸残基构成的,其中包括6个半胱氨酸残基。同年,科学家利用圆二色光谱测定出阿米巴穿孔素的二级结构为全α-螺旋构象。2004年,Hecht等[27]利用核磁共振波谱学方法解析了阿米巴穿孔素A的三级结构,证实了此前推测阿米巴穿孔素A的全α-螺旋构象(图 1A),并且通过分子筛色谱、化学修饰和蛋白质交联实验证明了阿米巴穿孔素的激活与pH依赖的蛋白质二聚化有关。

|
| A:A ribbon diagram illustrating the tertiary structure of Amoebapore A.Disulfide bonds are colored in blue, and the N and C terminus are labeled.Helices are numbered by Roman numerals.B:A ribbon representation of the amoebapore A dimer.Residues responsible for electrostatic interactions aredepicted.Helices 1 and 2 forming the hydrophobic surface are labeled.C:The electrostatic potentialof the polymeric molecular surface.Positive potential is indicated in blue and negative potential in red.Reproduced from HECHT O, VAN NULAND NA, SCHLEINKOFER K, et al.Solution structure of the pore-forming protein of Entamoeba histolytica[27], with permission from J Biol Chem. 图 1 阿米巴穿孔素A的三级结构 Fig 1 The tertiary structure of Amoebapore A |
阿米巴穿孔素是水溶性蛋白质,但同时也可以分散在细胞膜上即具有一定的脂溶性。在阿米巴穿孔素A的三维结构中观察到该蛋白质的一侧是亲水性的,另一侧是疏水性的,具有两亲性。阿米巴滋养体可以通过调节酸性物质的分泌调控穿孔素的活性。在酸性环境下,阿米巴穿孔素通过静电作用发生二聚化,亲水结构聚集在内侧,疏水结构暴露在外侧,形成整体疏水性的环状结构(图 1B、C),使穿孔素可以更好地分布在靶细胞膜上,从而导致靶细胞膜出现寡聚孔。阿米巴穿孔素的二聚体活化机制提示,与阿米巴穿孔素结构类似的可溶性球状蛋白质可能具有破坏细胞膜,从而杀死细菌和真核细胞的作用[27]。
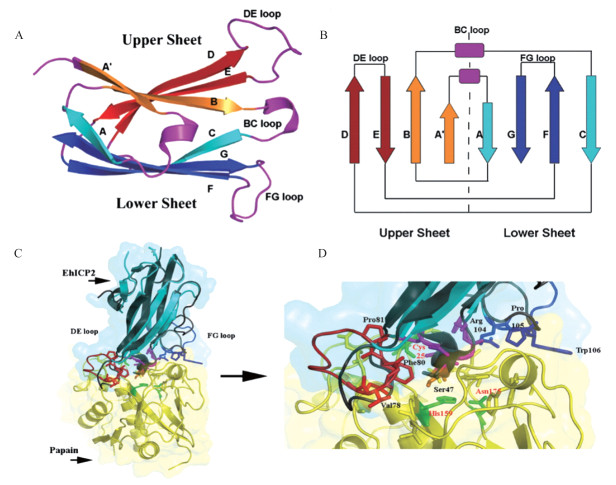
CP抑制剂 CP是Eh另一种重要的毒力因子。Eh胞内含有至少50种CP,其中最主要的是EhCP1、EhCP2和EhCP5。CP最初表达为无活性的酶原,其活性由阿米巴精密的调控机制进行调控,其中包含Chagasin样抑制剂家族的Eh CP抑制剂(Entamoeba histolytica inhibitor forcysteine proteases,EhICP),即EhICP1和EhICP2[30-33]。2011年,Casados-Vázquez等[34]利用X射线衍射技术解析出EhICP2的晶体结构(图 2A)。
|
| A:A Ribbon representation of EhICP2 crystal structure (Orange and red:the upper β-sheet; Cyan and blue:the lower β-sheet; Pink:the helix and the BC, FG, and DE loops).B:Topology diagram of EhICP2.This diagram depicts EhICP2 as a pseudosym metrical molecule in which four β-strands contribute to the formation of each β-sheet. C:Ribbon and surface representation of EhICP2 docked onto Papain (Yellow:Papain; Cyan: EhICP2;Pink: the BC loop; Red: the DE loop; Blue:the FG loop).D:Side chains of EhICP2 that participate in putative interactions in the DE and FG loops are shown in ball-and-stick representation. Green:The catalytic triad of Papain (His-159, Asn-175, and Cys-25).Reproduced from CASADOS-VÁZQUEZ LE, LARA-GONZÁ LEZ S, BRIEBA LG.Crystal structure of the cysteine protease inhibitor 2 from Entamoeba histolytica:functional convergence of a common protein fold[34], with permission from Gene. 图 2 EhICP2的三维晶体结构及其与半胱氨酸蛋白酶相互作用的模型 Fig 2 The crystal structure of EhICP2 and its interaction model with a cysteine protease(Papain) |
EhICP2的空间结构由8条β链构成的2个β-折叠片组成,与免疫球蛋白的折叠方式非常相似。EhICP2中含有致密的疏水核心集团,起到稳定整体结构的作用(图 2A、B)。BC、DE和FG三个环状结构具有一定的柔性,其功能主要与蛋白质间相互作用有关,其中BC环中苏氨酸和丝氨酸残基可以与CP活性中心即催化三联体(His-159,Asn-175和Cys-25)中的半胱氨酸和组氨酸残基相互作用,从而抑制CP活性,而DE和FG环在EhICP2结合CP时可转变构象,起到稳定整体结构的作用(图 2C、D)[34]。CP抑制剂EhICP2晶体结构的解析,有助于在原子水平上理解Eh中CP的活性调控机制,为开发抑制虫体CP药物奠定了基础。
结构生物学在Eh研究中的发展前景 目前在Eh致病过程中起重要作用的Gal/GalNAc凝集素、CP和过氧化物氧化还原酶等毒力因子蛋白质的三维结构尚未得到解析[2, 35-37],未来对这些毒力因子三维结构的解析可能会成为结构生物学在Eh研究领域的热点。
以Eh Gal/GalNAc凝集素为例。研究表明该凝集素包含170 000重链亚单位(heavy subunit of Gal/GalNAc lectin,hgl)、150 000中间亚单位(intermediate subunit of Gal/GalNAc lectin,igl)和35/31 kDa轻链亚单位(light subunit of Gal/GalNAc lectin,lgl),其中hgl是跨膜蛋白,igl和lgl通过糖基磷脂酰肌醇(Glycosyl phosphatidyl inositol,GPI)锚定在细胞膜胞外侧(图 3)。已知hgl由于含有特异性的糖类识别位点(carbohydrate recognition domain,CRD)而具有重要作用,而igl和lgl在阿米巴黏附和入侵过程中发挥的作用尚不明确。另外,Gal/GalNAc凝集素富含半胱氨酸以及由半胱氨酸残基构成的CXXC或CXC基序(“C”为半胱氨酸残基,“X”为任意氨基酸残基),研究表明这一特点与滋养体吞噬宿主细胞和导致宿主细胞凋亡相关[38-41]。由于Gal/GalNAc凝集素的三维结构尚未解析,特别是CXXC或CXC基序的结构特点尚不清楚,Gal/GalNAc凝集素特别是igl和lgl两个亚单位在致病过程中所扮演的角色难以从原子水平上进行推测和理解。Eh中Gal/GalNAc凝集素定位于细胞膜上,属于膜蛋白,不易结晶,难以通过X射线晶体学方法解析其结构,预计将来可能通过冷冻电镜技术获得其三维结构。在Gal/GalNAc凝集素中,hgl和lgl通过共价键连接形成分子量为260 000的异源二聚体,再与igl通过非共价键的方式结合。实验条件下蛋白质间的非共价键不易形成,因此可能需要分别解析hgl-lgl异源二聚体与igl的三维结构,再对Gal/GalNAc凝集素整体的分子结构进行分析。

|
| The Gal/GalNAc lectin of Entamoeba histolytica is composed of three subunits.The transmembrane hgl has a CRD and a short cytoplasmic tail implicated in intracellular signaling.Hgl connects with a lipid-anchored lgl by disulfide bonds, and igl noncovalently associates with the hgl-lgl heterodimer. 图 3 Gal/GalNAc凝集素结构模型 Fig 3 Schematic structural model of Gal/GalNAc lectin |
结语 在Eh致病机制研究中,结构生物学作为一种新兴的生物学研究方法,已经得到一定程度的应用。近些年,越来越多重要的虫体酶、毒力因子和致病蛋白的三维结构被解析,对于Eh致病机制的理解不断深入,并为抗虫药物和潜在疫苗的研发提供了必要条件。但目前Eh中还有很多重要蛋白质的三维结构尚待研究,如与阿米巴对宿主组织和细胞黏附相关的Gal/GalNAc凝集素等。另一方面,冷冻电镜技术尚未在Eh研究领域得到成功的运用。随着技术的进步,特别是冷冻电镜技术的发展,结构生物学方法将更加深入地运用到Eh致病机制的研究当中。
| [1] |
STANLEY SL, J R. Amoebiasis[J]. Lancet, 2003, 361(9362): 1025-1034.
[DOI]
|
| [2] |
MIN X, FENG M, GUAN Y, et al. Evaluation of the C-terminal fragment of entamoeba histolytica Gal/GalNAc lectin intermediate subunit as a vaccine candidate against amebic liver abscess[J]. PLoS Negl Trop Dis, 2016, 10(1): e0004419.
[DOI]
|
| [3] |
BEGUM S, QUACH J, CHADEE K. Immune evasion mechanisms of entamoeba histolytica:progression to disease[J]. Front Microbiol, 2015, 6: 1394.
|
| [4] |
RALSTON KS, SOLGA MD, MACKEY-LAWRENCE NM, et al. Trogocytosis by Entamoeba histolytica contributes to cell killing and tissue invasion[J]. Nature, 2014, 508(7497): 526-530.
[DOI]
|
| [5] |
SHI Y. A glimpse of structural biology through X-ray crystallography[J]. Cell, 2014, 159(5): 995-1014.
[DOI]
|
| [6] |
YAN N. A glimpse of membrane transport through structures-advances in the structural biology of the GLUT glucose transporters[J]. J Mol Biol, 2017, 429(17): 2710-2725.
[DOI]
|
| [7] |
WATSON JD, CRICK FH. Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid[J]. Nature, 1953, 171(4356): 737-738.
[DOI]
|
| [8] |
KENDREW JC, BODO G, DINTZIS HM, et al. A three-dimensional model of the myoglobin molecule obtained by x-ray analysis[J]. Nature, 1958, 181(4610): 662-666.
[DOI]
|
| [9] |
KENDREW JC, DICKERSON RE, STRANDBERG BE, et al. Structure of myoglobin:a three-dimensional Fourier synthesis at 2 A.resolution[J]. Nature, 1960, 185(4711): 422-427.
[DOI]
|
| [10] |
TAYLOR GL. Introduction to phasing[J]. Acta Crystallogr D Biol Crystallogr, 2010, 66(Pt 4): 325-338.
|
| [11] |
DENG D, XU C, SUN P, et al. Crystal structure of the human glucose transporter GLUT1[J]. Nature, 2014, 510(7503): 121-125.
[DOI]
|
| [12] |
BIRCH J, AXFORD D, FOADI J, et al. The fine art of integral membrane protein crystallisation[J]. Methods, 2018, 147: 150-162.
[DOI]
|
| [13] |
MAGNANI F, SERRANO-VEGA MJ, SHIBATA Y, et al. A mutagenesis and screening strategy to generate optimally thermostabilized membrane proteins for structural studies[J]. Nat Protoc, 2016, 11(8): 1554-1571.
[DOI]
|
| [14] |
SAUNDERS M, WISHNIA A, KIRKWOOD JG. The nuclear magnetic resonance spectrum of ribonuclease[J]. J Am Chem Soc, 1957, 79(12): 3289-3290.
[DOI]
|
| [15] |
KUMAR A, WELTI D, ERNST RR. NMR fourier zeugmatography[J]. J Magn Reson, 1975, 18(1): 69-83.
|
| [16] |
CORY DG, FAHMY AF, HAVEL TF. Ensemble quantum computing by NMR spectroscopy[J]. Proc Natl Acad Sci U S A, 1997, 94(5): 1634-1639.
[DOI]
|
| [17] |
BUNDY JG, PAPP B, HARMSTON R, et al. Evaluation of predicted network modules in yeast metabolism using NMR-based metabolite profiling[J]. Genome Res, 2007, 17(4): 510-519.
[DOI]
|
| [18] |
BOTHWELL JH, GRIFFIN JL. An introduction to biological nuclear magnetic resonance spectroscopy[J]. Biol Rev Camb Philos Soc, 2011, 86(2): 493-510.
[DOI]
|
| [19] |
SPRANGERS R, KAY LE. Quantitative dynamics and binding studies of the 20S proteasome by NMR[J]. Nature, 2007, 445(7128): 618-622.
[DOI]
|
| [20] |
JIANG Y, KALODIMOS CG. NMR studies of large proteins[J]. J Mol Biol, 2017, 429(17): 2667-2676.
|
| [21] |
LIAO M, CAO E, JULIUS D, et al. Structure of the TRPV1 ion channel determined by electron cryo-microscopy[J]. Nature, 2013, 504(7478): 107-112.
[DOI]
|
| [22] |
ZHOU ZH. Atomic resolution cryo electron microscopy of macromolecular complexes[J]. Adv Protein Chem Struct Biol, 2011, 82: 1-35.
[DOI]
|
| [23] |
CAO E, LIAO M, CHENG Y, et al. TRPV1 structures in distinct conformations reveal activation mechanisms[J]. Nature, 2013, 504(7478): 113-118.
[DOI]
|
| [24] |
YAN Z, ZHOU Q, WANG L, et al. Structure of the Nav1.4-beta1 complex from electric eel[J]. Cell, 2017, 170(3): 470-482 e411.
[DOI]
|
| [25] |
ZHANG JB, CHANG S, XU P, et al. Structural basis of the proton sensitivity of human GluN1-GluN2A NMDA receptors[J]. Cell Rep, 2018, 25(13): 3582-3590.
[DOI]
|
| [26] |
LEIPPE M, BRUHN H, HECHT O, et al. Ancient weapons:the three-dimensional structure of amoebapore A[J]. Trends Parasitol, 2005, 21(1): 5-7.
|
| [27] |
HECHT O, VAN NULAND NA, SCHLEINKOFER K, et al. Solution structure of the pore-forming protein of Entamoeba histolytica[J]. J Biol Chem, 2004, 279(17): 17834-17841.
[DOI]
|
| [28] |
LYNCH EC, ROSENBERG IM, GITLER C. An ion-channel forming protein produced by Entamoeba histolytica[J]. EMBO J, 1982, 1(7): 801-804.
[DOI]
|
| [29] |
LEIPPE M, TANNICH E, NICKEL R, et al. Primary and secondary structure of the pore-forming peptide of pathogenic Entamoeba histolytica[J]. EMBO J, 1992, 11(10): 3501-3506.
[DOI]
|
| [30] |
WILSON IW, WEEDALL GD, LORENZI H, et al. Genetic diversity and gene family expansions in members of the genus entamoeba[J]. Genome Biol Evol, 2019, 11(3): 688-705.
[DOI]
|
| [31] |
BRUCHHAUS I, JACOBS T, LEIPPE M, et al. Entamoeba histolytica and entamoeba dispar:differences in numbers and expression of cysteine proteinase genes[J]. Mol Microbiol, 1996, 22(2): 255-263.
|
| [32] |
SATO D, NAKADA-TSUKUI K, OKADA M, et al. Two cysteine protease inhibitors, EhICP1 and 2, localized in distinct compartments, negatively regulate secretion in Entamoeba histolytica[J]. FEBS Lett, 2006, 580(22): 5306-5312.
[DOI]
|
| [33] |
CORNICK S, MOREAU F, CHADEE K. Entamoeba histolytica cysteine proteinase 5 evokes mucin exocytosis from colonic goblet cells via αvβ3 integrin[J]. PLoS Pathog, 2016, 12(4): e1005579.
[DOI]
|
| [34] |
CASADOS-VÁZQUEZ LE, LARA-GONZÁLEZ S, BRIEBA LG. Crystal structure of the cysteine protease inhibitor 2 from Entamoeba histolytica:functional convergence of a common protein fold[J]. Gene, 2011, 471(1-2): 45-52.
[DOI]
|
| [35] |
GONZALEZ-VÁZQUEZ MC, CARABARIN-LIMA A, BAYLON-PACHECO L, et al. Obtaining of three recombinant antigens of Entamoeba histolytica and evaluation of their immunogenic ability without adjuvant in a hamster model of immunoprotection[J]. Acta Trop, 2012, 122(2): 169-176.
[DOI]
|
| [36] |
MILLER HW, SULEIMAN RL, RALSTON KS. Trogocytosis by entamoeba histolytica mediates acquisition and display of human cell membrane proteins and evasion of lysis by human serum[J]. mBio, 2019, 10(2): e00068-19.
[DOI]
|
| [37] |
GUO X, BARROSO L, LYERLY DM, et al. CD4+and CD8+T cell-and IL-17-mediated protection against Entamoeba histolytica induced by a recombinant vaccine[J]. Vaccine, 2011, 29(4): 772-777.
[DOI]
|
| [38] |
MARIE C, PETRI WA, J R. Regulation of virulence of Entamoeba histolytica[J]. Annu Rev Microbiol, 2014, 68: 493-520.
[DOI]
|
| [39] |
PETRI WA, HAQUE R, MANN BJ. The bittersweet interface of parasite and host:lectin-carbohydrate interactions during human invasion by the parasite Entamoeba histolytica[J]. Annu Rev Microbiol, 2002, 56: 39-64.
[DOI]
|
| [40] |
CHENG XJ, HUGHES MA, HUSTON CD, et al. Intermediate subunit of the Gal/GalNAc lectin of Entamoeba histolytica is a member of a gene family containing multiple CXXC sequence motifs[J]. Infect Immun, 2001, 69(9): 5892-5898.
[DOI]
|
| [41] |
CHENG XJ, TSUKAMOTO H, KANEDA Y, et al. Identification of the 150-kDa surface antigen of Entamoeba histolytica as a galactose-and N-acetyl-D-galactosamine-inhibitable lectin[J]. Parasitol Res, 1998, 84(8): 632-639.
[DOI]
|
 2020, Vol. 47
2020, Vol. 47


