肺癌的发生率和死亡率居高不下,全世界每年约有180万例肺癌病例。肺癌具有异质性,分为多种亚型,肺腺癌是最常见的亚型。由于肺癌被诊断时多已处于晚期且伴随转移,预后相对较差,探究肺癌发病与转移的分子机制意义重大[1-2]。ADP-核糖基化是一种由细胞内聚(ADP-核糖)聚合酶[poly(ADP-ribose)polymerase,PARP]催化的翻译后修饰反应,PARP家族中已鉴定出17个成员[3]。PARP家族成员及其介导的ADP-核糖基化反应在细胞生命进程中发挥重要作用,包括DNA修复、细胞黏附、脂质代谢、细胞增殖或死亡等[4-5]。
PARP家族成员9(PARP9),又被称为B侵袭性淋巴瘤蛋白(B-aggressive lymphoma-1 protein,BAL1),最初作为弥漫性大B细胞淋巴瘤(diffuse large B-cell lymphoma,DLB-CL)的风险基因被报道,PARP9过表达会促进恶性B细胞迁移[6]。PARP9的N末端为两个Macro结构域,C末端为PARP催化结构域,因缺乏对自身的催化活性,早期认为PARP9是无活性的单-ADP-核糖基转移酶[7]。然而,Yang等[8]指出PARP9/DTX3L异二聚体介导对泛素的单-ADP-核糖基化。研究表明,PARP9主要在DNA损伤修复和病毒感染过程中发挥作用[9-10]。多项研究揭示PARP9在肿瘤中表达上调,如弥漫性大B淋巴瘤、前列腺癌和乳腺癌等,并在肿瘤发展中发挥重要作用[6, 11-12],但其在肺腺癌中的作用尚未见报道。
本研究首先对PARP9在肺腺癌中的表达和预后意义进行分析,并通过实验探究其对肺腺癌细胞H1299与A549迁移和侵袭能力的影响,以增强对于PARP9在肺腺癌中的作用认识,同时为探寻肺腺癌的诊疗靶点提供新思路。
材料和方法数据集和数据库 采用常见肿瘤数据库进行基因表达分析。癌症基因组图谱(The Cancer Genome Atlas,TCGA)计划收录了多种癌症的临床信息和基因组数据,临床蛋白质组学肿瘤分析联盟(clinical proteomic tumor analysis consortium,CPTAC)整合了肿瘤基因组和蛋白质组的数据。通过UALCAN[13](http://ualcan.path.uab.edu/index.html)对TCGA和CPTAC数据进行挖掘和分析,在UALCAN中进行基因表达分析和生存分析。基因表达数据库(Gene Express Omnibus,GEO)由美国国立生物技术信息中心创建,储存高通量测序数据。我们使用GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)的肺腺癌数据集GSE31210进行基因表达分析。
免疫组化分析 75例肺腺癌组织及配对正常组织样本由上海芯超生物科技有限公司提供(编号:HLugA150CS03),3例因脱片无法统计,且存在少量临床数据缺失。使用PARP9抗体(17535-1-AP,美国Proteintech公司)进行免疫组化染色(1∶1 000),根据免疫染色强度分级(0:无染色;1:弱染色;2:中等染色;3:强染色),同时按照免疫染色百分比分级(0级:0;0 < 1级≤25%;25% < 2级≤50%;51% < 3级≤75%;75% < 4级≤100%)。免疫染色强度评分与免疫染色百分比评分的乘积为PARP9染色总分,然后将样本分为PARP9表达阴性(总分≤3)和PARP9表达阳性(总分 > 3)。
细胞系和细胞培养 人肺腺癌细胞A549和H1299来自中国科学院上海细胞库。细胞在含有10%胎牛血清(德国PAN公司)和1%青霉素和链霉素(上海翊圣生物科技有限公司)的RPMI 1640培养基(美国Gibco公司)中培养,培养条件为37 ℃、5% CO2、潮湿培养。
PARP9缺陷和PARP9回补细胞系的构建 使用CRISPR/Cas9敲除PARP9,将靶向PARP9的两条sgRNA连接至质粒载体(sgRNA1:5'-GAGATA-GCTGTCACGGGAGCAGG-3'和sgRNA2:5'-TT-ATCCCATTCCATCCACCGAGG-3'),然后转染A549和H1299细胞,梯度分盘后使用嘌呤霉素筛选3天,正常培养,直至长出肉眼可见的克隆。对单克隆基因组测序,并进行Western blot检测,筛选成功敲除的细胞。使用慢病毒感染来恢复PARP9表达缺陷细胞中的PARP9。将慢病毒包装系统的3个质粒(PCDH、PxpaX2、PMD2g)转染到HEK293T细胞中进行病毒包装,用含有病毒的上清液感染敲除细胞。流式分选后,通过Western blot鉴定阳性感染的细胞。
Western blot 检测 使用SDS-PAGE分离蛋白,用半干转膜仪将蛋白质转移到经甲醇激活的PVDF膜上,将PVDF膜与5%的脱脂牛奶一起孵育,封闭30 min后洗膜,然后与含Flag(M20008)、β-actin(P30002)和PARP9(17535-AP)的一抗,在4 ℃下孵育过夜,洗膜后将PVDF膜与偶联辣根过氧化物酶的二抗在室温下孵育30 min。使用ECL系统记录结果。
细胞迁移和侵袭检测 使用无血清培养基重悬细胞,计数后加入Transwell小室的内室,将小室置于24孔板中,外室加入完全培养基,培养36 h。室温下用4%多聚甲醛固定细胞,并用0.2%结晶紫染色,用棉签轻轻擦去内室的细胞,对外室迁移的细胞进行拍照计数。进行侵袭实验时,提前在Transwell小室的内室加入用无血清培养基稀释的Matrigel,后续操作同迁移实验。
统计学分析 使用GraphPad Prism 5.0对实验结果进行绘图和统计分析;使用Image J对细胞进行计数;使用独立样本t检验比较两组数据,以x±s显示结果;使用Log-rank检验进行PARP9高表达和低表达患者的生存差异分析;使用χ2检验进行PARP9表达与患者临床病理信息的相关性分析。实验重复3次,P < 0.05为差异有统计学意义。
结果PARP9在肺腺癌患者中高表达且预后不良
我们首先通过分析TCGA数据库中的mRNA数据来评估PARP9在肺腺癌中的表达水平。结果显示,PARP9在肺腺癌组织中的表达水平显著高于邻近正常组织(P < 0.001,图 1A)。同时,来自GEO数据库的数据进一步证实了这一结果(P=0.002,图 1B)。接下来,我们分析了CPTAC数据库中PARP9的蛋白水平。与mRNA结果一致,肺腺癌组织中PARP9的蛋白水平也显著高于正常组织(P < 0.001,图 1C)。使用PARP9的抗体对72例肺腺癌样本及其配对正常组织进行免疫组化染色,进一步评估了PARP9的表达,结果同样显示,与正常组织相比,PARP9在肺腺癌中显著高表达(t=8.909,P < 0.001,图 1D)。结合免疫组化染色结果,我们对PARP9的表达与肺腺癌患者临床病理信息之间的相关性进行了分析,结果表明PARP9的表达水平与肺腺癌患者的临床分期(χ2=4.309,P=0.038)和淋巴结转移(χ2=3.916,P=0.048)具有相关性,而与性别、年龄、肿瘤大小无相关性(表 1)。此外,TCGA数据库的Kaplan-Meier生存分析表明,PARP9高表达的肺腺癌患者总生存期显著短于PARP9低表达的患者(P=0.041,图 1E),提示PARP9的表达与肺腺癌患者的不良预后正相关。
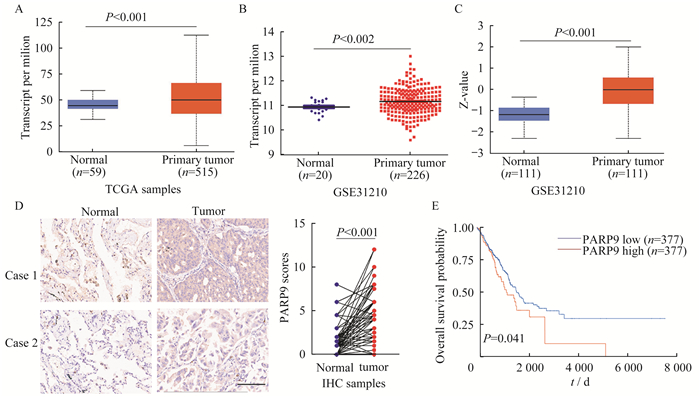
|
| A-C:PARP9 mRNA in lung adenocarcinoma and normal tissues in TCGA database,GSE31210(GEO database)and CPTAC database;D:Immunohistochemical staining and scoring statistics of PARP9 in lung adenocarcinoma tissue chips(bar=100 µm);E:Overall survival of lung adenocarcinoma patients with high or low PARP9 expression in the TCGA database. 图 1 PARP9在肺腺癌及正常组织中的表达与预后 Fig 1 Expression and prognosis of PARP9 in lung adenocarcinoma and normal tissues |
| Clinical information | Case(n) | PARP9 expression in lung adenocarcinoma | χ2 | P | |
| Negtive | Positive | ||||
| Sex | 0.210 | 0.647 | |||
| Female | 40 | 15 | 25 | ||
| Male | 31 | 10 | 21 | ||
| Age(y) | 0.098 | 0.754 | |||
| ≤50 | 13 | 5 | 8 | ||
| >50 | 59 | 20 | 39 | ||
| Tumor size(cm) | 0.006 | 0.941 | |||
| ≤5 | 6 | 2 | 4 | ||
| >5 | 66 | 23 | 43 | ||
| Clinical stage | 4.309 | 0.038 | |||
| Ⅰ-Ⅱ | 46 | 20 | 26 | ||
| Ⅲ-Ⅳ | 26 | 5 | 21 | ||
| Lymph node metastasis | 3.916 | 0.048 | |||
| No | 41 | 17 | 24 | ||
| Yes | 27 | 5 | 22 | ||
PARP9缺陷显著抑制肺腺癌细胞的迁移与侵袭 我们设计了两条靶向PARP9第3号外显子的向导RNA,在H1299与A549细胞中敲除PARP9,对获得的细胞单克隆提取基因组在敲除位点进行DNA扩增与Sanger测序,选择基因组含有非3整数倍碱基缺失的单克隆,使用PARP9抗体进行Western blot验证,结果显示敲除组内PARP9蛋白的表达丢失(图 2A、2B),表明PARP9缺陷细胞株构建成功。由于PARP9的表达水平与肺腺癌患者淋巴结转移显著相关(表 1),使用Transwell小室对PARP9在肺腺癌细胞迁移中的功能进行测定。与野生型细胞相比,PARP9缺陷型H1299细胞与A549细胞的迁移能力均显著降低(H1299组:KO1,t=4.206,P=0.003;KO2,t=15.81,P < 0.001;A549组:KO1,t=9.310,P < 0.001;KO2,t=7.795,P < 0.001)(图 2C),提示PARP9缺陷抑制肺腺癌细胞的迁移。肿瘤细胞侵袭能力同样是影响肿瘤转移的重要因素,在Transwell小室的内室添加基质胶预处理,进行侵袭实验。与野生型细胞相比,PARP9缺陷的H1299细胞和A549细胞的侵袭能力显著降低(H1299组:KO1,t=18.460,P < 0.001;KO2,t=12.730,P < 0.001;A549组:KO1,t=3.754,P=0.006;KO2,t=4.573,P=0.002)(图 2D),表明PARP9缺陷抑制肺腺癌细胞的侵袭。
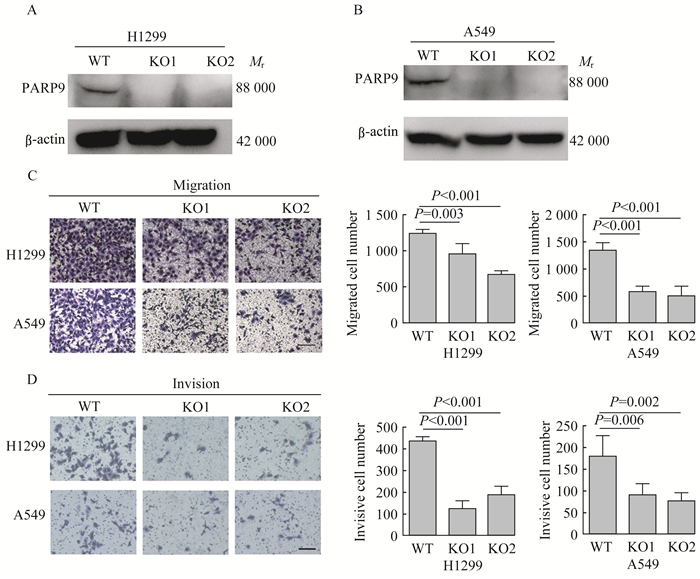
|
| Validation of PARP9 knockout in H1299 cells(A)and A549 cells(B)assessed by Western blot. Migration assay(C)and Invasion assay(D) of H1299 cells and A549 cells after PARP9 knockout(bar=100 µm). 图 2 PARP9缺陷对肺腺癌细胞的迁移和侵袭能力的影响 Fig 2 Effects of PARP9 deficiency on the migration and invision of lung adenocarcinoma cells |
PARP9恢复表达恢复细胞的迁移和侵袭 为了排除CRISPR-Cas9技术可能存在的脱靶效应,进一步证实PARP9在肺腺癌细胞迁移和侵袭中的作用,通过慢病毒感染在PARP9缺陷的A549细胞中恢复表达PARP9。以空病毒感染作为对照,使用Transwell小室检测A549细胞的迁移和侵袭能力。Western blot结果表明(图 3A),慢病毒感染后,PARP9缺陷型A549细胞中PARP9表达恢复至与野生型细胞相当的水平。Transwell迁移和侵袭实验结果表明(图 3B、3C),PARP9缺陷显著减弱A549细胞的迁移和侵袭能力(迁移:t=11.250,P < 0.001;侵袭:t=4.916;P=0.001),恢复表达PARP9后,迁移和侵袭的减弱得以恢复(迁移:t=7.576,P < 0.001;侵袭:t=3.632;P=0.007),由此证实PARP9在调节肺腺癌细胞迁移和侵袭中的作用。

|
| A:Reconstitution of PARP9 in PARP9-deficient A549 cells was confirmed by Western blot;B:Migration assay of A549 cells after reconstitution of PARP9;C:Invasion assays of A549 cells after reconstitution of PARP9(bar=100 µm). 图 3 PARP9回复表达对肺腺癌细胞的迁移和侵袭能力的影响 Fig 3 Effects of PARP9 reconstitution on the migration and invasion of lung adenocarcinoma cells |
肺癌中非小细胞肺癌占比85%,肺腺癌和肺鳞癌是最常见的非小细胞肺癌亚型,肺腺癌占肺癌总数的40%,不同肺癌亚型具有不同的遗传驱动因素和预后特征[14]。基因组学研究揭示了肺腺癌的驱动基因,如TP53、KRAS和EGFR等的突变,对肺腺癌诊疗具有重要的指导意义,推动了靶向治疗进展[15]。本研究中,我们发现PARP9在肺腺癌组织表达上调且预后不良,使用同样的数据库分析时,肺鳞癌中PARP9的转录与预后相关性不显著,提示PARP9可能与肺腺癌进展相关,该结果是否由不同肺癌亚型表达模式的差异导致,需通过规模化分析去验证。
既往研究揭示了PARP9在一些实体瘤中的表达与作用:Bachmann等[11]发现在前列腺癌中PARP9参与调节细胞增殖与存活,Yang等[12]发现PARP9在乳腺癌中高表达并促进细胞迁移,也有研究通过数据挖掘发现PARP9表达与低级别胶质瘤的肿瘤微环境显著相关,可能通过调控肿瘤微环境参与胶质瘤进展[16]。基于此,本研究首次探究了PARP9在肺腺癌中的表达与作用。通过数据库与组织芯片样本分析,我们发现PARP9在肺腺癌中上调表达,临床病理特征相关性分析结果提示PARP9高表达预示更高的淋巴转移风险。为了探究PARP9在肺腺癌中的生物学作用,我们构建了PARP9敲除与恢复表达的肺腺癌细胞株,通过Transwell实验证明PARP9可以促进肺腺癌细胞的迁移与侵袭,该结果与组织样本中PARP9高表达预示肺腺癌转移高风险结果一致。
目前对于PARP9作用的分子机制的研究多与免疫相关,PARP9参与IFN-γ信号传导,调控机体免疫应答,控制病毒感染[10, 17]。免疫细胞中IFN-γ诱导PARP9的表达,PARP9通过刺激信号转导与转录激活因子1(signal transducerand activator of transcription 1,STAT1)磷酸化影响下游信号[18-19]。STAT1在肿瘤中异常激活,参与肿瘤侵袭转移、化疗耐药等过程,其表达在多种实体癌中具有预后价值[20-21]。有研究表明PARP9通过调控下游STAT1参与前列腺癌与DLB-CL的进展[11, 19],此外,Xu等[22]对胶质瘤的研究中发现PARP9高表达患者体内存在多条免疫相关信号通路的富集,如JAK-STAT信号通路、Toll样受体信号通路等。另有两项报道指出PARP9在食管癌与宫颈癌中的表达受上游非编码RNA的调控,进而影响细胞的致瘤性[23-24]。我们的研究表明PARP9在调控肺腺癌细胞迁移中发挥重要作用,可能与肺腺癌转移相关,但具体的机制仍有待探究。TCGA数据库的肺腺癌样本中,与PARP9表达存在高度相关性的基因也主要与免疫相关,越来越多的研究揭示了免疫信号通路在肺腺癌转移和预后中的重要作用[25-26],PARP9是否会通过免疫相关信号参与对肺腺癌细胞迁移侵袭的调控仍有待探究。
本研究存在一些不足:首先是PARP9对肺腺癌细胞迁移侵袭能力影响的探究局限于细胞层面,我们曾尝试进行裸鼠尾静脉注射实验进行体内迁移验证,但注射后野生组与敲除组均未见转移瘤,考虑细胞体内转移能力较弱或未达到合适的实验条件,仍需进一步摸索,我们采用两个肺腺癌细胞株并进行恢复表达以验证结果的可靠性;其次是对于PARP9调控肺腺癌细胞迁移和侵袭中的分子机制尚未明确,我们目前考虑通过生物信息学分析结合互作蛋白筛选进一步展开对PARP9在肺腺癌中作用的分子机制探索。
综上所述,本项研究发现PARP9在肺腺癌组织中高表达,高表达PARP9的肺腺癌患者具有更短的总体生存期、更高的临床分期且更易出现淋巴结转移,在肺腺癌细胞中敲除PARP9后细胞的迁移和侵袭能力被显著抑制,而恢复表达PARP9则恢复了肺腺癌细胞的迁移和侵袭能力。本研究指出PARP9与肺腺癌的进展相关,PARP9可能参与肺腺癌转移,提示PARP9具有成为肺腺癌预测性生物标记物的可能,为进一步明确PARP9在肿瘤中的作用提供了新的理论依据。我们将继续开展对PARP9作用的分子机制探索,以进一步明确PARP9在肺腺癌中的作用。
作者贡献声明 郑芬 实验设计和执行,数据整理和分析,论文撰写和修订。周晓敏 实验执行,论文撰写和修订。吴家雪 实验设计和指导,论文审阅和修订。
利益冲突声明 所有作者均声明不存在利益冲突。
| [1] |
WADOWSKA K, BIL-LULA I, TREMBECKI Ł, et al. Genetic markers in lung cancer diagnosis: a review[J]. Int J Mol Sci, 2020, 21(13): 4569.
[DOI]
|
| [2] |
DE SOUSA VML, CARVALHO L. Heterogeneity in lung cancer[J]. Pathobiology, 2018, 85(1-2): 96-107.
[DOI]
|
| [3] |
GRIMALDI G, CORDA D. ADP-ribosylation and intracellular traffic: an emerging role for PARP enzymes[J]. Biochem Soc Trans, 2019, 47(1): 357-370.
[DOI]
|
| [4] |
GIBSON BA, KRAUS WL. New insights into the molecular and cellular functions of poly(ADP-ribose) and PARPs[J]. Nat Rev Mol Cell Biol, 2012, 13(7): 411-424.
[DOI]
|
| [5] |
HOPP AK, HOTTIGER MO. Uncovering the invisible: mono-ADP-ribosylation moved into the spotlight[J]. Cells, 2021, 10(3): 680.
[DOI]
|
| [6] |
AGUIAR RC, YAKUSHIJIN Y, KHARBANDA S, et al. BAL is a novel risk-related gene in diffuse large B-cell lymphomas that enhances cellular migration[J]. Blood, 2000, 96(13): 4328-4334.
[DOI]
|
| [7] |
AGUIAR RC, TAKEYAMA K, HE C, et al. B-aggressive lymphoma family proteins have unique domains that modulate transcription and exhibit poly(ADP-ribose)polymerase activity[J]. J Biol Chem, 2005, 280(40): 33756-33765.
[DOI]
|
| [8] |
YANG CS, JIVIDEN K, SPENCER A, et al. Ubiquitin modification by the E3 ligase/ADP-ribosyltransferase Dtx3L/Parp9[J]. Mol Cell, 2017, 66(4): 503-516.
[DOI]
|
| [9] |
YAN Q, XU R, ZHU L, et al. BAL1 and its partner E3 ligase, BBAP, link Poly(ADP-ribose) activation, ubiquitylation, and double-strand DNA repair independent of ATM, MDC1, and RNF8[J]. Mol Cell Biol, 2013, 33(4): 845-857.
[DOI]
|
| [10] |
XING J, ZHANG A, DU Y, et al. Identification of poly(ADP-ribose) polymerase 9(PARP9) as a noncanonical sensor for RNA virus in dendritic cells[J]. Nat Commun, 2021, 12(1): 2681.
[DOI]
|
| [11] |
BACHMANN SB, FROMMEL C, CAMICIA R, et al. DTX3L and ARTD9 inhibit IRF1 expression and mediate in cooperation with ARTD8 survival and proliferation of metastatic prostate cancer cells[J]. Mol Cancer, 2014, 13(1): 125.
[DOI]
|
| [12] |
TANG X, ZHANG H, LONG Y, et al. PARP9 is overexpressed in human breast cancer and promotes cancer cell migration[J]. Oncol Lett, 2018, 16(3): 4073-4077.
|
| [13] |
CHANDRASHEKAR DS, BASHEL B, BALASUBRA-MANYA SAH, et al. UALCAN: a portal for facilitating tumor subgroup gene expression and survival analyses[J]. Neoplasia, 2017, 19(8): 649-658.
[DOI]
|
| [14] |
RELLI V, TREROTOLA M, GUERRA E, et al. Abandoning the notion of non-small cell lung cancer[J]. Trends Mol Med, 2019, 25(7): 585-594.
[DOI]
|
| [15] |
XU JY, ZHANG C, WANG X, et al. Integrative proteomic characterization of human lung adenocarcinoma[J]. Cell, 2020, 182(1): 245-261.
[DOI]
|
| [16] |
LIANG H, HUANG C. Identification of tumor microenvironment-related genes in lower-grade gliomas by mining TCGA database[J]. Transl Cancer Res, 2020, 9(8): 4583-4595.
[DOI]
|
| [17] |
RUSSO LC, TOMASIN R, MATOS IA, et al. The SARS-CoV-2 Nsp3 macrodomain reverses PARP9/DTX3L-dependent ADP-ribosylation induced by interferon signaling[J]. J Biol Chem, 2021, 297(3): 101041.
[DOI]
|
| [18] |
IWATA H, GOETTSCH C, SHARMA A, et al. PARP9 and PARP14 cross-regulate macrophage activation via STAT1 ADP-ribosylation[J]. Nat Commun, 2016, 7: 12849.
[DOI]
|
| [19] |
CAMICIA R, BACHMANN SB, WINKLER HC, et al. BAL1/ARTD9 represses the anti-proliferative and pro-apoptotic IFNγ-STAT1-IRF1-p53 axis in diffuse large B-cell lymphoma[J]. J Cell Sci, 2013, 126(9): 1969-1980.
|
| [20] |
VERHOEVEN Y, TILBORGHS S, JACOBS J, et al. The potential and controversy of targeting STAT family members in cancer[J]. Semin Cancer Biol, 2020, 60: 41-56.
[DOI]
|
| [21] |
ZHANG J, WANG F, LIU F, et al. Predicting STAT1 as a prognostic marker in patients with solid cancer[J]. Ther Adv Med Oncol, 2020, 12: 1-16.
|
| [22] |
XU H, CHAI S, WANG Y, et al. Molecular and clinical characterization of PARP9 in gliomas: a potential immunotherapeutic target[J]. CNS Neurosci Ther, 2020, 26(8): 804-814.
[DOI]
|
| [23] |
MA Y, ZHANG D, WU H, et al. Circular RNA PRKCI silencing represses esophageal cancer progression and elevates cell radiosensitivity through regulating the miR-186-5p/PARP9 axis[J]. Life Sci, 2020, 259: 118168.
[DOI]
|
| [24] |
TAO L, WANG X, ZHOU Q. Long noncoding RNA SNHG16 promotes the tumorigenicity of cervical cancer cells by recruiting transcriptional factor SPI1 to upregulate PARP9[J]. Cell Biol Int, 2020, 44(3): 773-784.
[DOI]
|
| [25] |
LIN W, CHEN Y, WU B, et al. Identification of the pyroptosis-related prognostic gene signature and the associated regulation axis in lung adenocarcinoma[J]. Cell Death Discov, 2021, 7(1): 161.
[DOI]
|
| [26] |
XING Y, LIN Y, ZHANG Y, et al. Novel cytoplasmic lncRNA IKBKBAS promotes lung adenocarcinoma metastasis by upregulating IKKβ and consequential activation of NF-κB signaling pathway[J]. Cell Death Dis, 2021, 12(11): 1004.
[DOI]
|
 2022, Vol. 49
2022, Vol. 49


