2. 复旦大学附属中山医院心血管内科 上海 200032;
3. 上海市心血管病研究所 上海 200032;
4. 中国科学院上海营养与健康研究所营养代谢与食品安全重点实验室 上海 200031
2. Department of Cardiology, Zhongshan Hospital, Fudan University, Shanghai 200032, China;
3. Shanghai Institute of Cardiovascular Disease, Shanghai 200032, China;
4. Key Laboratory of Nutrition, Metabolism and Food Safety, Shanghai Institute of Nutrition and Health, Chinese Academy of Sciences, Shanghai 200031, China
肥胖作为一种全球流行病[1],会增加多种慢性疾病风险[2],降低预期寿命[3],带来严重的个人和社会经济负担[4]。目前,大多数流行病学研究都将体重指数(body mass index,BMI)作为衡量肥胖的指标,但BMI无法区分瘦质量(去除脂肪后质量)和脂肪质量[5]。同时,身体不同区域的脂肪分布能够独立于BMI对健康造成影响[6]。研究表明,Gynoid区域[7]、Android区域[7]及Android与Gynoid区域脂肪含量之比(Android-to-Gynoid fat ratio,AGR)均是与健康和疾病有关的、用于表征肥胖表型的重要指标[8-10](图 1)。AGR是中国肥胖儿童和青少年心脏代谢(特别是糖代谢)危险因素的强效独立预测因子之一[11],其与胰岛素抵抗、低密度脂蛋白和极低密度脂蛋白的浓度升高密切相关[9, 11]。双能X射线吸收法(dual-energy X-ray absorptiometry,DXA)是一种利用身体不同组织(如瘦质量、脂肪质量等)对X光吸收率不同的原理来测量体内脂肪含量的方法,具有较高的准确性和敏锐度[12]。

|
| Android region was defined as the area between the ribs and the pelvis, and was completely covered by the trunk region. The upper demarcation was 20% of the distance from the iliac crest and the neck; the lower demarcation was at the top of the pelvis. Gynoid region included the area of the hips and the upper thighs, and it overlapped the leg and trunk areas; the upper demarcation was below the top of the iliac crest at a distance as 1.5 times as the height of the Android region.The total height of the Gynoid region was as 2 times as the height of the Android region[7]. 图 1 Android和Gynoid的脂肪区域 Fig 1 Fat areas in Android and Gynoid region |
肥胖是一种由多种因素引起的慢性代谢性疾病,与遗传因素、不良的饮食习惯和生活方式(如久坐不动、缺乏运动、睡眠不足等)、心理因素(如压力和情绪等)、药物服用以及社会环境的改变等有关[13]。其中,饮食摄入是导致肥胖和治疗肥胖的重要环境因素[14]。Meta分析发现:摄入红肉和加工肉类与肥胖风险增加以及较高的BMI直接相关[15],减少含糖饮料的摄入量将降低肥胖症及其相关疾病的患病率[16]。越来越多的证据表明,肠道微生物在代谢中的作用能够影响肥胖等代谢性疾病的发生[17]。肠道微生物群很可能是饮食影响人类健康和疾病的中介因素。目前关于由饮食诱导的肠道菌群变化及与其临床结局(如肥胖)相关的研究非常有限,且大多集中在特定膳食的研究。Dong等[18]发现,高蛋白低能量的减重膳食可改变肥胖人群中的肠道微生物结构。目前尚缺乏来自于整体饮食结构、肠道微生物及肥胖表型的整合人群研究。基于以上背景,本研究旨在对饮食结构和肥胖之间相关联的肠道微生物因素作初步的探索。
资料和方法研究对象 本研究纳入来自复旦大学自我报告健康的8名研究生志愿者(6男2女,年龄22~28岁)。入组标准:(1)在粪便采集前2周内未用过抗生素类药物或益生菌;(2)无腹泻、肠炎、肠癌等肠道相关疾病;(3)无盆腔放疗、化疗史。志愿者从S01~S08随机编号。本研究通过复旦大学生命科学学院伦理委员会伦理审查(批件号:BE1828)。所有志愿者均签署知情同意书。
粪便样本采集、DNA提取及宏基因组文库制备 粪便样本采集于2019年8月,使用未添加任何保存液的5 mL粪便样本采集管(中国朗斌生物试剂耗材有限公司)采集8名志愿者经匀质化的新鲜粪便,所有样本均在采集后30 min内置于-80 ℃冰箱冷冻保存。使用TIANamp Stool DNA试剂盒[中国天根生化科技(北京)有限公司],根据说明书提取粪便样本总DNA。DNA浓度通过EqualbitTM dsDNA HS分析试剂盒(南京诺唯赞生物科技股份有限公司)在Invitrogen Qubit3.0(美国赛默飞公司)上进行荧光定量。使用Tn5转座酶DNA文库构建试剂盒(美国APExBIO公司),投入1 ng DNA构建Illumina测序文库,用Agilent生物分析仪2200(美国安捷伦公司)确定每个文库的插入片段大小和浓度。
全基因组鸟枪法测序和数据处理 委托南京诺禾致源生物信息科技有限公司采用Illumina Novaseq6000平台对粪便微生物宏基因组文库进行鸟枪法测序。在此过程中,使用FastQC软件(版本号0.11.8)对原始下机数据进行质量检测;使用过滤软件Trimmomatic(版本号0.33),切除接头序列、引物序列以及低质量的碱基;采用Bowtie2(版本号2.3.4.3)将质控后的数据与人类参考基因组进行比对,进一步剔除污染序列;获得质控数据之后,使用MetaPhlAn 3.0软件,先从全基因组数据库中找出物种特异性标记基因,再利用这些标志基因的数据库对高通量测序得到的鸟枪序列进行注释[19],获得微生物群落的组成,并在种水平计算各细菌相对丰度。α-多样性分析中,使用Shannon指数和Simpson指数衡量样品内微生物丰富度和均一度。β-多样性分析中,使用Bray-Curtis矩阵法对样品间群落结构差异进行计算。
3天24 h饮食回顾数据收集 为了评估志愿者平均每日的谷类、蔬菜、水果、畜禽肉类、奶及奶制品、豆及豆制品、鱼虾贝类、甜点和饮料等饮食组分的摄入情况,在粪便样本采集的前3天(包含2个工作日和1个周末),经过培训的调查员采用开放式调查表,每天面对面询问志愿者前1天(从调查日午夜到前一日午夜)摄入的所有食物和饮料。使用汪之顼等[20]编撰的《回顾性饮食调查辅助参照食物图谱》估计每种食物的摄入量。
肥胖表型数据采集 肥胖表型数据采集所用DXA测量仪为美国GE Lunar Prodigy(美国GE Healthcare公司)扫描仪,分析软件为13.31版。在DXA扫描期间,志愿者被要求身体笔直平躺并处于静止状态直到扫描结束。DXA扫描后,7种与肥胖相关的身体脂肪表型(即肥胖表型)被纳入研究,包括躯干脂肪含量(简称Trunk)、Android区域脂肪含量(简称Android)、Gynoid区域脂肪含量(简称Gynoid)、AGR、全身脂肪含量(简称TBF)、BMI、腰臀比(waist-to-hip ratio,WHR)。Trunk、Android和Gynoid以该区域脂肪质量占该区域脂肪、肌肉、骨矿盐含量三者总质量的百分比表示,而全身脂肪含量以全身脂肪质量占总体重的百分比表示。肥胖表型采集过程中,要求受试者脱掉鞋子,使用测深仪测量受试者的身高;使用数字刻度测量受试者的体重;BMI以体重(kg)除以身高(m)的平方表示。使用软尺在呼气末、吸气未开始时测量经脐部中心的水平围长,即腰围;使用软尺在呼气末、吸气未开始时测量臀部最宽处,即臀围;WHR为腰围与臀围的比率。
统计学分析 基于样本间的Jensen-Shannon距离,利用围绕中心点划分算法(partitioning around medoid,PAM)[21],把肠道微生物属水平在高维数据空间中客观存在的聚集效应(即肠型)呈现出来,最佳分类数目通过Calinski-Harabasz(CH)指数确定[22],肠型的可视化通过使用R软件的ade4包的类间分析(between-class analysis,BCA)构建图形来实现[23]。在对饮食结构和肠道微生物做关联性分析时,使用Bray-Curtis距离估算微生物距离矩阵,使用Euclidean距离估算3天24 h饮食结构数据距离矩阵,使用普氏分析探索饮食结构和肠道微生物之间的关联。使用双侧t检验分析不同肠型之间饮食组分是否存在统计学差异。使用Wilcoxon秩和检验分析性别对肠道微生物的构成是否存在统计学差异,以及肥胖表型高、低两组志愿者之间粪便微生物的α-多样性和β-多样性是否存在统计学差异,使用错误发现率(false discovery rate,FDR)对P值进行校正。使用线性回归分析计算相对丰度前20的菌属或菌种与肥胖表型之间的关联程度时,校正了协变量年龄和性别,且对菌群和肥胖表型数据分别进行归一化处理。以P<0.05为差异有统计学意义,以P<0.01为差异有显著统计学意义。使用R包(4.1.1版)vegan[24]、ade4[23]、ggplot2、pheatmap对数据进行分析和可视化。
结果志愿者的菌群结构特征 在我们的志愿者中,肠道微生物主要来自两个属。志愿者S01和S05的肠道菌群主要由普氏菌属(Prevotella)组成,其中普氏菌属在S01的肠道菌群中比例高达56.0%,在S05中比例为41.9%;在其他志愿者中拟杆菌属(Bacteroides)的相对丰度最高(图 2A)。在相对丰度前10的菌种中,S01和S05以普氏菌属中的人体普氏菌(Prevotella copri)相对丰度为最高,分别为56.0%和42.0%;S03(15.8%)、S06(17.0%)和S08(25.3%)的肠道菌群中都含有一定比例普通拟杆菌(Bacteroides vulgatus);S02和S04中单形拟杆菌(Bacteroides uniformis)占比较高,分别为19.4%和27.4%;S04(10.4%)和S06(20.0%)中多形拟杆菌(Bacteroides thetaiotaomicron)占比较高(图 2B)。菌群属水平的分型结果表明,优势菌群结构相似的样本分别聚成两个簇,以肠道内的细菌种类和数量划分,8个志愿者分成了以拟杆菌属和普氏菌属为优势菌属的2种肠型:拟杆菌型和普氏菌型(图 3)。Wilcoxon秩和检验显示不同性别的志愿者在肠道菌群的α-多样性(Shannon index:P=0.64;Simpson index:P=0.64)和基于Bray-Curtis距离计算的β-多样性第一主成分(the first principal component analysis,PCoA1)(Bray-Curtis PCoA1:P=0.86)上差异无统计学意义。
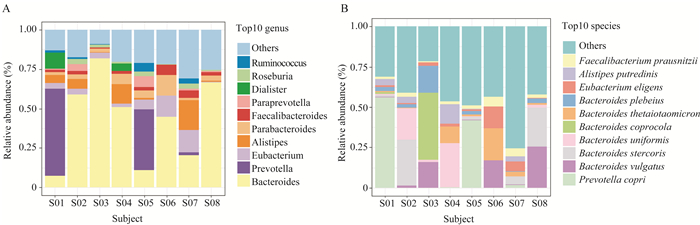
|
| 图 2 8名志愿者中相对丰度前10的菌属(A)和菌种(B)的堆积柱状分布图 Fig 2 Stacked columnar distribution of the top 10 genera (A) and species (B) in relative abundances from 8 subjects |
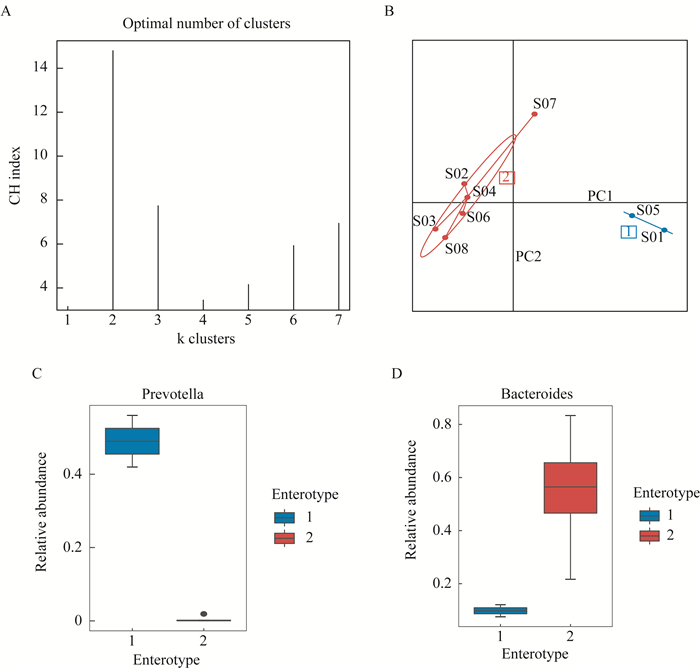
|
| A: The optimal number of clusters based on Calinski-Harabasz (CH) index; B: Between class analysis, which visualizes results from principal component analysis and clustering, of the genus compositions; C: Abundances of the main contributors of Prevotella enterotype; D: Abundances of the main contributors of Bacteroides enterotype. 图 3 不同肠型志愿者之间的系统发育差异 Fig 3 Phylogenetic difference among the subjects of different enterotypes |
肠道微生物与饮食结构的关联 食物相似性聚类结果显示,8个志愿者的饮食结构不同(图 4A)。基于饮食结构的主成分分析(principal components analysis,PCoA)能够将志愿者饮食结构差异可视化,其中S01和S05的饮食结构第一主坐标距离近,S03、S04、S06和S07的第一主坐标距离近,饮食结构分型与肠型聚类有吻合之处(图 4B)。我们用Euclidean距离计算3天24 h饮食回顾的膳食摄入量β-多样性,用Bray-Curtis距离计算微生物(种水平β-多样性),普氏分析的平均饮食和平均微生物群距离之间具有显著的一致性(P=0.01,图 5)。双侧t检验结果显示两种肠型志愿者之间的十字花科蔬菜(P=0.032)、豆类及豆制品(P=0.015)、精加工谷物(P=0.008)和全谷物(P=0.048)摄入量差异有统计学意义。普氏菌型志愿者十字花科蔬菜、精加工谷物和全谷物的食用量约是拟杆菌型志愿者的2倍,而豆类及豆制品的食用量则远低于拟杆菌型志愿者;拟杆菌型志愿者鱼和其他海鲜类产品、动物内脏、红肉的食用量高于普氏菌型志愿者(表 1)。

|
| A: Heatmap of dietary structure clustering analysis; B: The principal component analysis of dietary structure based on Euclidean distance from eight subjects. 图 4 志愿者3天24 h饮食回顾数据的饮食结构分析 Fig 4 Analysis of dietary structure based on 3-day, 24-hour diet recall data from the subjects |

|
| 图 5 志愿者饮食结构与肠道菌群普氏分析 Fig 5 Procrustes analysis between dietary structure and gut microbiome from the subjects |
| Diet composition(g) | Enterotype | P | |
| Prevotella(n=2) | Bacteroides(n=6) | ||
| Cruciferous vegetables | 108.8 | 50.0 | 0.032 |
| Dairy products | 125.0 | 123.8 | 0.983 |
| Fish and other seafood | 0 | 85.4 | 0.125 |
| Fruits | 305.0 | 190.0 | 0.292 |
| Legume | 0 | 83.3 | 0.015 |
| Organ meats | 0 | 47.5 | 0.142 |
| Other vegetables | 120.0 | 114.6 | 0.918 |
| Poultry | 68.8 | 71.7 | 0.894 |
| Red meat | 96.3 | 143.5 | 0.343 |
| Refined grains | 482.5 | 250.8 | 0.008 |
| Sweets and desserts | 5.0 | 33.2 | 0.142 |
| Whole grains | 35.0 | 13.3 | 0.048 |
肠道微生物与肥胖表型的关系 根据每个肥胖表型(BMI、Trunk、Android、Gynoid、AGR、TBF及WHR)分别对志愿者进行分组(表 2),低于中位值视为低肥胖表型组,高于中位值视为高肥胖表型组,分析两组之间α-多样性和β-多样性的区别。结果提示,体脂含量低的志愿者可能拥有更高的菌群物种丰富度(Shannon指数)与更低的物种均匀度(Simpson指数),但差异无统计学意义(图 6A、6B)。Trunk、Android、Gynoid和TBF这4个肥胖表型与β-多样性具有相关性(P<0.05,图 6C)。就Android、Gynoid、TBF和Trunk这4个肥胖表型而言,在体脂较高和体脂较低的两组人中,肠道微生物物种组成差异明显。肥胖表型与相对丰度前20的菌属之间的线性回归分析显示:BMI、WHR分别与副杆菌属(Parabacteroides)和萨特氏菌属(Sutterella)呈正相关(图 7A);拟杆菌属与Trunk、Android以及TBF呈显著正相关(图 7A);而普氏菌属与Trunk、Android、Gynoid及TBF呈显著负相关(图 7A);小杆菌属(Dialister)与Gynoid呈负相关(图 7A)。当使用相对丰度前20的菌种进行回归分析时时,BMI、WHR分别与多形拟杆菌、狄氏副拟杆菌(Parabacteroides distasonis)以及挑剔真杆菌(Eubacterium eligens)呈正相关(图 7B);普通拟杆菌与Trunk、Gynoid及TBF呈显著正相关(图 7B);而人体普氏菌与Trunk、Android、Gynoid及TBF这4种肥胖表型呈显著负相关(图 7B);Gynoid与Dialister succinatiphilus呈负相关(图 7B)。
| Adiposity phenotype | Median(IQR) | High group(n) | Low group(n) |
| BMI(kg/m2) | 20.2(19.7,23.5) | 3 | 5 |
| Trunk(%) | 28.6(16.8,33.0) | 5 | 3 |
| Android(%) | 34.0(19.4,39.8) | 5 | 3 |
| Gynoid(%) | 28.2(25.8,32.7) | 4 | 4 |
| AGR | 1.01(0.86,1.26) | 4 | 4 |
| TBF(%) | 24.8(16.6,29.3) | 5 | 3 |
| WHR | 0.81(0.80,0.85) | 3 | 5 |
| IQR:Inter quartile range;BMI:Body mass index;AGR:Android-to-Gynoid fat ratio;TBF:Total body fat;WHR:Waist-to-hip ratio. | |||
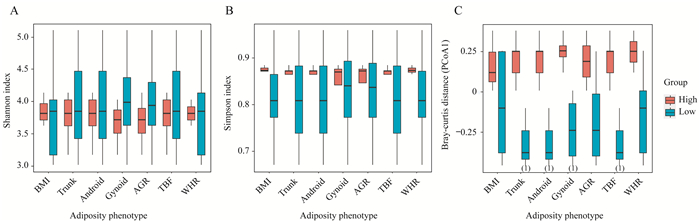
|
| A: Shannon index based on high and low adiposity phenotype groups; B: Simpson index based on high and low adiposity phenotype groups; C: The first principal component of Bray-Curtis distance. (1)P < 0.05. 图 6 肥胖表型高、低组志愿者之间粪便微生物的α-多样性和β-多样性 Fig 6 α- and β-diversity of the fecal microbiome in subjects with high and low adiposity phenotype |
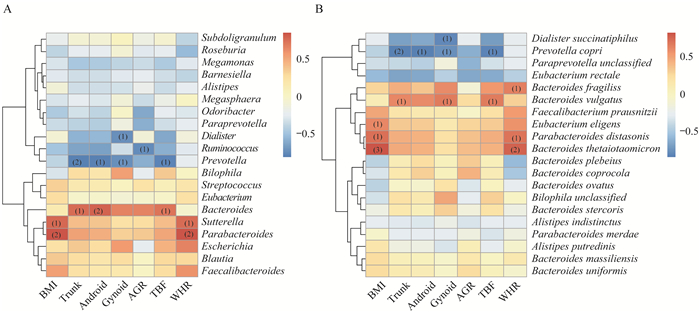
|
| (1)P < 0.05, (2)P < 0.01, (3)P < 0.001 图 7 肠道微生物相对丰度排名前20的属(A)和种(B)与肥胖表型及微生物多样性的关联 Fig 7 Relationship between the top 20 genera (A) and species (B) in the relative abundance of gut microbes with adiposity phenotype and microbial diversity |
肠型的研究最早于2011年提出,当时的研究(n=39)根据肠道内的细菌种类和数量划分出三种肠型:拟杆菌型、普氏菌型和瘤胃球菌型,分别反映了各肠道生态系统中的优势菌[25]。随后,Wu等[22]研究发现,粪便微生物群落聚集成的肠型,主要以拟杆菌和普氏菌的水平来区分,肠型与长期饮食密切相关,尤其是蛋白质、动物脂肪、碳水化合物。2017年,29位肠道菌群研究专家对肠型这一备受争议的概念做了充分探究[26],考虑到准确量化肠道群落结构的挑战,肠道稳定状态的确切定义难以确定。尽管如此,在许多研究(主要为西方)中,拟杆菌属和普氏菌属都是个体间最主要的差异类群,并能够解释菌群最主要的变异[26]。本研究根据肠道内的细菌种类和数量把志愿者分成两种肠型:拟杆菌型和普氏菌型,与既往报道相一致。本研究尚处于初步探索阶段,需要在大样本人群中进一步验证。既往研究表明,拟杆菌型人群擅长消化各类食物;普氏菌型人群对脂类代谢较弱。肠型和血型一样,与年龄、性别、国籍、体重指数等特征无关,但受到长期饮食习惯的影响[25]。
我们在做饮食结构与肠道菌群的普氏分析时发现,饮食结构与肠道微生物群落结构之间显著相关,而且普氏菌型志愿者摄入十字花科蔬菜和谷物的量远高于拟杆菌属志愿者。
该研究确定了肠道菌群改变、循环氨基酸和肥胖症之间的未知联系,为靶向肠道菌群进行肥胖症干预提供了思路。在益生元干预评估超重和肥胖儿童肠道微生物组成和粪便胆汁酸变化总量的随机对照研究中,给予富含低聚果糖的菊粉对照组在干预16周后,体重评分明显下降,肠道微生物群中双歧杆菌增加,普通拟杆菌则显著减少,躯干脂肪的变化与普通拟杆菌的变化呈显著正相关[27-28],本研究结果与文献报道一致。Vadder[29]研究发现,人体普氏菌通过与膳食纤维发酵过程中产生琥珀酸酯的机制改善了葡萄糖代谢和胰岛素敏感性,发现富含可发酵膳食纤维饮食与高琥珀酸酯的产生有关。就肥胖表型相关基因或通路而言,Guo等[30]研究发现,在中国成年人群中,脂肪酸相关的基因多态性与脂肪分布和代谢危险因素有关,并且相关性存在性别差异。肠道微生物主要通过菌群代谢产物和衍生物调控人类宿主表型,它们作为菌群的“信使”,影响宿主能量稳态,调控宿主代谢,从而影响肥胖表型的产生和变化。例如,在胆汁酸通路上,肠道微生物将初级胆汁酸转换成为次级胆汁酸,后者具有促进能量代谢、增加胰岛素敏感性、减轻炎症的作用;在短链脂肪酸通路上,肠道微生物作用于膳食纤维,生成短链脂肪酸,为肠道微生物提供能量来源,同时可刺激饱腹感激素的产生[31]。
本研究发现饮食和肠道微生物之间具有相关性,进一步发现不同肠型之间饮食组分的差异:普氏菌型志愿者比拟杆菌型志愿者食用更多的十字花科蔬菜和谷物。肠道微生物和肥胖表型之间的关系显示,普氏菌属与Trunk、Android、Gynoid和TBF呈负相关,拟杆菌属与Trunk、Android和TBF呈正相关。根据饮食与肠道菌群的相关性以及肠道菌群与肥胖表型的相关性,我们做出合理推测:肠道菌群可能介导饮食对于肥胖表型的作用。影响肠道微生物构成的因素很多,本研究主要探讨了饮食和性别的影响,虽然控制了年龄、地理位置、药物使用以及胃肠道疾病这些变量,但在研究设计中没有考虑到志愿者的运动情况。由于样本量相对较小,缺乏深入的生物和生化信息,无法解释饮食、微生物、宿主和其他潜在因素之间的复杂生理联系。本研究表明肠道微生物群的改变是肥胖表型改变的标志,可能介导饮食对于肥胖的作用。未来的研究应当就影响肠道菌群介导饮食作用于肥胖的混杂因素(如运动情况、年龄、地理位置等),针对不同特征的大样本人群(如老人、儿童或疾病患者)进行研究,进而结合个体差异性来认知肠道微生物群与健康人群之间的流行病学关联。
作者贡献声明 管慧慧,刘成林 论文构思、撰写和修订,数据采集和统计分析,制图。蒲彦霓 饮食结构数据分析,制图,论文修订。戴宇翔 论文指导和修订。宗耕 项目构建,论文修订。郑琰 论文构思、指导和修订。
利益冲突声明 所有作者均声明不存在利益冲突。
| [1] |
WANG YC, MCPHERSON K, MARSH T, et al. Health and economic burden of the projected obesity trends in the USA and the UK[J]. Lancet, 2011, 378(9793): 815-825.
[DOI]
|
| [2] |
LEE JE, NAM CM, LEE SG, et al. The health burden of cancer attributable to obesity in Korea: a population-based cohort study[J]. Cancer Res Treat, 2019, 51(3): 933-940.
[DOI]
|
| [3] |
GROVER SA, KAOUACHE M, REMPEL P, et al. Years of life lost and healthy life-years lost from diabetes and cardiovascular disease in overweight and obese people: a modelling study[J]. Lancet Diabetes Endocrinol, 2015, 3(2): 114-122.
[DOI]
|
| [4] |
LEUNG MY, POLLACK LM, COLDITZ GA, et al. Life years lost and lifetime health care expenditures associated with diabetes in the U.S., National Health Interview Survey, 1997-2000[J]. Diabetes Care, 2015, 38(3): 460-468.
[DOI]
|
| [5] |
ROMERO-CORRAL A, SOMERS VK, SIERRA-JOHNSON J, et al. Accuracy of body mass index in diagnosing obesity in the adult general population[J]. Int J Obes (Lond), 2008, 32(6): 959-966.
[DOI]
|
| [6] |
LE ROY CI, BEAUMONT M, JACKSON MA, et al. Heritable components of the human fecal microbiome are associated with visceral fat[J]. Gut Microbes, 2018, 9(1): 61-67.
[DOI]
|
| [7] |
LEE JJ, FREELAND-GRAVES JH, PEPPER MR, et al. Prediction of android and gynoid body adiposity via a three-dimensional stereovision body imaging system and dual-energy X-ray absorptiometry[J]. J Am Coll Nutr, 2015, 34(5): 367-377.
[DOI]
|
| [8] |
BOUCHI R, FUKUDA T, TAKEUCHI T, et al. Gender difference in the impact of gynoid and android fat masses on the progression of hepatic steatosis in Japanese patients with type 2 diabetes[J]. BMC Obes, 2017, 4: 27.
[DOI]
|
| [9] |
AUCOUTURIER J, MEYER M, THIVEL D, et al. Effect of android to gynoid fat ratio on insulin resistance in obese youth[J]. Arch Pediatr Adolesc Med, 2009, 163(9): 826-831.
[DOI]
|
| [10] |
SAMSELL L, REGIER M, WALTON C, et al. Importance of android/gynoid fat ratio in predicting metabolic and cardiovascular disease risk in normal weight as well as overweight and obese children[J]. J Obes, 2014, 2014: 846578.
[URI]
|
| [11] |
JIN B, LIN H, YUAN J, et al. Abdominal adiposity and total body fat as predictors of cardiometabolic health in children and adolescents with obesity[J]. Front Endocrinol (Lausanne), 2020, 11: 579.
[DOI]
|
| [12] |
MARTÍN HOLGUERA R, TURRIÓN NIEVES AI, RODRÍGUEZ TORRES R, et al. Relationship of muscle mass determined by DEXA with spirometric results in healthy individuals[J]. Arch Bronconeumol, 2017, 53(7): 375-380.
[DOI]
|
| [13] |
GONZÁLEZ-MUNIESA P, MáRTINEZ-GONZÁLEZ MA, HU FB, et al. Obesity[J]. Nat Rev Dis Primers, 2017, 3: 17034.
[DOI]
|
| [14] |
HRUBY A, HU FB. The epidemiology of obesity: a big picture[J]. Pharmacoeconomics, 2015, 33(7): 673-689.
[DOI]
|
| [15] |
ROUHANI MH, SALEHI-ABARGOUEI A, SURKAN PJ, et al. Is there a relationship between red or processed meat intake and obesity? A systematic review and meta-analysis of observational studies[J]. Obes Rev, 2014, 15(9): 740-748.
[DOI]
|
| [16] |
HU FB. Resolved: there is sufficient scientific evidence that decreasing sugar-sweetened beverage consumption will reduce the prevalence of obesity and obesity-related diseases[J]. Obes Rev, 2013, 14(8): 606-619.
[DOI]
|
| [17] |
TILG H, KASER A. Gut microbiome, obesity, and metabolic dysfunction[J]. J Clin Invest, 2011, 121(6): 2126-2132.
[DOI]
|
| [18] |
DONG TS, LUU K, LAGISHETTY V, et al. A high protein calorie restriction diet alters the gut microbiome in obesity[J]. Nutrients, 2020, 12(10): 3221.
[DOI]
|
| [19] |
SEGATA N, WALDRON L, BALLARINI A, et al. Metagenomic microbial community profiling using unique clade-specific marker genes[J]. Nature methods, 2012, 9(8): 811-814.
[DOI]
|
| [20] |
汪之顼, 孙忠清, 钟春梅. 回顾性膳食调查辅助参照食物图谱的研制[C]//中国营养学会妇幼分会. 中国营养学会妇幼营养第七次全国学术会议论文汇编, 2010: 484-488.
|
| [21] |
MUSHTAQ H, KHAWAJA SG, AKRAM MU, et al. A parallel architecture for the partitioning around medoids (PAM) algorithm for scalable multi-core processor implementation with applications in healthcare[J]. Sensors (Basel), 2018, 18(12): 4129.
[DOI]
|
| [22] |
WU GD, CHEN J, HOFFMANN C, et al. Linking long-term dietary patterns with gut microbial enterotypes[J]. Science, 2011, 334(6052): 105-108.
[DOI]
|
| [23] |
DRAY S, DUFOUR AB. The ade4 package: implementing the duality diagram for ecologists[J]. J Stat Softw, 2007, 22(4): 1-20.
[URI]
|
| [24] |
OKSANEN J. Multivariate analysis of ecological communities in R: vegan tutorial[CP]. 2013.
|
| [25] |
ARUMUGAM M, RAES J, PELLETIER E, et al. Enterotypes of the human gut microbiome[J]. Nature, 2011, 473(7346): 174-180.
[DOI]
|
| [26] |
COSTEA PI, HILDEBRAND F, ARUMUGAM M, et al. Enterotypes in the landscape of gut microbial community composition[J]. Nature Microbiology, 2018, 3(1): 8-16.
[DOI]
|
| [27] |
NICOLUCCI AC, HUME MP, MARTÍNEZ I, et al. Prebiotics reduce body fat and alter intestinal microbiota in children who are overweight or with obesity[J]. Gastroenterology, 2017, 153(3): 711-722.
[DOI]
|
| [28] |
DEWULF EM, CANI PD, CLAUS SP, et al. Insight into the prebiotic concept: lessons from an exploratory, double blind intervention study with inulin-type fructans in obese women[J]. Gut, 2013, 62(8): 1112-1121.
[DOI]
|
| [29] |
VADDER FD, KOVATCHEVA-DATCHANG P, ZITOUN C, et al. Microbiota-produced succinate improves glucose homeostasis via intestinal gluconeo-genesis[J]. Cell Metab, 2016, 24(1): 151-157.
[DOI]
|
| [30] |
GUO HL, ZHANG LC, ZHU CN, et al. A single nucleotide polymorphism in the FADS1 gene is associated with plasma fatty acid and lipid profiles and might explain gender difference in body fat distribution[J]. Lipids Health Dis, 2017, 16(1): 67.
[DOI]
|
| [31] |
BROWN JM, HAZEN SL. The gut microbial endocrine organ: bacterially derived signals driving cardiometabolic diseases[J]. Annu Rev Med, 2015, 66: 343-359.
[DOI]
|
 2021, Vol. 48
2021, Vol. 48


