2. 国际医疗中心 上海 200040
2. WorldWide Medical Center, Huashan Hospital, Fudan University, Shanghai 200040, China
骨髓增生异常综合征(myelodysplastic syndromes,MDS)是一组造血干/祖细胞克隆性疾病,表现为髓系发育不良,血细胞减少,并有进展为急性髓系白血病的风险[1]。对MDS发生的分子机制认识至今仍十分有限,阻碍了MDS治疗的进展[2]。近年来表观遗传学改变在MDS发生中的作用日益受到重视。长链非编码RNA(long non-coding RNA,LncRNA)是一类由RNA聚合酶Ⅱ合成、长度大于200个核苷酸、没有长阅读框架的RNA [3]。功能研究表明LncRNA在表观遗传学水平对细胞生命活动发挥着重要的调控作用[4]。虽然目前已有研究表明HOXB-AS3[5]、XIST[6]、MEG3[7]等LncRNA与MDS有关,但相对LncRNA众多的基因数量和在高等真核生物复杂调控网络中的重要作用,仍有大量可能参与MDS病理过程的LncRNA未被发现。我们在前期研究中发现ABAT基因在MDS患者中具有高甲基化低表达的特点,ABAT的异常表达与MDS的不良预后有关,且可影响MDS转白细胞系的增殖和凋亡[8-9],但对于调控ABAT的上游分子仍不明确。近年来的研究表明,通过对多种高通量分析方法得到的数据进行整合分析,相比只对单一芯片数据进行分析更加有助于揭示肿瘤发生过程中复杂的生物调控过程[10-11]。因此,本研究中我们将MDS患者与对照者骨髓标本的DEGs、DEMs、DELs进行整合分析,构建具有MDS特征的调控网络,从中筛选出调控ABAT的LncRNA,并对其在MDS中的功能进行初步研究。本研究有助于进一步了解LncRNA在MDS发生过程中的作用,以期为MDS的治疗提供新的策略和靶点。
资料和方法标本来源 LncRNA检测选择2015年1月1日至2016年12月30日复旦大学附属华山医院血液科收治的23例MDS患者,其中男性15例、女性8例,平均年龄65岁(29~82岁);作为对照的7例非造血肿瘤患者(如缺铁性贫血、免疫性血小板减少症、脾功能亢进症等)平均年龄38岁(23~78岁),两组性别分布差异无统计学意义。芯片检测病例组为4例MDS患者,其中2例分型为RAEB-1(refractory anemia with excess blasts 1),2例为RAEB-2,男性3例、女性1例,平均年龄67岁(24~80岁);另以年龄(±5岁)、性别匹配,选择同期行骨髓穿刺检查的4例非造血肿瘤患者作为对照组。所有患者采集骨髓标本时均为初次诊断、未接受化疗、排除MDS/骨髓增殖性疾病(myeloproliferative neoplasms,MPN)及MDS转白患者。采集骨髓标本2~3 mL后,均使用Ficoll淋巴细胞分离液(美国GE公司)密度梯度离心法获得骨髓单个核细胞(bone marrow mononuclear cells,BMMNCs)。MDS诊断及分型均按照2016年版WHO淋巴造血系统肿瘤分类标准[1]。本研究经复旦大学附属华山医院伦理委员会批准(批准号:2015-269),患者均签署了临床研究知情同意书。
细胞系 人急性白血病细胞系THP-1购自中国科学院(上海),人MDS转白急性白血病细胞系SKM-1购自日本细胞库(the Japanese Collection of Research Bioresources,LCRB)。作为对照的HEK-293T细胞系购自中国科学院(上海)。所有细胞系均在RPMI-1640培养基(Hyclone,美国GE公司)中培养,添加10%胎牛血清(Gibco-BRL,美国Thermo Fisher Scientific公司),置于5%CO2,37 ℃培养箱,每2天换液1次。
芯片分析 通过血液基因组DNA和RNA提取试剂盒(美国Qiagen公司)对4例MDS患者和4例对照患者的骨髓标本提取基因组DNA和RNA。采用Infinium Human Methylation 450K BeadChips甲基化芯片和Agilent human genomewide gene expression 60K BeadChips表达谱基因芯片(上海欧易生物科技有限公司)检测。60K BeadChip人类全基因组表达芯片(Agilent)中共包含27 958个Entrez Gene RNA和7 419个LncRNA。芯片扫读后会获得原始信号值、标准化信号值以及检出情况,对所有样本进行主成分分析(principal component analysis,PCA),考察样品的分布情况,确定重复样品的均一性,之后根据芯片确认单中提供的分析方法进行数据比对。分位数归一化和随后的数据处理通过Genespring软件(版本12.0,美国Agilent Technologies公司)完成。之后对原始数据进行筛选,表达量在两组间表达差异超过2倍,Student's t检验P < 0.01被认为存在差异表达。
为了构建MDS相关的调控网络,我们采用一种基于LncRNA-微RNA(microRNA,miRNA)靶向方法来预测LncRNA的潜在靶标。首先,对每种DELs,通过miRcode数据库[12]获得潜在的miRNA产物。之后在数据库miRbase v21、mirTarbase、miRanda 2010、miRbase v18、Tarbase5.0、targetscan v6.2中预测miRNA的潜在靶标,过滤掉了少于3个数据库注释的基因。将剩余的基因与芯片筛选出的差异表达基因组进一步比较。然后使用chipBase数据库分析LncRNA的转录因子,进一步分析转录因子与差异基因,利用此种方法构建了MDS相关的LncRNA-miRNA-DEGs-转录因子(transcription factors,TFs)调控网络。
基于LncRNA-TFs-DEGs调控网络筛选ABAT基因的调控LncRNA 采用Pearson相关系数(PCC)评估来构建ABAT与相关LncRNA的共表达调控网络。通过计算绝对PCC,取P值≤ 0.01,PCC倍数 > 0.99作为阈值,建立ABAT基因和DELs的共表达网络模型。我们通过计算PCC和P值来评估LncRNA-信使RNA(messenger RNA,mRNA)的相关性。用Cytoscape software 3.2.0(美国The Cytoscape Consortium公司)绘制ABAT-DELs-DEGs共表达网络。
qRT-PCR法检测LncRNA-SET2-1和ABAT的表达 用Trizol(Invitrogen,美国Thermo Fisher Scientific公司)抽提骨髓标本及培养细胞的总RNA,用Takara PrimeScript RT Master Mix试剂盒(日本Takara公司)将RNA反转录为cDNA。使用Applied BiosystemsTM 7500 Real-Time PCR system(美国Thermo Fisher Scientific公司)进行qRT-PCR扩增,反应体系为20 μL,冰上加入反转录产物2 μL,每条引物0.4 μL(10 μmol/L),SYBR® Premix Ex TaqTM(日本Takara公司)10 μL,ROX Reference Dye Ⅱ(日本Takara公司)0.4 μL,双蒸水6.8 μL。反应条件:95 ℃反应30 s预变性,95 ℃反应5 s,60 ℃反应34 s,共40个循环。引物序列如下,LncRNA-SET2-1,Forward:5'-TCGCCGGAGTC-ATATTATCG-3',Reverse:5'-CAGCAGCAGGA-AGAGCAGTT-3';ABAT,Forward:5'-CCGACT-ACAGCATCCTCTCC-3',Reverse:5'-GGTTCTC-TTTCACAAACTCTTCC-3';GAPDH,Forward:5'-GACCTGACCTGCCGT-CTA-3',Reverse:5'-AGGAGTGGGTGTCGCTG-T-3'。采用2-ΔΔCT方法计算LncRNA-SET2-1和ABAT表达水平,以GAPDH为内参,每组取3个复孔均值,根据各样本的平均CT值,以2-ΔΔCT表示目的基因表达水平,LncRNA-SET2-1或ABAT相对表达量=2-ΔΔCT(LncRNA-SET2-1或ABAT)-2-ΔΔCT(GAPDH)。
构建LncRNA-SET2-1过表达慢病毒载体和稳定转染的细胞系 按照分子克隆技术指南[13]构建含有LncRNA-SET2-1过表达片段的GV470慢病毒载体(上海吉凯基因医学科技股份有限公司)。取对数生长期的SKM-1细胞和THP-1细胞,按1×106/mL接种于6孔板,过夜后将LncRNA-SET2-1过表达的慢病毒载体和对照空载体分别转染SKM-1细胞和THP-1细胞。由于慢病毒载体带有绿色荧光蛋白序列,转染后72 h通过使用流式细胞仪检测绿色荧光蛋白强度来评估细胞转染效率,阳性细胞比例大于80%。之后,在SKM-1细胞和THP-1细胞的培养基中分别加入1 μg/mL、2 μg/mL的嘌呤霉素。培养2周后获得稳定转染带有嘌呤霉素抗性慢病毒载体的细胞系。继续培养4天后,应用qRT-PCR检测稳定转染细胞系中LncRNA-SET2-1的表达水平。
CCK-8法检测细胞增殖活力 使用Cell Counting Kit-8(CCK-8)试剂盒检测细胞增殖活力。将稳定过表达LncRNA-SET2-1的SKM-1细胞、THP-1细胞和转染空载体的对照细胞,按1×103/孔接种于96孔板,每组设3个复孔,培养5天,每3天更换一次细胞培养液。分别培养至24 h、48 h、72 h、96 h时,每孔加入5 μL CCK-8试剂,继续培养4 h后分光光度计上测量各孔450 nm处吸光度值(D),计算细胞增殖指数。
流式细胞术检测细胞凋亡 将稳定过表达LncRNA-SET2-1的SKM-1细胞、THP-1细胞和转染空载体的对照细胞按1×106/mL接种于6孔板,培养48 h后收集细胞,PBS洗2次后加入200 μL结合缓冲液重悬细胞,加入5 μL Annexin V-APC和5 μL 7-AAD(美国Thermo Fisher Scientific公司)混匀,避光室温孵育15 min。流式细胞仪(BD Accuri C6,美国BD Biosciences公司)检测细胞凋亡率,使用Flowjo 7.6软件进行数据统计分析。每次实验重复3次。
统计学处理 应用SPSS 25.0软件处理数据。符合正态分布的计量资料用x±s表示,两组间比较采用独立样本t检验,多组间比较采用单因素方差分析,两两比较采用LSD检验。P < 0.05为差异有统计学意义。
结果芯片整合分析 MDS患者相对对照者的BMMNCs,应用表达谱基因芯片共筛选出1 937个DEGs,其中853个基因表达上调,1 084个基因表达下调;筛选出214个DELs,其中102个LncRNA表达上调,112个LncRNA表达下调。应用甲基化基因芯片筛选出515个DEMs,其中高甲基化基因304个,低甲基化基因211个。对筛选出的DEGs、DELs、DEMs进行整合分析,构建MDS的LncRNA-miRNA-DEGs-TFs调控网络(图 1)。该调控网络包括:LncRNA-TFs-DEGs调控网络,TFs-miRNA-LncRNA调控网络,miRNA-DEGs- DEMs调控网络。GO(Gene ontology)分析差异表达基因参与的生物学过程,前10位GO富集的生物学过程包括:细胞内级联信号,凋亡调控,磷酸代谢过程,免疫反应,防御反应,细胞增殖调控,生物合成过程正向调控,磷酸化过程,通过RNA聚合酶II启动子的转录调控,生物黏附。

|
| The circles in the figure are composed of differentially expressed genes (DEGs), differentially expressed LncRNAs (DELs), differentially methylated genes (DEMs), LncRNA related microRNA (miRNA), predicted target genes (TGs), and predicted transcription factors (TFs) respectively.The lines among the six circles represent the interaction between different factors. 图 1 MDS整合调控网络 Fig 1 The integrated regulatory network of MDS |
基于LncRNA-TFs-DEGs调控网络筛选ABAT基因的调控LncRNA 在前期的研究中,我们筛选出6个高甲基化低表达的基因(ABAT,DAPPI,FADD,LRRFIPI,PLBDI,SMPD3)并建立了MDS的CpG岛甲基化表型,可能成为MDS诊断的潜在分子生物学标志[8]。本研究对以上6个基因进行了深入的信号通路整合分析,仅4个基因(ABAT,DAPP1,FADD和SMPD3)有功能信号通路。我们在前期研究中发现,ABAT基因异常低表达与MDS的不良预后有关,下调其表达水平可促进髓系肿瘤细胞系的增殖能力、抑制细胞凋亡,提示ABAT可能参与MDS的发生过程[9]。因此,我们在构建的MDS调控网络中,对ABAT参与的LncRNA-mRNA调控网络进一步分析。通过建立ABAT-DELs-DEGs共表达网络,我们发现了6个与ABAT共表达且相关性最强的DELs。根据PCC对这6个LncRNA进行排序发现,LncRNA-SET2-1与ABAT变化趋势的PCC最高(表 1)。利用UCSC数据库(http://genome.ucsc.edu)检索发现,LncRNA-SET2-1位于人类基因组6号染色体,为反义RNA,包括3个外显子,长度约385bp。对其进行PhyloCSF评分,确定其为非编码RNA。经DELs-DEGs共表达网络分析发现,LncRNA-SET2-1与ABAT、NCF2、SPTLC2、QPRT基因共表达。进一步通过LncRNA-miRNA-DEGs-TFs调控网络分析和数据库信息的匹配,预测到LncRNA-SET2-1可能为miRNA96的前体,而ABAT可能为miRNA96的靶分子。根据以上生物信息学分析,LncRNA-SET2-1可能对ABAT具有调控作用,且参与了MDS发展过程。因此,我们进一步研究LncRNA-SET2-1在MDS患者及细胞系中的表达情况,及对MDS转白细胞功能的影响。
| No. | LncRNA | Chromosomal localization | Expression change | PCC | P |
| 1 | LncRNA-SET2-1 | chr6:167382710-167411729 | Down | 0.988 | 0.011 |
| 2 | Loc100131564 | chr1:93796837-93806487 | Down | 0.979 | 0.021 |
| 3 | Jh806582.2 | chr17:49425-59050 | Down | 0.959 | 0.041 |
| 4 | None | chr4:13067152-13347902 | Up | -0.968 | 0.018 |
| 5 | Jh591181.2 | chr10:46972944-46982894 | Up | -0.963 | 0.036 |
| 6 | None | chr5:61931044-61948469 | Up | -0.954 | 0.046 |
| PCC:Pearson correlation coefficient. | |||||
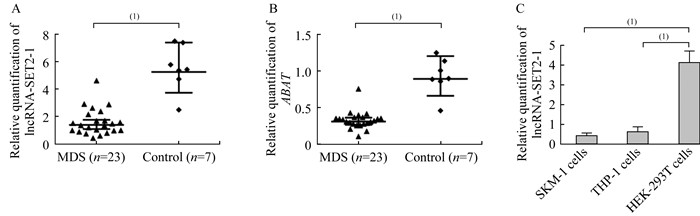
LncRNA-SET2-1在MDS患者和MDS转白细胞系中的表达情况 MDS患者骨髓细胞中LncRNA-SET2-1的相对表达量明显低于对照组(1.63±0.96 vs. 5.55±1.69,P < 0.000 1),同时检测ABAT基因在MDS患者骨髓细胞中的相对表达量也显著低于对照组(0.33±0.12 vs. 0.93±0.25,P < 0.000 1)。在MDS转白细胞系SKM-1和急性白血病细胞系THP-1中,LncRNA-SET2-1的表达量显著低于对照细胞HEK-293T(0.42±0.12 vs.4.12±0.60,0.62±0.23 vs.4.12±0.60,P < 0.000 1)(图 2)。
|
| A:Relative expression level of LncRNA-SET2-1 was downregulated in MDS patients detected by qRT-PCR; B:The relative expression level of ABAT gene was also downregulated in MDS patients; C:The expression of LncRNA-SET2-1 was downregulated in MDS cell lines SKM-1 and THP-1, HEK-293T cells were used as a control.(1)P < 0.000 1. 图 2 LncRNA-SET2-1和ABAT在MDS患者和MDS转白细胞系中的表达水平 Fig 2 Expression levels of LncRNA-SET2-1 and ABAT in MDS patients and cell lines |
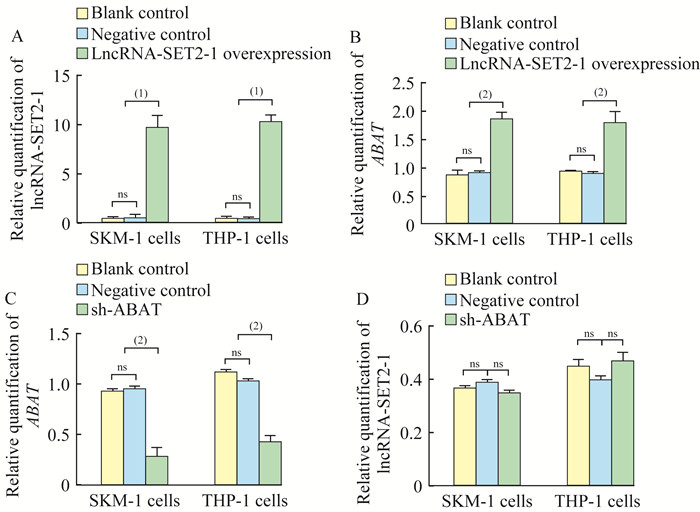
MDS转白细胞系中LncRNA-SET2-1过表达对ABAT基因表达的影响 稳定转染LncRNA-SET2-1过表达慢病毒载体的SKM-1细胞系和THP-1细胞系中,LncRNA-SET2-1的相对表达量高于对照空载体组(9.65±1.24 vs. 0.44±0.34,10.10±0.89 vs. 0.38±0.21,P < 0.000 1)。稳定过表达LncRNA-SET2-1的SKM-1细胞和THP-1细胞中ABAT基因的相对表达量较对照组上调2倍(1.86±0.13 vs.0.91±0.02,1.79±0.21 vs.0.89±0.03,P < 0.01)。但我们对前期研究中构建的ABAT敲减SKM-1和THP-1细胞系[9]中LncRNA-SET2-1的相对表达水平进行检测,与对照组相比未发生显著改变(0.35±0.01 vs. 0.39±0.01,0.47±0.03 vs. 0.40±0.01,P > 0.05)(图 3)。
|
| A:LncRNA-SET2-1 was significantly over-expressed in MDS cell lines which stably infected with lentivirus carrying LncRNA-SET2-1;B:The expression level of ABAT was upregulated in LncRNA-SET2-1 stable overexpression cell lines; C:ABAT was significantly under-expressed in MDS cell lines which ABAT was knocked down; D:The expression level of LncRNA-SET2-1 was not significantly changed in ABAT knockdown cell lines.(1)P < 0.000 1;(2)P < 0.01;ns:No statistical difference.Blank control:Control cells were not infected with a lentiviral vector; Negative control:Control cells were infected with empty lentiviral vectors; LncRNA-SET2-1 overexpression:LncRNA-SET2-1 overexpression cell lines; sh-ABAT:ABAT knockdown cell lines. 图 3 LncRNA-SET2-1过表达对ABAT基因表达的影响 Fig 3 Effect of LncRNA-SET2-1 overexpression on ABAT gene |
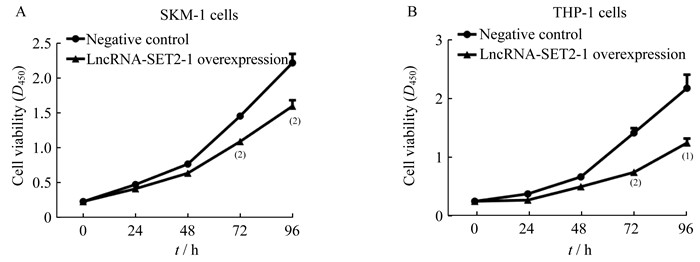
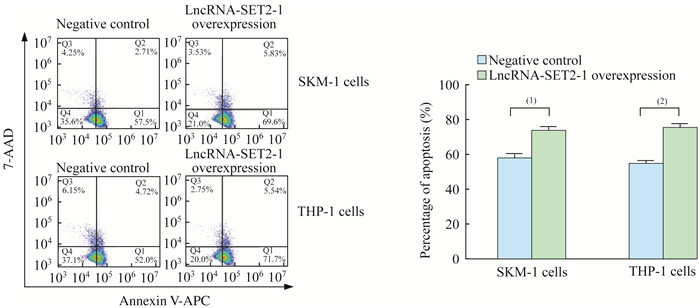
MDS转白细胞系中LncRNA-SET2-1过表达对细胞增殖和凋亡的影响 稳定过表达LncRNA-SET2-1的SKM-1细胞和THP-1细胞培养24 h后,与对照组相比,细胞增殖活力在72 h(D值分别为1.10±0.03 vs.1.47±0.02,0.75±0.02 vs.1.42±0.08,P < 0.001)、96 h(D值分别为1.61±0.09 vs. 2.23±0.13,P < 0.001;1.25±0.08 vs.2.18±0.23,P < 0.05)均显著下降(图 4)。流式细胞术检测过表达LncRNA-SET2-1的SKM-1细胞凋亡细胞比例显著高于对照组(73.39%±2.78% vs.57.77%±2.36%,P=0.02),同样,过表达LncRNA-SET2-1的THP-1细胞凋亡细胞比例也显著高于对照组(74.59%±3.07%vs.54.48%±2.19%,P=0.001)(图 5)。说明LncRNA-SET2-1过表达对MDS转白细胞的增殖活力具有抑制作用,并促进MDS转白细胞凋亡。
|
| A:The cell viability of SKM-1 cells with LncRNA-SET2-1 overexpression was reduced; B:The cell viability of THP-1 cells with LncRNA-SET2-1 overexpression was also reduced; (1)P < 0.05;(2)P < 0.001. Negative control:Control cells were infected with empty lentiviral vectors; LncRNA-SET2-1 overexpression:LncRNA-SET2-1 overexpression cell lines. 图 4 LncRNA-SET2-1过表达抑制MDS转白细胞增殖活力 Fig 4 LncRNA-SET2-1 overexpression reduced the viability of MDS transformed leukemia cell |
|
| The fraction of apoptotic cells in the SKM-1 and THP-1 cells with LncRNA-SET2-1 overexpression was increased.(1)P < 0.05;(2)P < 0.01.Negative control:Control cells were infected with empty lentiviral vectors; LncRNA-SET2-1 overexpression:LncRNA-SET2-1 overexpression cell lines. 图 5 LncRNA-SET2-1过表达促进MDS转白细胞凋亡 Fig 5 LncRNA-SET2-1 overexpression induced MDS transformed leukemia cell apoptosis |
LncRNA作为人类细胞中大量存在的非编码RNA,在信号转导、细胞增殖分化、细胞周期、个体发育等重要生命活动中发挥关键的调控作用,涉及到表观遗传水平、转录水平以及转录后水平等多个层面的复杂调控[14]。目前,在多种类型肿瘤中都已发现存在LncRNA的失调,说明其与肿瘤的发展密切相关[15]。LncRNA在调控造血发育中也起到重要作用,其异常调控参与了造血肿瘤的发展,如AML,MPN,ALL等[16-17]。虽然目前已有LncRNA与MDS发展有关的报道,但相比AML,对参与MDS病理过程的LncRNA认识仍十分有限。我们在前期研究中发现,ABAT基因在MDS中具有诊断和预后价值,并可影响MDS转白细胞的增殖活力和凋亡。本研究中,我们基于基因芯片整合分析构建ABAT-DELs-DEGs共表达网络,筛选出ABAT基因可能的调控LncRNA,并对该LncRNA在MDS患者和MDS转白细胞中的表达情况及功能进行了初步研究。
高通量基因芯片是帮助发现MDS发生过程中新的调控分子的重要工具,但是,仅依靠单一的LncRNA芯片或者mRNA芯片,难以从海量的数据中筛选出有价值的信息。利用多种不同芯片进行生物信息学整合分析、构建分子调控网络有助于揭示复杂的生物调控过程、筛选发现生物调控网络中的关键分子[18]。本研究中,我们对MDS患者和对照者骨髓标本中的DEGs、DELs和DEMs进行整合分析,通过对DELs靶分子与结合转录因子的预测,与表达芯片筛出的DEGs进行对比分析,构建了MDS相关的LncRNA-miRNA-DEGs-TFs调控网络。在该网络的基础上,我们寻找到可能作为LncRNA-SET2-1剪切产物的miRNA96,推测miRNA96可能作为ABAT的转录因子对ABAT基因进行调控,提示可能存在LncRNA-SET2-1-miRNA96-ABAT调控轴。
我们首次在MDS患者骨髓标本和MDS转白细胞系中检测发现LncRNA-SET2-1表达显著下调,与同时检测的ABAT基因表达变化趋势一致。过表达LncRNA-SET2-1后,ABAT基因表达上调,而下调ABAT表达,LncRNA-SET2-1表达不受影响,提示LncRNA-SET2-1可能为ABAT上游调控分子,且对ABAT具有正向调控作用。功能研究表明,LncRNA-SET2-1过表达会抑制MDS转白细胞增殖、促进细胞凋亡,说明在MDS中,LncRNA-SET2-1异常下调可能对造血细胞肿瘤化具有一定作用。由于在MDS患者和MDS转白细胞系中,LncRNA-SET2-1与ABAT基因表达变化一致,对细胞表型影响一致,且LncRNA-SET2-1可能为ABAT上游调控分子,结合芯片整合分析可能存在LncRNA-SET2-1-miRNA96-ABAT调控轴的结果,我们推测LncRNA-SET2-1和ABAT可能存在竞争性内源RNA(competing endogenous RNA,ceRNA)的关系。ceRNA是LncRNA发挥生物学功能的9种主要方式之一,代表了一种全新的基因表达调控模式,相比miRNA调控网络,涉及更多的RNA分子,更为精细和复杂,是目前肿瘤研究领域的一个热点[19-21]。对LncRNA-SET2-1-miRNA 96-ABAT调控轴的研究可为我们从转录组水平深入探索MDS的发病机制提供一个新的视角。
本研究首次基于MDS患者骨髓标本不同基因芯片的整合分析,发现可能存在LncRNA-SET2-1-miRNA96-ABAT调控轴,并初步验证了LncRNA-SET2-1对ABAT基因具有正向调控作用,且LncRNA-SET2-1表达失调可能参与了MDS的病理过程。由于本文仅对LncRNA-SET2-1在MDS中的功能进行了初步探索,因此具有一定局限性,关于LncRNA-SET2-1与ABAT之间具体调控方式和是否存在ceRNA的实验正在进行中。本研究为从转录组水平深入探索MDS发生的分子机制提供了新的生物信息学分析方法,并提出了可能参与MDS发生的新的LncRNA及其作用方式,对LncRNA-SET2-1-ABAT调控轴的深入研究将为认识MDS的病理机制提供新的方向。
作者贡献声明 王倩 论文构思,数据采集,数据统计和分析,论文撰写和修订。王小钦 数据审核,论文修订。陈燕珍 临床样本分析。赵光杰 细胞转染实验。吴宛玲 细胞活性和凋亡实验。李念夷 生物信息学分析,数据统计和分析,论文修订。
利益冲突声明 所有作者均声明不存在利益冲突。
| [1] |
ARBER DA, ORAZI A, HASSERJIAN R, et al. The 2016 revision to the World Health Organization classification of myeloid neoplasms and acute leukemia[J]. Blood, 2016, 127(20): 2391-2405.
[DOI]
|
| [2] |
LIU L, WANG HY, WEN JG, et al. Mutated genes and driver pathways involved in myelodysplastic syndromes-a transcriptome sequencing based approach[J]. Mol Biosyst, 2015, 11(8): 2158-2166.
[DOI]
|
| [3] |
AKHADE VS, PAL D, KANDURI C. Long noncoding RNA:genome organization and mechanism of action[J]. Adv Exp Med Biol, 2017, 1008: 47-74.
|
| [4] |
SCHMITZ SU, GROTE P, HERRMANN BG. Mechanisms of long noncoding RNA function in development and disease[J]. Cell Mol Life Sci, 2016, 73(13): 2491-2509.
[DOI]
|
| [5] |
HUANG HH, CHEN FY, CHOU WC, et al. Long non-coding RNA HOXB-AS3 promotes myeloid cell proliferation and its higher expression is an adverse prognostic marker in patients with acute myeloid leukemia and myelodysplastic syndrome[J]. BMC Cancer, 2019, 19(1): 617.
[DOI]
|
| [6] |
YILDIRIM E, KIRBY JE, BROWN DE, et al. Xist RNA is a potent suppressor of hematologic cancer in mice[J]. Cell, 2013, 152(4): 727-742.
[DOI]
|
| [7] |
BENETATOS L, HATZIMICHAEL E, DASOULA A, et al. CpG methylation analysis of the MEG3 and SNRPN imprinted genes in acute myeloid leukemia and myelodysplastic syndromes[J]. Leukemia Res, 2010, 34(2): 148-153.
[DOI]
|
| [8] |
赵晓丽, 王小钦, 李爽, 等. 骨髓增生异常综合征CpG岛甲基化模式的建立及其诊断价值研究[J]. 中华血液学杂志, 2014, 10(35): 944-948. |
| [9] |
ZHAO GJ, LI NY, LI S, et al. High methylation of the 4-aminobutyrate aminotransferase gene predicts a poor prognosis in patients with myelodysplastic syndrome[J]. Int J Oncol, 2019, 54(2): 491-504.
|
| [10] |
LI LF, PENG ML, XUE WH, et al. Integrated analysis of dysregulated long non-coding RNAs/microRNAs/mRNAs in metastasis of lung adenocarcinoma[J]. J Transl Med, 2018, 16(1): 372.
[DOI]
|
| [11] |
SHANG JL, DING Q, YUAN SS, et al. Network analyses of integrated differentially expressed genes in papillary thyroid carcinoma to identify characteristic genes[J]. Genes (Basel), 2019, 10(1): pii:E45.
[DOI]
|
| [12] |
JEGGARI A, MARKS DS, LARSSON E. miRcode:a map of putative microRNA target sites in the long non-coding transcriptome[J]. Bioinformatics, 2012, 28(15): 2062-2063.
[DOI]
|
| [13] |
GEILING B, VANDAL G, POSNER AR, et al. A modular lentiviral and retroviral construction system to rapidly generate vectors for gene expression and gene knockdown in vitro and in vivo[J]. PLoS One, 2013, 8(10): e76279.
[DOI]
|
| [14] |
KOPP F, MENDELL JT. Functional classification and experimental dissection of long noncoding RNAs[J]. Cell, 2018, 172(3): 393-407.
[DOI]
|
| [15] |
PENG WX, KOIRALA P, MO YY. LncRNA-mediated regulation of cell signaling in cancer[J]. Oncogene, 2017, 36(41): 5661-5667.
[DOI]
|
| [16] |
ALVAREZ-DOMINGUEZ JR, LODISH HF. Emerging mechanisms of long noncoding RNA function during normal and malignant hematopoiesis[J]. Blood, 2017, 130(18): 1965-1975.
[DOI]
|
| [17] |
MERA S, LINDBERG J, NILSSON C, et al. Expression Levels of long non-coding RNAs are prognostic for AML outcome[J]. J Hematol Oncol, 2018, 11(1): 52.
[DOI]
|
| [18] |
TAWANA K, DRAZER MW, CHURPEK JE. Universal genetic testing for inherited susceptibility in children and adults with myelodysplastic syndrome and acute myeloid leukemia:are we there yet?[J]. Leukemia, 2018, 32(7): 1482-1492.
[DOI]
|
| [19] |
JARROUX J, MORILLON A, PINSKAYA M. History, discovery, and classification of lncRNAs[J]. Adv Exp Med Biol, 2017, 1008: 1-46.
|
| [20] |
TAY Y, RINN J, PANDOLFI PP. The Multilayered complexity of ceRNA crosstalk and competition[J]. Nature, 2014, 505(7483): 344-352.
[DOI]
|
| [21] |
CHAN JJ, TAY Y. Noncoding RNA:RNA regulatory networks in cancer[J]. Int J Mol Sci, 2018, 19(5): 1310.
[DOI]
|
 2021, Vol. 48
2021, Vol. 48


